Genetica 4
La replicazione del DNA
Dalla struttura tridimensionale del DNA scoperta nel 1953, si
possono ipotizzare 3 tipi di replicazione:
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Possibili Meccanismi di replicazione
del DNA
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
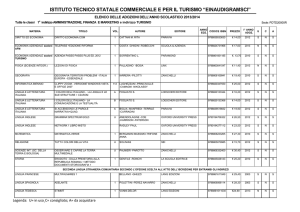
Esperimento di
Meselson e Stahl
Meselson e Stahl impiegarono la
centrifugazione in gradiente di densità
per distinguere DNA contenente 14N
(leggero) e DNA contenente 15N
(pesante). Batteri cresciuti in presenza
di 15N come unica fonte di azoto
incorporano 15N nelle basi puriniche e
pirimidiniche del loro DNA (basi azotate)
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Replicazione nei batteri che hanno
DNA circolare (E. coli per es.)
L’RNA E LA TRASCRIZIONE
La Trascrizione
La trascrizione comporta la sintesi di una catena di RNA che ha la stessa sequenza di un
filamento di una doppia elica di DNA.
•
Il filamento di DNA identico in sequenza all’RNA è chiamato filamento codificante
•
il filamento codificante è ovviamente complementare all’altro filamento, che funge da stampo per la
sintesi dell’RNA e che è chiamato per questa ragione filamento stampo o filamento non
codificante.
•
La sintesi della catena di RNA è catalizzata dall’enzima RNA polimerasi.
•
La trascrizione comincia quando la RNA polimerasi si lega ad una particolare regione
di DNA, chiamata promotore, che si trova all’inizio del gene.
•
fa parte della sequenza del promotore la prima coppia di basi trascritta in RNA, chiamata sito di
inizio o punto di inizio della trascrizione.
•
Partendo dal sito di inizio, la polimerasi si muove lungo lo stampo sintetizzando l’RNA,
fino a che non raggiunge una sequenza chiamata terminatore
•
Quest’azione definisce l’unità di trascrizione, che si estende dal promotore al
terminatore; l’unità di trascrizione è quindi una sequenza di DNA che viene espressa
mediante la sintesi di una singola molecola di RNA
•
una unità di trascrizione può codificare per più di una proteina.
Trascrizione
Sintesi di RNA a partire da un filamento di DNA stampo. Rappresenta il primo
passaggio nel trasferimento dell’informazione dal genotipo al fenotipo
Molecole di DNA in cui è in corso la trascrizione
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
La costituzione a singolo filamento permette alle
molecole di RNA di assumere varie conformazioni
secondarie. Dato che la struttura determina la
funzione, l’RNA può svolgere un’ampia gamma di
mansioni
Ribozimi: molecole di RNA ad attività catalitica
possibile grazie al fatto che essendo a singolo
filamento, la sua struttura secondaria puo’ variare
(nel DNA no perché a doppio filamento).
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Classi di RNA
RNA ribosomale assieme alle subunità proteiche costituisce il ribosoma
Alcuni snRNA partecipano al processo di maturazione dell’RNA
Gli snoRNA processano l’rRNA
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Classi di RNA
Classe di RNA
Tipo cellulare
Localizzazione della
funzione
Funzione
RNA ribosomale (rRNA)
Batterico ed
eucariotico
Citoplasma
Componenti funzionali e
strutturali del ribosoma
RNA messaggero (mRNA)
Batterico ed
eucariotico
Nucleo e citoplasma
Porta il codice genetico per
le proteine
RNA transfer (tRNA)
Batterico ed
eucariotico
Citoplasma
Aiuta a incorporare gli AA
nella catena polipeptidica
RNA nucleare piccolo (snRNA)
Eucariotico
Nucleo
processamento pre-mRNA
RNA nucleolare piccolo (snoRNA) Eucariotico
Nucleo
processamento e
assemblaggio del'rRNA
RNA piccolo citoplasmatico
(scRNA)
Citoplasma
Eucariotico
Il loro ruolo funzionale non è stato
ancora del tutto chiarito. Uno di
questi scRNA (RNA 7S) è coinvolto
nel processo di importazione cotraduzionale nel reticolo
endoplasmatico delle proteine
destinate alla secrezione.
Le molecole di RNA sintetizzate sono complementari e
antiparallele al filamento stampo
Lo stampo per la sintesi dell’RNA (e anche del DNA) è un singolo filamento della doppia elica di
DNA.
A differenza della replicazione, la trascrizione interessa solamente uno dei due filamenti, detto
filamento stampo. L’altro filamento è detto filamento codificante (non stampo)
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Un gene viene trascritto da un filamento, ma geni
diversi possono essere trascritti da filamenti diversi
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
L’ unità di trascrizione
Segmento di DNA che codifica per una molecola di RNA e che contiene le sequenze
necessarie alla sua trascrizione. Include 3 regioni: promotore, regione codificante e
terminatore
Il promotore è la regione del DNA che segnala:
1. quale dei due filamenti di DNA deve essere trascritto
2. qual è il sito di inizio della trascrizione
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
La trascrizione nei batteri
1. Promotori
2. RNA polimerasi batteriche
3. Terminatori
Promotori batterici
Hanno sequenze consenso a monte del sito di inizio, a -35 e a -10
La sequenza consenso a -10 è detta anche TATA-box. Essa è situata a circa -10 nei procarioti, e
a circa -30 negli eucarioti. È riconosciuta come la regione che facilita l'attacco della RNA polimerasi
e sulla quale la RNA polimerasi inizia ad aprire l'elica di DNA.
Le basi T ed A sono appaiate in modo da formare solo due legami idrogeno (regola
dell'appaiamento delle basi) in questo modo l'enzima RNA polimerasi è in grado di aprire la
bolla di replicazione senza grande dispendio energetico.
Alcuni promotori batterici contengono un terzo motivo di consenso, chiamato elemento a monte,
che contiene un certo numero di coppie A-T e si trova fra -40 e -60
Sequenze consenso dei promotori di
E. coli
Una sequenza consenso è costituita
da quei nucleotidi che si incontrano
più
frequentemente
in
una
determinata posizione
Il fattore Sigma si lega a -35 e a -10
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
RNA polimerasi batterica
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
RNA pol batterica costituita da 4 subunità che formano il CORE enzimatico: due ALFA, una BETA e una BETA
PRIMO (B’).
Il CORE catalizza l’allungamento della molecola di RNA mediante l’addizione di nucleotidi.
Il fattore sigma controlla il legame del Core al promotore. Quando il fattore sigma si attacca al core, formando un
oloenzima, la RNA pol si lega stabilmente al promotore e inizia la trascrizione . Dopo la sintesi dei primi nucleotidi
il fattore si stacca dato che non è più necessario.
Ci sono molteplici fattori sigma: 28,32,54 e 70 sulla base del peso molecolare. Ogni sigma promuove il legame
della RNA pol ad una particolare serie di promotori
Un enzima privo del cofattore che ne rende possibile l'attività enzimatica è detto apoenzima. Il legame tra
cofattore ed apoenzima permette la formazione del cosiddetto oloenzima
Nei batteri
Sintesi RNA: circa 40
nucleotidi al secondo.
Molto piu’ lenta della
sintesi del DNA:
1500 nucleotidi al
secondo.
La trascrizione
avviene all’interno di
un breve tratto di 18
nucleotidi di DNA
svolto: la bolla di
trascrizione. In ogni
momento 8 nucleotidi
di RNA neosintetizzato
sono appaiati al DNA
stampo
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Terminazione
Rho dipendente
(attività elicasica che svolge
l’ibrido RNA-DNA nella bolla
di trascrizione)
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Terminatori
rho-indipendenti
Il terminatore contiene sequenze
ripetute invertite cosi’ che l’RNA
forma una forcina. Questa forcina
rallenta la RNA polimerasi. Seguono
6 A subito dopo la forcina, per
favorire il distacco dell’RNA dal DNA
stampo
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
La traduzione nei procarioti
Sequenza di Shine-Dalgarno: sequenza del filamento di mRNA presso cui il
complesso ribosomale dei batteri è in grado di ancorarsi. Tale sequenza permette al
ribosoma di posizionarsi correttamente e di iniziare la traduzione con il corretto
frame di lettura.
Nel 1975 i ricercatori australiani John Shine e Lynn Dalgarno hanno individuato la
composizione di tale sequenza (AGGAGG) ed il suo posizionamento
immediatamente a monte del codone di inizio (AUG) della traduzione
La trascrizione negli eucarioti
1. Promotori
2. RNA polimerasi eucariotiche
3. Processamento (maturazione) dell’RNA
4. Terminatori
RNA polimerasi negli eucarioti
RNA polimerasi I
rRNA (rRNA28S, 18S, 5.8S)
RNA polimerasi II
pre-mRNA, snoRNA e alcuni snRNA
RNA polimerasi III
tRNA, piccoli rRNA, snRNA
Trascrizione ad opera dei fattori di trascrizione + RNA pol
Promotori, enhancer, soppressori
Le RNA polimerasi negli eucarioti
La RNA polimerasi I sintetizza gli rRNA28S, 18S, 5.8S
Per terminare la trascrizione la RNA pol I richiede un fattore di terminazione
che si lega al DNA a valle del sito di terminazione bloccando l’avanzamento
della RNA pol
La RNA polimerasi II sintetizza gli mRNA, gli snoRNAe alcuni snRNA
La RNA pol II continua a trascrivere anche a valle del 3’ del gene. L’estremità
3’ del trascritto viene determinata da un taglio effettuato da un complesso
enzimatico specifico
La RNA polimerasi III sintetizza l’rRNA5S, i tRNAe alcuni snRNA
La RNA pol III termina la trascrizione dopo avere trascritto una fila di U che
destabilizzano l’ibrido DNA-RNA
I fattori di trascrizione sono una classe di proteine accessorie che
unitamente alle RNA polimerasi costituiscono l’apparato basale della
trascrizione
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
L’inizio della trascrizione negli
eucarioti
Sono coinvolte due grandi classi di sequenze di DNA:
I promotori
Sono sempre adiacenti al gene che regolano, e mostrano una localizzazione
fissa rispetto al punto di inizio della trascrizione
Gli enhancer
Non sono necessariamente adiacenti al gene che regolano, e la loro
localizzazione rispetto al punto di inizio della trascrizione può variare
I promotori eucariotici
Y: T or C (pYrimidine)
N: G,A,T,C
Nei promotori eucariotici sono presenti diverse sequenze consenso
Non tutte le sequenze mostrate si rilevano in qualsiasi promotore
TFIIB è un fattore di trascrizione. Al posto del TATA box, alcuni promotori possiedono un elemento
iniziatore (Inr). Questi, generalmente possiedono anche un elemento 30 basi a valle del promotore (DPE:
downstream promoter element).
Tutte queste sequenze sono riconosciute da fattori di trascrizione che si legano ad esse e che fungono da
supporto per l’assemblaggio dell’apparato trascrizionale
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
http://www.uic.edu/classes/bios/bios100/lectf03am/eukprot.gif
TFIID è il fattore generale che riconosce il core promoter. Sui promotori riconosciuti dalla RNApol II, la
trascrizione inizia con il legame del fattore trascrizionale TFIID al TATA box, cui segue il legame dell’oloenzima
preassemblato che contiene fattori di trascrizione generali e da un complesso di proteine noto come
mediatore
Il promotore regolativo è localizzato immediatamente a monte del core promoter e contiene una vasta
gamma di sequenze consenso differenti, attivatori o repressori
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Some transcription factors ("Enhancer”) bind to regions of DNA that may be even
thousands of base pairs away from the gene they control. Binding increases the rate of
transcription of the gene.
Enhancers can be located upstream, downstream, or even within the gene they
control.
How does the binding of a protein to an enhancer regulate the transcription of a
gene thousands of base pairs away? One possibility is that enhancer- in addition to
their DNA-binding site, have sites that bind to transcription factors ("TF") assembled at
the promoter of the gene.
This would draw the DNA into a loop (as shown in the figure).
• Enhancers can work even if their normal 5' to 3' orientation is flipped
• Enhancers can work even if they are moved to a new location
• Regulatory sequences with similar characteristics, but the opposite effect,
exist. These are called silencers.
Terminazione
Terminazione della trascrizione è meno compresa che nei procarioti
Le differenti RNA polimerasi eucariotiche utilizzano meccanismi diversi per
la terminazione
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Processamento dell’mRNA
eucariotico
1. Aggiunta del cappuccio all’estremità 5’
2. Aggiunta della coda di poliA all’estremità 3’
3. Rimozione degli introni e giunzione degli esoni
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Aggiunta del cap in 5’
Subito dopo l’inizio della trascrizione, la 7metilguanina è aggiunta all’estremità 5’ del
trascritto attraverso un legame covalente 5’-5’
Il cappuccio serve:
-a proteggere l’mRNA dalla degradazione
-a posizionare correttamente l’mRNA sui
ribosomi
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Aggiunta della coda poli A
La lunghezza della coda di poli A aggiunta varia tra le 50 e le 200 adenine
La coda di poli A conferisce stabilità agli mRNA, aumentandone la vita media
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Gene della distrofina
Localizzato sul cromosoma X
Gene 2.000.000 basi
Proteina 4.000 AA = 12.000 basi
Per quale motivo è così voluminoso?
Concetto di COLINEARITA’: corrispondenza diretta DNAproteina
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Gli introni
Gli introni sono sequenze di un gene che
vengono trascritte ma che vengono
rimosse durante la maturazione dell’RNA.
Esse quindi non sono tradotte in proteine
Negli eucarioti la sequenza di un gene
contenente introni e quella del suo
mRNA maturo non sono colineari
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Gli INTRONI
Sono presenti quasi esclusivamente nei geni eucariotici
• Variano per numero (nell’uomo da 0 a 60 per gene)
• Variano per lunghezza (da 200 fino a 50.000 nucleotidi)
• Sono in genere più lunghi degli esoni
• Negli eucarioti superiori il numero medio di introni per gene aumenta
all’aumentare della complessità degli organismi
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Lo splicing avviene nel nucleo
Y = pirimidina, R= purina, N= qualsiasi base, A= adenina. Punto di ramificazione: A presente fra 18 e 40
basi dallo splicing 3’
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
L’RNA messaggero:
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Gene → pre-mRNA → mRNA
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
http://jcs.biologists.org/cgi/reprint/117/26/6261
Da una molecola di pre-mRNA si
possono ottenere diverse
molecole di mRNA maturo in
seguito a combinazioni diverse
degli esoni
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Da una molecola di pre-mRNA
si possono ottenere diverse
molecole di mRNA maturo in
seguito a combinazioni diverse
degli esoni
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Pre-mRNA codificato dal gene della calcitonina
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
EDITING dell’RNA
Osservato per la prima volta nel 1986 quando
si è visto che la sequenza dell’RNA differiva da
quella del DNA stampo.
Avviene o mediante RNA guida o mediante la
deaminazione della citosina, che genera
uracile.
• RNA guida
• Deaminasi (C->U)
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Differenze nella trascrizione tra procarioti ed eucarioti
Procarioti
Eucarioti
1. Un unico tipo di RNA polimerasi
2. L’mRNA viene tradotto mentre la
trascrizione è in corso
3. I geni non sono interrotti
4. Gli mRNA sono spesso policistronici
1. Tre diverse RNA polimerasi
2. L’ mRNA viene maturato, trasportato nel
citoplasma e poi tradotto
3. I geni contengono introni ed esoni
4. Gli mRNA sono monocistronici
I geni
• Un gene - un carattere
Gregor Mendel; seconda metà dell’800
• Un gene - un enzima
• Un gene - una proteina
Archibald Garrod; prima metà del 900
George Beadle ed Edward Tatum
• Un gene - un polipeptide
(+ subunità per alcuni enzimi)
• Un gene - un RNA
Il gene è l’unità di trascrizione, costituita da un segmento di DNA che
codifica per una molecola di RNA e dalle sequenze necessarie alla sua
trascrizione
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Nuovo concetto di gene
Il gene è l'unità ereditaria degli organismi viventi. I geni sono contenuti nel
genoma di un organismo, che può essere composto di DNA o di RNA, e dirigono lo
sviluppo fisico e comportamentale dell'organismo.
La maggior parte dei geni codifica proteine, che sono le macromolecole
maggiormente coinvolte nei processi biochimici e metabolici della cellula. Molti
geni non codificano proteine, ma producono RNA non codificante, che può
giocare un ruolo fondamentale nella biosintesi delle proteine e nell'espressione
genica.
A modern working definition of a gene is "a locatable region of genomic sequence,
corresponding to a unit of inheritance, which is associated with regulatory regions,
transcribed regions, and or other functional sequence regions”
Rather than striving to reach a single definition — and coming to blows in
the process — most geneticists are instead incorporating less ambiguous
words into their vocabulary such as transcripts and exons. When it is used,
the word 'gene' is frequently preceded by 'protein-coding' or another
descriptor.
(http://www.nature.com/nature/journal/v441/n7092/full/441398a.html)
IL CODICE GENETICO E LA
TRADUZIONE
Le molecole richieste per la traduzione
• mRNA
• Ribosomi
• tRNA + aa
• Enzimi vari
I Ribosomi
I ribosomi sono formati da tre molecole di RNA ribosomale e da proteine che si
associano a formare due subunità.
Il ribosoma batterico ha una massa di circa 2700 kDa ed un coefficiente di
sedimentazione di 70 S. Esso si può suddividere in due parti o subunità, una
più grande di 50 S ed una più piccola di 30 S:
•
•
la subunità grande di 50 S è costituita da almeno 34 proteine (L1-L34) e due molecole di RNA (23
S e 5 S),
la subunità piccola di 30 S è costituita da almeno 21 proteine (S1-S21) ed un RNA di 16 S.
Il ribosoma eucariote (fatta eccezione per quelli contenuti nei mitocondri e nei
cloroplasti) ha una massa molecolare di 4000 kDa ed un coefficiente di
sedimentazione di 80 S. Anch'esso è composto da due subunità, maggiore di
60 S e minore di 40 S:
•
•
la subunità maggiore è costituita da tre molecole di rRNA, una di 28 S, una di 5,8 S e un'ultima di
5 S.
la subunità minore è costituita di una sola catena di RNA 18 S.
Nel complesso le due subunità presentano più di 80 proteine.
I Ribosomi
rRNA
Proteine
Subunità
Ribosomi
assemblati
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
The translation apparatus cannot go
past a nonsense codon (UAG in this
case), because there is no tRNA that can
recognize the UAG triplet. This leads to
the termination of protein synthesis and to
the subsequent release of the polypeptide
fragment
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Solo quando una proteina assume la sua corretta struttura
tridimensionale, o conformazione, e’ in grado di
funzionare efficacemente. La funzione dipende dalla
struttura tridimensionale e la struttura tridimensionale e’
determinata dalla sequenza aminoacidica
Tutte le proteine presentano ad un’estremita’ un gruppo
aminico libero (–NH2) (estremità N-terminale) e all’altra un
gruppo carbossilico libero (-COOH) (estremità Cterminale).
Le catene polipeptidiche si accrescono per aggiunta di
singoli residui aminoacidici a partire dalla metionina Nterminale fino all’estremo C-terminale. Spesso enzimi
proteolitici eliminano poi questa metionina.
Alcune proteine sono costituite da
subunità codificate da geni diversi
Ipotesi un gene – un enzima -> un gene – un polipeptide
Le caratteristiche del CODICE
GENETICO
Il codice genetico rappresenta la corrispondenza fra una serie di sequenze di 3
nucleotidi e gli specifici aminoacidi. Quasi tutti gli esseri viventi usano il
medesimo codice genetico, chiamato codice genetico standard.
La sequenza di nucleotidi dell’mRNA si legge in gruppi di tre basi ciascuno, chiamati
codoni. Ad ogni codone corrisponde uno specifico aminoacido, si dice quindi che il codone
codifica per quell'aminoacido.
Il codice genetico è:
• Degenerato
• Non sovrapposto
• Universale (o quasi)
CODICE DEGENERATO
20 aminoacidi
64 possibili codoni
Ogni amminoacido è codificato da
una sequenza di tre nucleotidi
consecutivi detta codone. Il codice
genetico è costituito da 64 codoni,
di cui 61 sono senso, ossia
corrispondono ad amminoacidi, e 3
codoni sono non senso: segnali di
terminazione della traduzione
Degenerato significa che gli
aminoacidi
possono
essere
codificati da più di un codone
(codoni sinonimi).
Solo il triptofano e la metionina
sono codificati da una sola tripletta
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Il vacillamento
Ci sono 30-50 tRNA diversi nelle cellule, ma 64 triplette possibili: vacillamento.
Es. Alanina codificata da GC + U,A,C o G. I legami a idrogeno delle prime due basi
sono forti abbastanza da permettere un appaiamento non convenzionale sulla terza
base
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
CODICE NON SOVRAPPOSTO
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Il codice genetico è (quasi)
UNIVERSALE
Il codice genetico per il DNA cromosomale e’ universale, cioè, è comune dai
procarioti piu’ semplici agli eucarioti piu’ complessi. Fa parzialmente
eccezione il DNA mitocondriale nel quale il codone UGA viene letto come
Triptofano (invece che stop) e il codone AUA come Metionina (invece che
Isoleucina).
Il codice genetico è (quasi) universale
Negli eucarioti, l’mRNA prodotto nel nucleo è
trasportato nel citoplasma per la traduzione
Il DNA contenuto negli organelli cellulari
DNA mitocondriale
DNA cloroplastico
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
I genomi degli organuli
• Piccoli, ma essenziali
• Elevato numero di copie
• Organizzati in complessi nucleoproteici (nucleoidi)
• Eredità non mendeliana
• Informazione genetica necessaria ma non sufficiente per la completa
funzionalità dell’organulo
• Interazione fra i sistemi genetici degli organuli e del nucleo. Per es.,
geni per molte proteine ed enzimi strutturali dei mitocondri sono in realtà
codificati da DNA nucleare, tradotti dai ribosomi e trasportati nei
mitocondri
DNA mitocondriale
umano
16.569 bp
Codifica per 37 geni (che codificano
per 13 proteine, 22 tRNA e 2 rRNA),
coinvolti nella produzione di proteine
necessarie alla respirazione cellulare
Le mutazioni del DNA mitocondriale possono
portare ad un gran numero di malattie, tra le quali
l'exercise intolerance e la sindrome di KearnsSayre (KISS), che causa la perdita della piena
funzionalità nei movimenti di cuore, occhi e
muscoli.
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
DNA mitocondriale di
lievito
78.000 bp
Codifica per 2 rRNA, 25 tRNA e 16 proteine
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
DNA cloroplastico
IL GENOMA CLOROPLASTICO
120-160 kbp
Due sequenze ripetute invertite (6-76
kbp), assenti nelle Conifere e alcune
Leguminose
Circa 100 geni suddivisi in due gruppi:
• Geni per trascrizione,
traduzione
• Geni per le proteine dei
complessi delle membrane
tilacoidali
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Omoplasmia ed
eteroplasmia
Si definisce eteroplasmia la coesistenza di
diversi genomi mitocondriali (un genoma
"selvatico" o "wild-type", cioè senza
mutazioni, ed uno o più genomi mutati)
all'interno dei mitocondri di cellule diverse
(eteroplasmia intercellulare) o, addirittura,
all'interno
di
una
stessa
cellula
(eteroplasmia intracellulare).
Si definisce omoplasmia la presenza,
all'interno di una cellula, del solo DNA
mitocondriale normale o del solo DNA
mitocondriale mutato.
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Eredità materna
Virtualmente tutti i mitocondri dello zigote derivano
dall’oocita e perciò la modalità di trasmissione delle
mutazioni mt differisce dalla trasmissione mendeliana
classica:
madre portatrice → trasmissione a tutta la progenie, ma
solo le figlie femmine possono trasmettere la mutazione
ai loro figli.
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
I mitocondri contenuti nello sperma dei mammiferi non entrano nella cellula uovo, in
quanto le modalità di penetrazione dello spermatozoo e la costituzione anatomica dello stesso
consentono l'ingresso della sola testa, pertanto i mitocondri che hanno sede nel corpo non
vengono inseriti. In alcuni casi alcuni mitocondri paterni possono penetrare tuttavia vengono
distrutti dalla cellula uovo subito dopo la fecondazione.
In rari casi i mitocondri possono essere ereditati dal padre, ad esempio nelle banane
L'ipotesi che il DNA mitocondriale umano fosse ereditato dalla madre, spinse i ricercatori a
tracciare la linea uterina già molto tempo fa (anche il cromosoma Y, ereditato dal padre, viene
utilizzato in un modo analogo per studiare la linea maschile). Questo è completato, negli esseri
umani, dal sequenziamento di una o più regioni ipervariabili (HVR1 o HVR2) del mtDNA.
Il tentativo della teoria dell'Eva mitocondriale di scoprire l'origine dell'umanità si basa sullo
stesso tipo di analisi. In particolare, studi sul DNA mitocondriale umano hanno permesso al
genetista inglese Bryan Sykes di chiarire le modalità con cui le popolazioni agricole si sono
diffuse dal Medio oriente all'Europa preistorica popolata da cacciatori raccoglitori, oltre all'origine
delle popolazioni polinesiane, dimostrata essere nel sud est asiatico: questi e molti altri risultati
della tecnica del mtDNA sono esposti nel volume "Le sette figlie di Eva. Le comuni origini
genetiche dell'umanità". Saggi Mondadori 2003.
MUTAZIONI GENICHE
Pierce: per mutazione si intende il cambiamento dell’informazione genetica che
viene ereditato (? No way!). Per mutazione genetica si intende ogni modifica
stabile ed ereditabile nella sequenza nucleotidica di un genoma o più
generalmente di materiale genetico
• Mutazioni somatiche
• Mutazioni della linea germinale
Nel corpo umano vi sono circa 1014 cellule, freq mutazione 1 ogni c.a. 10-6 divisioni
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
MUTAZIONI PUNTIFORMI
Per mutazione si intende la modificazione della sequenza codificante di un gene.
• Sostituzioni: quando una base viene sostituita da un’ altra
• Inserzioni: aggiunta di una o più basi
• Delezioni: rimozione di una o più basi
Transizione e Trasversione
Le mutazioni per sostituzione di basi sono dette transizioni quando una purina è
sostituita da un’altra purina o una pirimidina è sostituita da un’altra pirimidina; sono
dette trasversioni quando una purina è sostituita da una pirimidina o viceversa.
Single Nucleotide Polymorphism (SNP)
InDel in fase se riguarda intere triplette
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Espansione della ripetizione di trinucleotidi
Causa mutazioni in FMR-1, responsabile della sindrome del cromosoma X fragile (causa
del ritardo mentale) ripetizione di CGG: 60 ripetizioni nel normale, centinaia/migliaia
nel mutato
La Sindrome dell’ X-Fragile è la causa di ritardo mentale ereditario più frequente. Circa 1:4000
maschi nella popolazione generale sono affetti dalla sindrome.
E’ meno frequente nelle femmine: infatti queste ultime possedendo 2 cromosomi X hanno
anche una copia del gene che può funzionare correttamente. Lo sviluppo mentale delle persone
affette da FraX è molto vario. Alcune mostrano capacità cognitive quasi normali, altre un lieve
ritardo mentale, altre ancora un ritardo mentale più grave.
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Lo slittamento dei filamenti
durante la replicazione è alla
base dell’espansione della
ripetizione dei trinucleotidi
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
EFFETTI FENOTIPICI DELLE MUTAZIONI
PUNTIFORMI
Mutazione in avanti: altera il fenotipo selvatico
Mutazione inversa: (retromutazione): ripristina il fenotipo selvatico
Mutazione soppressiva: rispristina il fenotipo selvatico con una seconda
mutazione
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Mutazione soppressiva intragenica
E’ una mutazione (v.) che sopprime il fenotipo dovuto ad un’altra mutazione, così che
le due insieme danno luogo ad un fenotipo normale. Entrambe le mutazioni sono a
carico dello stesso gene
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Mutazione soppressiva intergenica
The amber mutation (Alteration of a codon to UAG, one of the three codons that result in premature polypeptide chain
termination in all living organism) replaces a wild-type codon with the chain-terminating nonsense codon UAG. The
suppressor mutation in this case produces a tRNATyr with an anticodon that recognizes the mutant UAG stop codon. The
suppressed mutant thus contains tyrosine at that position in the protein.
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
Mutazione nonsenso (amber): mutazione puntiforme che converte un
codone qualsiasi in un codone di stop e quindi si produce l’interruzione prematura
della catena polipeptidica. A volte può essere eliminata da geni soppressori che codificano per
tRNA mutanti. Affinche’ le cellule sopravvivano devono pero’ essere presenti anche i tRNA normali.
Mutazione missenso:
a seguito di una sostituzione nucleotidica viene
codificato un aminoacido differente. La funzione della proteina può però non essere
compromessa (mutazione neutra)
Mutazione silente:
la sostituzione nucleotidica non varia l’aminoacido
codificato (codice degenerato)
Mutazione neutra:
a seguito di una sostituzione viene inserito un aminoacido
differente senza però alterare la funzionalità della proteina
Mutazione con perdita di funzione: recessiva, altera il fenotipo selvatico
Mutazione con acquisto di funzione:
dominante, altera il fenotipo
selvatico
Mutazione condizionale: espressa solo in determinate condizioni
Mutazione letale: causa la morte
Cause delle mutazioni
• Mutazioni spontanee: dovute a cambiamenti naturali nella struttura del DNA
• Mutazioni indotte: causate da agenti chimici (bromuro di etidio, UV) o radiazioni
Tasso di mutazione
Frequenza con cui il gene cambia da tipo selvatico a mutante. E’ espressa come
numero di mutazioni per unità biologica (divisone cellulare, gamete o ciclo di
replicazione. Es. 1 ogni 100.000 gameti)
Frequenza di mutazione
Incidenza di un tipo specifico di mutazioni all’interno di un gruppo di organismi (es.
1 persona su 20.000)
Il test di Ames
The Ames test uses a battery of strains of
Salmonella typhimurium, all of which carry a
different kind of mutation in the histidine (his)
biosynthetic pathway. Hence these strains of
Salmonella are auxotrophs because they require
the amino acid histidine in the medium in order to
grow.
The mutations that affects the his gene are a
variety of in-frame mutations (such as base-pair
substitutions) and frameshift mutations (such as
base-pair deletions or insertions). Since there is
always a low level of mutations occurring these
strains can revert to his + at a low frequency (back
to its prototroph). This type of reversion is called a
back mutation. If cells are exposed to a mutagen,
the frequency of mutation increases. Hence one
can easily measure the frequency of reversion of
Salmonella typhimurium from his- to his+ to
determine if an unknown chemical is a possible
mutagen. A mutagen will increase the
frequency of reversion
Pierce, GENETICA, Zanichelli editore S.p.A. Copyright © 2005
From The RED BOOK BULLETIN of
Current Protocols in Molecular Biology: