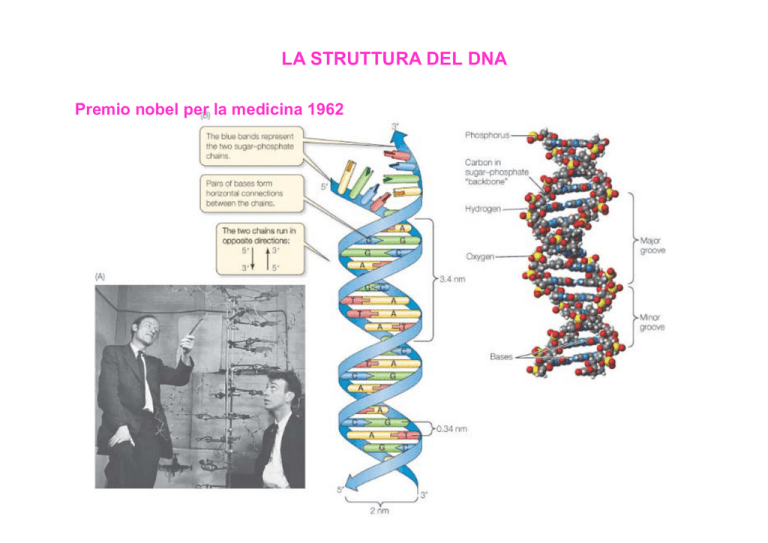
LA STRUTTURA DEL DNA
Premio nobel per la medicina 1962
STRUTTURA DNA (acido deossiribonucleico)
BASI AZOTATE
adenina (A)
citosina (C)
guanina (G)
timina (T).
Adenina e guanina sono composti eterociclici chiamati purine, mentre citosina e timina
sono anelli pirimidinici.
Doppia elica antiparallela
Una sequenza di DNA è definita senso
se la sua sequenza è la stessa del
relativo mRNA. La sequenza posta sul
filamento opposto è invece detta
antisenso. Dal momento che le RNA
polimerasi lavorano producendo una
copia complementare, il filamento
necessario per la trascrizione è
l'antisenso.
gruppo fosfato
deossiribosio (zucchero pentoso)
base azotata
Si forma un legame covalente tra il gruppo fosfato di un nucleotide e lo zucchero del nucleotide
precedente
I NUCLEOTIDI
BASE AZOTATA
Il DNA è un polimero
I suoi monomeri
sono i NUCLEOTIDI
I nucleotidi sono formati
Gruppo fosfato
zucchero
Gruppo fosfato
zucchero deossiribosio
base azotata
Le basi azotate
PURINE
PIRIMIDINE
La complementarietà
una purina si appaia con una
pirimidina
L’adenina si appaia con la
timina (2 legami H)
La guanina si appaia con la
citosina (3 legami H)
Le 2 catene di
nucleotidi si
avvolgono l’una
sull’altra a spirale
per questo la
molecola di DNA
viene chiamata
la DOPPIA ELICA
•la molecola può venire duplicata facilmente
•il DNA viene utilizzato dai viventi come un codice
per registrare e trasmettere informazioni
Funzioni del DNA
• Materiale ereditario: viene duplicato e
trasmesso alle cellule figlie
• Programma della cellula: contiene le
informazioni per la sintesi di tutte le proteine
dell’organismo
• Ogni tratto di DNA che porta l’informazione per
la sintesi di una proteina→GENE
• Nel DNA delle cellule della specie umana ci
sono circa 30.000 geni, in quello di un batterio,
poche migliaia
REPLICAZIONE SEMICONSERVATIVA
È il processo attraverso il quale da una molecola madre si formano due molecole figlie ciascuna formata da
un’elica vecchia e un’elica nuova. La duplicazione del Dna avviene nella fase S (sintesi) del ciclo cellulare.
L’enzima che permette questo processo si chiama DNA polimerasi. Una volta che è avvenuta la
duplicazione del DNA la cellula deve dividersi.
Nei batteri il DNA si duplica a partire da una
forcella di replicazione in modo bidirezionale
fino a quando tutta la molecola si è duplicata
Meselson e Stahl fecero crescere dei batteri in un terreno di coltura ricco dell'isotopo pesante 15N. Questi microorganismi
metabolizzarono l'15N che quindi venne ad essere introdotto in molte molecole biologiche; tra queste molecole bisogna
ricordare le basi azotate del DNA.
In questo modo il DNA presente nei batteri era un "DNA pesante", poiché inglobava atomi di azoto più pesanti della norma.
Alcuni batteri furono poi prelevati, lisati e, con opportune tecniche di laboratorio, venne estratto il loro DNA.
Quest'ultimo venne aggiunto ad una provetta contenente una soluzione concentrata di Cloruro di Cesio (CsCl 6M).
La provetta fu poi centrifugata. In queste condizioni nella provetta si forma un gradiente di densità dal momento che il CsCl
tende a concentrarsi verso il fondo della stessa.
•Una volta che si è formato il gradiente di densità, il DNA migra per fermarsi
nella regione della soluzione che ha densità uguale alla sua.
Il DNA può essere messo in evidenza in seguito all'introduzione di particolari
sostanze che si legano ad esso e divengono visibili se illuminate da luce
ultravioletta.
Ciò che si vide sperimentalmente fu una banda verso il fondo della provetta.
•A questo punto alcuni batteri furono trasferiti dal primo terreno di coltura in un
nuovo terreno "standard", in cui cioè era presente 14N anziché 15N. I
microorganismi metabolizzarono l'azoto includendolo nelle basi azotate dei
nucleotidi che andranno a formare (tra le altre cose) le nuove eliche di DNA.
•Alcuni batteri furono prelevati dal terreno "standard", furono lisati e venne
estratto il DNA.
•In seguito ad una centrifugazione in gradiente di densità si ottenne un'unica
banda posta in posizione superiore rispetto a quella del caso precedente: il
DNA era quindi più leggero.
•Trascorso il tempo necessario per la successiva replicazione del DNA venne
ripetuta la suddetta procedura.
In questo caso si ottennero due bande: la prima in posizione nettamente
superiore a quelle ottenute nei casi precedenti, la seconda nella stessa
posizione di quella dell'esperimento precedente.
A questo punto possiamo identificare:
un DNA "pesante". In cui per forza erano presenti due filamenti 15N.
un DNA "medio". In cui per forza era presente un filamento 15N e un filamento
14N.
un DNA "leggero". In cui per forza erano presenti due filamenti 14N.
…….l'unico modello in grado di spiegare i "pesi" delle varie molecole di DNA era quello della replicazione
semiconservativa
Ingredienti della replicazione del DNA
REPLICAZIONE DNA PROCARIOTICO
1 ORIGINE DI REPLICAZIONE
PROCESSO BIDIREZIONALE
CI SONO 2 FORCELLE DI REPLICAZIONE CHE SI MUOVONO IN VERSI OPPOSTI E CHE AL TERMINE DEL
PROCESSO SI “INCONTRANO
L'origine di replicazione di Escherichia coli, nota come oriC, consiste di
ripetizioni di una sequenza di nucleotidi.
La DnaA è una proteina deputata al riconoscimento e al legame di tali
ripetizioni.determina la denaturazione della regione ricca in A/T e crea
quindi regioni a singolo filamento
La DNA ELICASI separa i filamenti dall’origine in direzione 5’-3’
Ci sono proteine che impediscono che le eliche si richiudano.
La DNA POLMERASI e’ l’enzima che catalizza la polimerizzazione dei
desossiribonucleotidi. catalizza il legame covalente tra il gruppo fosfato
di un nucleotide e lo zucchero del nucleotide precedente
DNA polimerasi I meccanismo di riparazione
DNA polimerasi II meccanismo di riparazione
DNA polimerasi III sintesi dei filamenti di DNA. Estende la catena aggiungendo un
nucleotide per volta all’estremo 3’-OH
Le DNA polimerasi
batteriche hanno
un’attività esonucleolitica
3’-5’ (direzione opposta a
quella di sintesi)
La sintesi del DNA inizia in un punto in cui la molecola viene
preventivamente srotolata chiamato origine. Si forma così un'ansa di
replicazione alla cui estremità troviamo le "forcelle di replicazione" e la
sintesi può proseguire in due direzioni. Nel DNA batterico circolare, le
forcelle si incontrano in un punto della circonferenza opposto
all'origine.
Poiché le eliche sono antiparallele, ne consegue che un'elica, chiamata catena
leader, o stampo, viene letta e copiata nella direzione di avanzamento della
forcella e procede in modo continuo e veloce (un unico innesco a RNA). L'altra
elica dovrà essere sintetizzata con ritardo e in modo discontinuo dovendo
attendere lo srotolamento dell'elica veloce. Okazaki, biologo giapponese, scoprì
che la catena discontinua viene sintetizzata, con frammenti, in direzione opposta
all'avanzamento della forcella.
E' da tenere presente che ogni frammento di Okazaki termina con un'estremità
3'OH libera
Ha una struttura asimmetrica:
– leading strand sintetizzato con continuità
– Lagging strand sintetizzato con discontinuità
•Le DNA polimerasi non possono sintetizzare catene di DNA senza un primer, un piccolo segmento
di acido nucleico con un gruppo ossidrilico libero in posizione 3’-OH.
•Le DNA polimerasi sono capaci di leggere nucleotidi solo nella direzione 5’-3’
Durante la replicazione del materiale genetico, il primer è costituito da RNA.
Una RNA polimerasi DNA-dipendente, detta primasi, sintetizza un piccolo tratto di RNA
complementare allo stampo molecolare, utilizzato come primer dalla DNA polimerasi .
Una volta iniziata la sintesi di DNA, il primer di RNA viene digerito da enzimi con attività 5’ →3’
esonucleasica.
La reazione di polimerizzazione richiede i quattro precursori attivati, dATP, dGTP, dCTP e dTTP ed
avviene direttamente sulla catena di DNA stampo.
Nel filamento guida la DNA polimerasi può attaccare i nucleotidi in direzione 5’-3’ a partire
dall’innesco
Nel filamento lento la sintesi procede sempre in direzione 5’-3’ ma in direzione opposta a quella
della forcella di replicazione
LE PROTEINE “INIZIATRICI” SI LEGANO CON INTERAZIONI
SEQUENZE SPECIFICHE ALLE ORIGINI DI REPLICAZIONI
RICCHE IN A-T
Il legame dei dNTP allo stampo avviene mediante i legami idrogeno, ma anche la forma delle basi e
controlli sterici nel sito attivo della DNA-polimerasi, concorrono al corretto appaiamento delle basi
La frequenza di errore viene ridotta anche grazie ad accurati meccanismi enzimatici.
Tutte le DNA polimerasi possiedono un’attività esonucleasica 3’→5’ che consente di rimuovere un
nucleotide appena inserito Tale attività di “proof reading” (lettura delle bozze) è altamente specifica
per gli appaiamenti sbagliati e aggiunge un ulteriore fattore di accuratezza che riduce la frequenza di
errore totale.
La DNA polimerasi è in grado di rimuovere gli inneschi a RNA e di riempire i gap con il DNA. I
frammenti vengono poi uniti dalla DNA ligasi
Dalla parte opposta rispetto a Ori C il cromososma di E. Coli contiene un paio di sequenze di
terminazione TER
Una proteina si lega a queste sequenze e vengono bloccate le forcelle di replicazione
La DNA ligasi lega tutti e quattro i filamenti di DNA creando due molecole circolari a doppio
filamento concatenate.
Le topoisomerasi sono enzimi che provocano delle rotture temporanee nei filamenti di DNA e poi li
riuniscono dopo la formazione di due molecole circolari distinte
L'elicasi è un enzima che agisce rompendo i legami idrogeno instaurati tra le basi complementari.
Inoltre questo enzima agisce in coppia con la DNA-girasi che separa i 2 filamenti di DNA.
Le topoisomerasi determinano un aumento o una diminuzione del grado di superavvolgimento. Possono
operare tagli a singolo o doppio filamento. Tali enzimi svolgono un ruolo fondamentale
nell'impacchettamento e nella replicazione del DNA.
Primasi:All'inizio della replicazione del DNA, le primasi legano le elicasi a formare un complesso
chiamato primosoma. Attivate proprio dalle elicasi, le primasi sintetizzano una breve molecola di RNA
(detta appunto primer), complementare alla molecola di DNA adiacente. Il primer viene utilizzato poi
come punto di partenza per le DNA polimerasi.
Ligasi: lega due frammenti di DNA che hanno subito una rottura a doppio filamento (ad esempio nei
processi di riparazione del DNA).
Può anche legare una rottura a singolo filamento, come ad esempio accade durante il processo di
replicazione del DNA, nel quale la DNA ligasi agisce sul filamento che ha la direzione 3'->5', sintetizzato a
frammenti .
Questo enzima catalizza infatti la formazione di legami covalenti fosfodiesterici tra nucleotidi di DNA
adiacenti (lega l'estremità 3' del nuovo tratto di DNA con l'estremità 5' del tratto precedentemente
sintetizzato).
ATP + (deossiribonucleotide)n + (deossiribonucleotide)m = AMP + difosfato +
(deossiribonucleotide)n+m
Il processo di replicazione del DNA nei procarioti e negli eucarioti è molto simile,
ma nelle cellule eucariote la quantità di DNA è maggiore ed è organizzato in
strutture lineari
Esistono 2 tipi di cromatina:
1)EUCROMATINA (+dispersa e si colora debolmente) e
2)ETEROCROMATINA (+ condensata si colora + intensamente).
I Genomi degli Eucarioti:
Eucromatina ed Eterocromatina
Eucromatina: regioni cromosomiche non condensate, attivamente trascritte e ad
alta densità genica (fibre 30 nm).
Eterocromatina: (facoltativa o costitutiva): cromatina mediamente o altamente
condensata e generalmente non trascritta, ad alta percentuale di sequenze
ripetute e contenuto di geni relativamente basso. Comprende regioni telomeriche
e centromeriche.
In una tipica cellula di mammifero il contenuto di eterocromatina è pari a circa il 10%. in
Drosophila melanogaster costituisce il 34% del genoma totale.
24
I Genomi degli Eucarioti: contenuto in DNA
Si osserva che il contenuto totale di DNA negli eucarioti e quindi la
dimensione del genoma è correlata alla complessità dell’organismo (ad
es., il genoma umano è più grande di quello degli insetti che è a sua volta
più grande di quello funghi).
Esistono però diverse eccezioni: es. il genoma di X. laevis è grande quanto
quello dei mammiferi; altri anfibi hanno un genoma circa 50 volte più
grande del genoma umano; tra le piante, il genoma di Zea Mais è più
grande di quello umano.
In genere, per un dato raggruppamento tassonomico, la dimensione del
genoma è approssimativamente proporzionale alla complessità
dell’organismo
25
Paradosso del valore C
La mancanza di correlazione tra la complessità genetica/morfologica di
un organismo e le dimensioni del suo genoma è definita
Paradosso del valore C.
Il Paradosso del valore C è spiegato considerando:
•
•
le dimensioni dei geni discontinui
(numero - grandezza introni)
La quantità di DNA ripetitivo, che rappresenta il componente più
abbondante della porzione di DNA non codificante dei genomi
eucariotici
REPLICAZIONE DNA EUCARIOTICO
PIU’ ORIGIN DI REPLICAZIONE ARS (sequenza replicante autonoma ricca in A-T))
Ogni origine di replicazione prende il nome di replicone, da cui il processo si avvia in maniera
bidirezionale
Riduzione del tempo di replicazione
LE FORCELLE REPLICATIVE ENTRANO IN CONTATTO TRA LORO
5 DNA POLIMERASI:
αβδε: agiscono nel nucleo
γ: agisce nei mitocondri
α,δ: replicazione DNA cromosomico
β, ε: riparazione danni al DNA
Sintesi di istoni che formano ottameri e si associano al DNA nella
forcella replicativa
Il telomero è la regione terminale del cromosoma, composta di DNA altamente ripetuto, che non
codifica per alcun prodotto proteico.
Ha un ruolo determinante nell'evitare la perdita di informazioni durante la duplicazione dei
cromosomi.
Le sequenze telomeriche variano da organismo in organismo.
Il DNA telomerico è sintetizzato dalle telomerasi, DNA polimerasi RNA-dipendenti altamente
specializzate che introducono centinaia di ripetizioni nucleotidiche alle estremità dei cromosomi.
E’ una classe di retrotrascrittasi specializzate, presenti in numerosi organismi (tra cui l'uomo), ma
non in tutti (e soprattutto non in tutte le fasi dello sviluppo). Utilizza frammenti di RNA come
stampo per l'elongazione dei telomeri.
E’ capace di allungare l'estremità 3'-OH del filamento stampo della catena tardiva.
Contiene un sito catalitico che polimerizza i desossiribonucleotidi complementari ad un RNA
stampo, che essa stessa contiene.
In particolare, nell'uomo le telomerasi sono attive solo nelle cellule della linea germinale: ciò
significa che, ad ogni replicazione, i telomeri umani si accorciano di un certo numero di paia di
basi.
La DNA polimerasi non è in grado di replicare il cromosoma fino alla sua terminazione; se non ci
fossero i telomeri, che quindi vengono accorciati ad ogni replicazione, la replicazione del DNA
comporterebbe in ogni occasione una significativa perdita di informazione genetica.
I telomeri si accorciano a causa del meccanismo di replicazione del filamento lagging del DNA.
l’ultimo primer di RNA viene tagliato ma non sostituito con il DNA
Questo genera un continuo processo di accorciamento di queste regioni
SEQUENZA DI RNA COMPLEMENTARE ALLE
SEQUENZE RIPETUTE NEI TELOMERI
LA TELOMERASI SI LEGA ALL’ESTREMITA’ 3’ A SINGOLO FILAMENTO DEL CROMOSOMA
L’ALLUNGAMENTO DEL 3’ DEL TELOMERO CREA LO SPAZIO PER LA
FORMAZIONE DI UN NUOVO FRAMMENTO DI OKAZAKI
Esistono teorie che associano il continuo accorciarsi dei telomeri con la senescenza delle
cellule della linea somatica e con la prevenzione del cancro. Questo perché i telomeri
agirebbero come una sorta di orologio biologico, legato cioè ad un numero massimo di
mitosi (e di replicazioni del DNA), al termine del quale la cellula sarebbe troppo vecchia per
essere mantenuta in vita e prenderebbe la via dell‘apoptosi.
La telomerasi è espressa dalle cellule embrionali ma non dalle cellule somatiche dell’organismo
Adulto
Le uniche eccezioni sono costituite dalle cellule germinali e da quelle attivamente proliferanti
(cellule staminali, strati basali epiteliali
il mantenimento della lunghezza dei telomeri è un segno distintivo di molti tipi di cancro nei
mammiferi. Nell'uomo, ad esempio, numerosi tumori sono in grado di aumentare l'attività
della telomerasi, ottenendo una capacità di replicazione pressoché infinita. Altri tipi di
carcinoma, invece, sono in grado di avviare pathway alternativi di allungamento dei telomeri
Tutte le cellule possono dividersi un numero
definito di volte: CPD (Divisioni Cellulari
Permesse)
Una volta raggiunto il CPD la cellula
entra nella “senescenza replicativa”,
cioè l’incapacità di
proliferare ulteriormente
Interviene il processo di
APOPTOSI,
che porta
alla morte della cellula
La lunghezza dei telomeri determina il numero di divisioni cellulari permesse. Il CPD dipende
dal tipo di cellula e dal tessuto di cui essa fa parte. Quando i telomeri raggiungono una
lunghezza “critica” la cellula entra nella senescenza replicativa perchè viene emesso un
segnale che impedisce alla cellula di dividersi ancora.
Se vengono a mancare i sistemi di controllo della p53 o della p16/Rb, la cellula supera la fase di
senescenza e può riattivare la telomerasi diventando immortale
Nelle cellule cancerose:
L’attività della telomerasi si
incrementa dell’80-90%
I telomeri sono più corti del normale
Per mutazione genetica si intende ogni modificazione stabile ed ereditabile nella
sequenza nucleotidica di un genoma o più generalmente di materiale genetico (sia
DNA che RNA) dovuta o al caso, ma non alla ricombinazione genetica. Una
mutazione modifica quindi il genotipo di un individuo e può eventualmente
modificarne il fenotipo a seconda delle sue caratteristiche e delle interazioni con
l'ambiente.
Le mutazioni spontanee sono mutazioni provocate da fattori chimici endogeni e da errori nei processi che si attuano sul
materiale genetico; la definizione di mutazione spontanea è di mutazione che avviene in assenza di agenti mutageni noti. Non
sono molto frequenti, ma sono comunque inevitabili vista la intrinseca imperfezione di ogni meccanismo molecolare.
Gli errori possono essere dovuti a:
Reazioni che trasformano una base azotata in una diversa
Errori nei processi di replicazione, della ricombinazione e della riparazione del DNA. Ad esempio può essere dovuta alla DNA
polimerasi che aggiunge nucleotidi non corretti; ciò può generare una trasversione se c'è lo scambio di una purina con una
pirimidina o viceversa; una transizione se c'è lo scambio di una purina con un'altra purina oppure di una pirimidina con un'altra
pirimidina.
Danni ossidativi
Dovuti alla formazione spontanea nella cellula di specie con atomi di ossigeno molto reattive, in grado di attaccare il DNA e
causare danni al singolo o al doppio filamento e danneggiamento delle basi azotate.
Le mutazioni indotte sono invece prodotte dall'azione di particolari agenti fisici o chimici detti appunto agenti mutageni. È detto
mutagenesi il processo che determina una mutazione indotta e mutagenizzato l'organismo in cui è stata prodotta.
Si distinguono i danni per mutazioni indotte in:
Sostituzione delle basi con molecole con struttura analoga a quelle comunemente presenti nel DNA ma che formano appaiamenti
diversi e quindi errati.
Aggiunta di gruppi sostituenti alle basi azotate: anche in questo caso generando molecole con capacità di appaiamento non
corrette.
Danneggiamento delle basi azotate
Inserzione o delezioni di basi.
I mutageni fisici sono soprattutto radiazioni ionizzanti (raggi X, raggi gamma) e non ionizzanti (raggi UV);
Gli agenti chimici sono molto numerosi e appartengono a diverse classi di composti. Oltre che per la natura i mutageni
differiscono anche per spettro mutazionale, ovvero per il tipo (o i tipi) di mutazione che possono provocare.
Una importante differenza tra mutageni fisici e chimici è che i primi agiscono indipendentemente dall'organismo; i mutageni
chimici invece possono avere effetti diversi in funzione del sistema biologico. Mentre una radiazione, infatti, colpisce direttamente
il materiale genetico, un composto chimico può interagire con altre molecole (enzimi, metaboliti, specie reattive...) presenti nella
cellula che ne possono variare le caratteristiche.
MUTAZIONI (classificazione in base ai meccanismi di
generazione):
•Inserzioni
•Delezioni
•Sostituzione di basi
inserzioni di basi:
•Riguardano uno o pochi nucleotidi
•Conseguenze gravi se nel DNA codificante
•Espansioni di triplette ripetute (CGG)
• Delezioni di basi
•Riguardano uno o pochi nucleotidi
•Conseguenze gravi se nel DNA codificante
•Piu’ comuni a livello di sequenze ripetute in tandem
(VNTR) per crossing-over ineguale
• Sostituzioni di basi:
•Transizioni = Sostituzioni di purina (A o G) con
un’altra purina (G o A) o di una pirimidina (T o C) con
un’altra pirimidina (C o T)
•Transversioni = Sostituzioni di purina (A o G) con
una pirimidina (T o C) o viceversa
• MUTAZIONI (classificazione in base alle conseguenze
funzionali)
•Sostituzione che riguarda regioni potenzialmente
rilevanti (esoni, regioni regolatrici espressione, siti di
splicing e poliadenilazione, regioni conservate in specie
diverse)
•Sostituzione che riguarda
significato funzionale
sequenze
senza
un
• MUTAZIONI (classificazione in base alle conseguenze
funzionali)
Sostituzioni in regioni potenzialmente rilevanti da un punto
di vista funzionale
•Sostituzione sinonima o silente: non determina un
cambio di aminoacido
•Sostituzione non sinonima: determina un codone
specificante un aminoacido differente rispetto a
quello originario.
Puo’ essere:
Conservativa (aminoacido simile da un punto di vista
biochimico)
Non conservativa (aminoacido diverso da un punto di
vista biochimico)
• Mutazione non senso: sostituzione di un codone che
specifica un aminoacido con un codone di fine sintesi
CODICE GENETICO
U
U
U
U
U
U
U
U
U
C
A
G
Phe
Phe
Leu
Leu
U
U
U
U
C
C
C
C
U
C
A
G
Ser
Ser
Ser
Ser
U
U
U
U
A
A
A
A
U
C
A
G
Tyr
Tyr
STOP
STOP
U
U
U
U
G
G
G
G
U
C
A
G
Cys
Cys
STOP
Trp
C
C
C
C
U
U
U
U
U
C
A
G
Leu
Leu
Leu
Leu
C
C
C
C
C
C
C
C
U
C
A
G
Pro
Pro
Pro
Pro
C
C
C
C
A
A
A
A
U
C
A
G
His
His
Gln
Gln
C
C
C
C
G
G
G
G
U
C
A
G
Arg
Arg
Arg
Arg
A
A
A
A
U
U
U
U
U
C
A
G
Ile
Ile
Ile
Met
A
A
A
A
C
C
C
C
U
C
A
G
Thr
Thr
Thr
Thr
A
A
A
A
A
A
A
A
U
C
A
G
Asn
Asn
Lys
Lys
A
A
A
A
G
G
G
G
U
C
A
G
Ser
Ser
Arg
Arg
G
G
G
G
U
U
U
U
U
C
A
G
Val
Val
Val
Val
G
G
G
G
C
C
C
C
U
C
A
G
Ala
Ala
Ala
Ala
G
G
G
G
A
A
A
A
U
C
A
G
Asp
Asp
Glu
Glu
G
G
G
G
G
G
G
G
U
C
A
G
Gly
Gly
Gly
Gly
MUTAZIONI FRAMESHIFT
Le mutazioni frameshift (dall'inglese, spostamento dell'ordine di lettura) sono dette anche
mutazioni da scivolamento. Sono causate dall'aggiunta (inserzione) o l'eliminazione
(delezione) di uno o pochi nucleotidi. Nel caso di inserzione o delezione di nucleotidi in
numero non multiplo di tre, viene alterato l'ordine di lettura di tutti i codoni successivi
nell'ordine a quello inserito o deleto. Scoperte da Crick e Brenner studiando il codice
genetico, si tratta di mutazioni quasi sempre dannose.
Gli effetti possono essere notevolmente diversi a seconda del tipo di mutazione e della posizione
in cui questa si verifica.
Una mutazione può non portare a nessuna conseguenza e questo quando interessa DNA che non
codifica (o meglio sembra non codificare) per nessun prodotto genico
Se la mutazione va invece ad alterare le sequenze codificanti, ovvero i geni, si ha una variazione
nel tipo o nella quantità del corrispettivo prodotto genico, che può essere una proteina o RNA
funzionale (rRNA, tRNA, snRNA ecc.). Parliamo in questo caso di mutazione biochimica.
se la mutazione biochimica porta a una variazione visibile del fenotipo si parla di mutazione
morfologica.
Inoltre distinguiamo, sempre in relazione agli effetti, in:
mutazione positiva: porta un vantaggio evolutivo
mutazione neutra: non risulta in un depotenziamento della capacità riproduttiva dell’individuo;
mutazione semiletale: rende più difficoltosa la perpetuazione riproduttiva dell’individuo (il tipico
esempio sono le malattie genetiche che debilitano in qualche modo l’individuo, rendendolo meno
capace di riprodursi, senza però impedirglielo totalmente)
mutazione subletale: non permette all’individuo di raggiungere l’età riproduttiva;
mutazione letale: porta alla morte dell'individuo in fase embrionale o fetale.
SISTEMI DI RIPARAZIONE
FOTODIMERIZZAZONE
Il dimero di timina si forma tra due residui di timina posti sullo stesso lato della doppia elica
del DNA, per azione della luce ultravioletta.
Il dimero causa una distorsione nella doppia elica del DNA che ne impedisce la replicazione
e l’espressione.
Questo danno viene riparato a opera di diversi enzimi (nell’ E. coli si tratta di tre attività
enzimatiche) che riconoscono la distorsione, tagliano la porzione danneggiata e
ricostruiscono le parti mancanti.
Il dimero di timina può anche essere tagliato fotochimicamente, in quanto quasi tutte le
cellule contengono un enzima fotoattivatore che si lega al dimero di timina e, assorbendo un
fotone (fotoriattivazione), taglia il dimero nelle sue basi originali, favorendo il ritorno alla
condizione iniziale.
FOTOLIASI
La fotoliasi ha alta affinità per queste strutture chimiche, alle quali si lega reversibilmente e
le ripara. Questo enzima funziona come un meccanismo di riparazione del DNA quando la
luce di lunghezza d'onda compresa fra 320 e 370 nm lo colpisce attivandolo. La reazione
enzimatica prevede la rottura del dimero e la ricostituzione della struttura corretta delle basi
(fotoriattivazione).
Deaminazione della citosina
La deaminazione ossidativa spontanea della
citosina determina la formazione di uracile, con
rilascio di ione ammonio.
Verrà quindi inserito un uracile al posto della
corretta citosina; l'appaiamento errato può
essere riconosciuto e riparato da particolari
sistemi di riparazione del DNA: in particolare
specifiche glicosidasi riconoscono questo
nucleotide che non è comune nel DNA e lo
sostituiscono con la citosina.
Deaminazione della 5-metilcitosina
La deaminazione di questa base provoca la
formazione di timina, un nucleotide
normalmente presente nel DNA. Alcune
gilcolidasi sono in grado di riconoscere gli
appaiamenti errarti che derivano da questa
deaminazione (T-G) e sostituire alla timina la
citosina. La 5-metilcitosina è presente
soprattutto nei procarioti, formata in seguito a
metilazione di una citosina tramite una metil
transferasi. La formazione di questa base
modificata è correlata a processi di
silenziamento genico. Spesso la 5-metilcitosina
è presenti nei siti CpG:
Meccanismi per escissione di basi (BER):
•Ripara il danno che coinvolge un solo nucleotide
•DNA glicosilasi riconosce e rimuove la base alterata
•L’endonucleasi rimuove lo zucchero e il gruppo fosforico
•Una DNA POLIMERASI riempie il buco
•Una DNA ligasi unisce i filamenti
Meccanismi per escissione di nucleotidi (NER):
•Ripara il danno di frammenti da 2 a 20 nucleotidi
•Utilizza differenti enzimi (nulceasi, elicasi, DNA
polimerasi, DNA ligasi) rispetto al BER e rimuove più
basi
Mismatch Repair (MMR)
corregge errori di replicazione e di ricombinazione
genetica che determinano la formazione di nucleotidi
male appaiati in seguito alla replicazione del DNA.
Meccanismi di risposta cellulare al danno del DNA
•La cellula reagisce ad un danno del DNA mettendo in opera
una serie di strategie per confinare e limitare il danno.
•In particolare si ha l’arresto del ciclo cellulare fino alla
riparazione del danno. Questo meccanismo avviene grazie ad
una serie di proteine
•La prima è chiamata ATM, un sensore intracellulare in grado
di avvertire il danno del DNA.
•ATM, rilascia un segnale ad una seconda proteina P53 che
viene fosforilata e stabilizzata. P53 è un fattore trascrizionale
che è stato denominato il guardiano del genoma.
•P53 quando è attivata da ATM attiva a sua volta altre
proteine (p21) che bloccano il ciclo cellulare fino alla
riparazione del danno.
Meccanismi di risposta cellulare al danno del DNA
•Se il danno del DNA non viene riparato P53 attiva altre
proteine che determinano apoptosi (morte cellulare
programmata).
•Difetti del gene TP53 sul cromosoma 17 che codifica per P53
causano una sindrome denominata Sindrome di Li-Fraunemi.
•Nelle famiglie con Sindrome di Li-Fraunemi si osservano
numerosi casi di differenti tumori ad esordio prima dei 50
anni di vita
Mutazioni che non vengono
riconosciute come tali e quindi non
vengono corrette
MALATTIE GENETICHE
MALATTIE MOLECOLARI
MALATTIE METABOLICHE
Una malattia genetica è una patologia la cui causa è insita nel genoma dell'individuo; può essere dovuta
alla presenza di uno o più alleli che producono polipeptidi con struttura e funzionalità anomala o alla mal
regolazione nell'espressione di geni "normali".
Una malattia molecolare è causata da un'anomalia o da una carenza di una particolare molecola
Una malattia metabolica è un’alterazione più o meno grave delle reazioni chimiche endocellulari e dei loro
sistemi di controllo
mutazione
protoncogeni
Proteine che controllano la
replicazione e il differenziamento
cellulare
oncogeni
cancro
Perdita del controllo dell’attività
proliferativa
Gli oncogeni e i geni oncosoppressori sono geni che in condizioni normali sono coinvolti nella regolazione della
crescita delle cellule: gli oncogeni stimolano la proliferazione cellulare, mentre gli oncosoppressori la inibiscono.
VIRUS ONCOGENI:
Presenza nel loro genoma di un oncogene derivato dal trasferimento di una parte di
un protooncogene proveniente dal genoma di una cellula eucariotica parassitata.
Dopo l’infezione, la proteina codificata dall’oncogene del genoma virale, inizia a
svolgere la sua funzione, portando ad un aumento della riproduzione cellulare, che
non viene più controllata


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)








