Nuovi approcci all’identificazione e
caratterizzazione di geni regolati
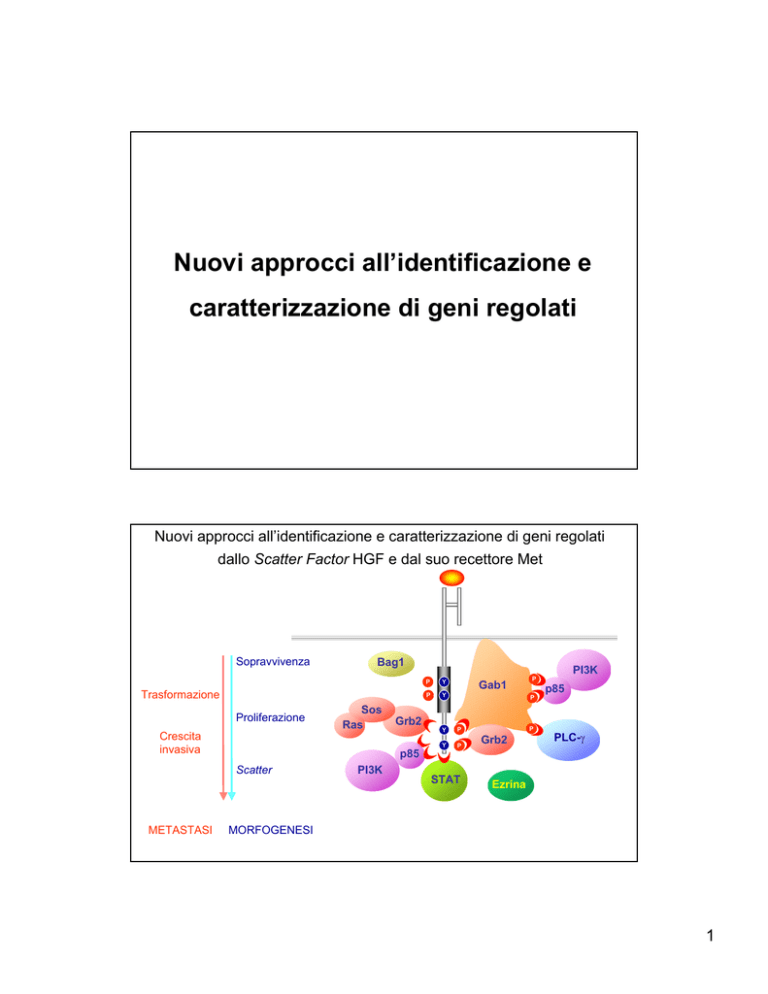
Nuovi approcci all’identificazione e caratterizzazione di geni regolati
dallo Scatter Factor HGF e dal suo recettore Met
HGF
Met
Sopravvivenza
Bag1
Trasformazione
Proliferazione
Sos
Ras
Crescita
invasiva
Y
P
Y
Gab1
Y
p85
P
Grb2
Y
P
P
P
P
Grb2
PLC-γ
p85
Scatter
METASTASI
PI3K
P
PI3K
STAT
Ezrina
MORFOGENESI
1
La risposta trascrizionale all’HGF
mRNA presenti
allo stato basale
mRNA basali
mRNA presenti dopo
stimolazione con HGF
Stimolazione con HGF
mRNA indotti
mRNA repressi
mRNA basali
mRNA basali
stimolazione
mRNA repressi
mRNA basali
mRNA indotti
1. DIFFERENTIAL DISPLAY
2. SUBTRACTED LIBRARIES
3. PROFILO DI ESPRESSIONE GENICA SU MICROARRAY
4. TECNICA DEL “GENE TRAP”
2
1.DIFFERENTIAL DISPLAY
2. SUBTRACTED LIBRARIES
3
4
3. PROFILO DI ESPRESSIONE GENICA
su MICROARRAY o MACROARRAY
Campione di controllo
SOPPRESSI INDOTTI
Estrazione dell’RNA,
sintesi del cDNA
e marcatura.
INVARIATI
10000 cDNA
Campione stimolato
MICROARRAY
Image from Gene-Chips (Microarray)
5
100000
stimulated
10000
2x induced
1000
2x suppressed
100
10
10
100
1000
10000
100000
Unstimulated
MACROARRAY
6
7
MACROARRAY SU NYLON
MICROARRAY SU SLIDES
Dimensioni campioni
5 µg RNA totale
0.5 µg mRNA
50-100 µg RNA totale
10 µg mRNA
Sistemi di rivelazione
33P
Fluorescenza (Cy3, Cy5)
Analisi di immagine
phosphorimager
Laser confocale
Ibridizzazione
Su due filtri diversi
Sullo stesso vetrino
Volume ibridizzazione
5-40 ml (come un Northern)
2-10 µl
Geni analizzati
500-9000
Fino a 15.000
Costi
contenuti
Elevati (attrezzatura)
Analisi dati
complessa
Molto complessa
, 32P
8
Major HGF-regulated genes and total
cDNAs corresponding to extracellular
matrix proteins contained on the
microarray
Genes regulated by HGF >3-fold are
listed in order of fold induction and
italicized. In addition, all of the cDNAs
corresponding to extracellular matrix
proteins, extracted from a total of 8735
spotted cDNAs, are listed in alphabetical
order. Average induction and SD were
obtained from a triplicate comparison
between unstimulated and HGFstimulated (6h) cells.
To enrich for target genes associated
with HGF-induced scattering, two
experiments were performed in 0.1%
serum and one in 2% serum.
MLP-29 cells respond to HGF and EGF by increasing OPN mRNA and protein
Western blot analysis of OPN protein expression by MLP-29 cells after different times
of HGF or EGF stimulation, as indicated. Total cell lysate (20 mg) was loaded per lane,
followed by fractionation in 8% SDS-PAGE and immunoblotting.
9
4. TECNICA DEL “GENE TRAP”
GFNR
(Green Fluorescent Nitro-Reductase)
GFNR
EGFP
NTR
• quando una cellula esprime la proteina di fusione GFNR
diventa al tempo stesso fluorescente e sensibile al
Metronidazolo (MN)
Vettore di espressione per GFNR
10
Validazione della proteina di fusione: fluorescenza e sensibilità al MN
Fluorescence and drug sensitivity of 293T cells transiently transfected with pEGFP, pGFNR, and
pNTR. Relative pGFNR efficiency was estimated either by flow cytometric analysis (left) or by
cell counting after 48 h of MN treatment (right). A fluorescence efficiency of 100% corresponds to
the mean fluorescence intensity of the green fluorescent subpopulation in the pEGFPtransfected cells. A killing efficiency of 100% corresponds to the difference in cell number
between mock-transfected and pNTR-transfected MN-treated cells. Error bars represent
standard deviation of triplicates.
Validazione della proteina di fusione:
correlazione fra fluorescenza e sensibilità al MN
Correlation between fluorescence and MN sensitivity in stable transfectants. MLP-29 cells were
transfected with pGFNR, selected with G418, and FACS-analyzed for green fluorescence before
and after 48 h of MN treatment. For the analysis, three classes of fluorescence were identified
and defined as low, medium, and high. In the untreated population, the number of cells in each
class was assigned the 100% value. After treatment, the abundance of each fluorescent
subpopulation was compared with the respective control sample, and percentage of survival was
estimated. Error bars are not present because data were obtained by a FACS-based population
analysis.
11
Vettore di espressione per GFNR e
trappola genica
Meccanismo di intrappolamento genico
Promotore
Gene
endogeno
Esone1
Esoni 2,3...
Introne1
Coda di poliA
mRNA
TRAPPOLA GENICA
Promotore
Gene
intrappolato
Esone1
SA
GFNR
EGFP
Esone1SA
Cassetta Neo
NTR
GFNR
PGK
prom
neo pA
Cassetta Neo
Coda di poliA
mRNA ibrido
EGFP
NTR
PGK neo pA
prom
12
Un reporter per i geni regolati: GFNR
(Green Fluorescent Nitro-Reductase)
GFNR
EGFP
NTR
• quando una cellula esprime la proteina di fusione GFNR diventa
al tempo stesso fluorescente e sensibile al Metronidazolo (MN)
• le cellule in cui la trappola genica è integrata a valle di un
promotore attivo possono essere identificate e selezionate
positivamente al FACS
• le integrazioni a valle di promotori costitutivi possono essere
controselezionate mediante trattamento con MN
• le integrazioni a valle di promotori regolati dall’HGF possono
essere selezionate stimolando le cellule con HGF prima della
selezione positiva (geni indotti) o negativa (geni soppressi)
Ottimizzazione delle procedure di selezione positiva
infezione
selezione contemporanea con G418
e con MN
sopravvivono tutti i cloni che hanno integrato il
DNA nel loro genoma (G418 è un analogo della
neomicina)
muoiono i cloni che hanno integrato la gene trap
in un gene trascritto attivamente (geni
housekeeping)
rimozione del MN
stimolazione con HGF
e isolamento (“sorting”) delle cellule
fluorescenti
le cellule fluorescenti sono quelle che hanno
integrato la gene trap in un gene indotto
dall’HGF
TRAPPOLA GENICA
screening dei cloni sopravvissuti per
SA
GFNR
Cassetta Neo
identificare quelli indotti dall’HGF
EGFP
NTR
PGK
prom
neo pA
13
Ottimizzazione delle procedure di selezione negativa
infezione
selezione con G418
sopravvivono tutti i cloni che hanno integrato il
DNA nel loro genoma (G418 è un analogo della
neomicina)
isolamento (“sorting”) delle cellule fluorescenti
stimolazione con HGF
e controselezione con MN
le cellule fluorescenti sono quelle che hanno
integrato la gene trap in un gene attivamente
trascritto (geni housekeeping)
muoiono i cloni che hanno integrato la gene
trap in un gene housekeeping o in un gene
indotto dall’HGF
screening dei cloni sopravvissuti per
sopravvivono i cloni che hanno integrato la
gene trap in un gene soppresso dall’HGF
identificare quelli soppressi dall’HGF
TRAPPOLA GENICA
SA
GFNR
EGFP
NTR
Cassetta Neo
PGK
prom
neo pA
Analisi al citofluorimetro
di popolazioni di cellule “trappate”
HGF induced traps
HGF induced traps
First round
Second round
HGF suppressed traps
14
ANALISI CITOFLUORIMETRICA DEI CLONI
Clone E3.2
Clone HF-57
Controllo
HGF
Abbondanza relativa (%)
Abbondanza relativa (%)
Responsività all’HGF dei cloni intrappolati
60
50
40
30
20
10
70
60
50
40
30
20
10
0
0
1.5 - 2
2-3
>3
Livelli di induzione
1.5 - 2
2-3
>3
Livelli di soppressione
15
RACE = RAPID AMPLIFICATION OF cDNA ENDS
TRAPPOLA GENICA
Promotore
Esone1
Gene
intrappolato
SA
GFNR
EGFP
Esone1 SA
Cassetta Neo
NTR
PGK
prom
GFNR
neo pA
Cassetta Neo
Coda di poliA
mRNA ibrido
EGFP
retrotrascrizione con primer
NTR
PGK neo pA
prom
su GFNR
cDNA
add oligo poly G
PCR
primer poly C
prodotto di PCR
Identificazione dei geni intrappolati
Sequenza del prodotto di 5’-RACE del clone 3E1.8
CCCNCNTCNC
CCTGGGTGGT
CAGTAGCGAT
ATGGNGGCGA
GGCNTTGTTA CNACNGCTTG CCNAAAGTAT CCAGTGAGAG AGGCAGATGC
AGAGCAGGCT GCTGGTCTGG TCCTCTCTTC AGGAGATTTG CAACCCTGGA
ACCGTCGATC CCCACTGGAA AGACGCGAAG AGTTTGTCCT CAACCGCGAG
CGGTAGCGCT
primer della RACE
gene Tmp
SA
EGFP
Su 10 cloni sequenziati:
2 corrispondono a geni noti: Tmp e Sprr2h
3 corrispondono a EST: AA274109, AI931556 e C89509
4 corrispondono a sequenze ripetitive
1 non corrisponde ad alcuna sequenza nota
16
Specificità delle traps
HGF
FBS
HGF+FBS
2.2
1.9
1.3
1.0
0.2
0.4
0.6
1.00
1.5
1.2
1.7
3.0
2.6
1.8
2.2
1.7
2.3
1.2
1.6
1.8
2.3
4.6
3.8
4.0
2.2
1.7
2.6
Fold Induction
3.7
10.00
0.10
HF-39
E3.2
(Novel)
EST
Tmp E4H-20-6 Sprr2H H5-16
H5-17 Unknown
Validazione della trappola
Abbondanza
relativa (%)
Abbondanza
relativa (%)
100
600
400
200
0
80
60
40
20
0
Sprr2h
EST
AI931556
GAPDH
GAPDH
Tempo
(ore)
0
1.5
3
6
16
Tempo
(ore)
0
1.5
3
6
16
Northern blot (RNA di cellule MLP-29 stimolate con HGF a tempi diversi)
17
Conclusioni e Prospettive
• è stato sviluppato un nuovo approccio di intrappolamento genico
che consente di identificare in modo veloce ed efficiente i geni la cui
trascrizione è regolata da stimoli esogeni (in questo caso l’HGF)
• l’ottimizzazione delle procedure permette lo screening funzionale
dell’intero genoma; sono stati così isolati numerosi cloni responsivi
all’HGF
• i cloni responsivi costituiscono un insieme di cellule reporter nelle
quali si possono valutare, mediante analisi citofluorimetrica, le
risposte trascrizionali dei geni intrappolati, usando anche stimoli
diversi dall’HGF per una caratterizzazione funzionale più dettagliata
18












