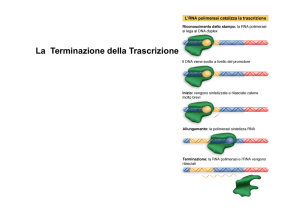
TERMINAZIONE DELLA TRASCRIZIONE NEGLI EUCARIOTI
La coda dell’rna messaggero è una coda di poliA, legata ai processi di terminazione della sintesi
di rna messaggero negli eucarioti: quanto più è lunga, piu è stabile l’mrna, perciò la lunghezza è
un modo per regolare l’espressione genica.
Per formare la coda di PoliA serve l’intervento di un complesso di proteine elaborato:
innanzitutto deve intervenire CPSF (fattore di stimolo di taglio e di poliadenilazione) che
riconosce una specifica sequenza di terminazione (di solito AAUAAA) che è il segnale di inizio
della terminazione della sintesi di rna.
Questo complesso CPSF lega altre proteine, tra cui CSTF, importante per reclutare i fattori di
taglio 1 e 2, che son endonucleasi che tagliano in un punto casuale l’rna: queste endonucleasi
possono digerire il messaggero, perche lo stato di fosforilazione della coda di poliA impedisce il
reclutamento delle proteine che formano il cappuccio (quindi l’estremita 5’ che viene fuori è
priva del cappuccio).
CPSF recluta anche la PoliA polimerasi, che sintetizza la coda di PoliA (senza stampo) dopo che è
stato effettuato il taglio. La poliA polimerasi è associata inoltre anche con PAB 2 (Poli A binding
protein), che dà processività alla poliA polimerasi (poiché di suo è poco processiva e
sintetizzerebbe una coda di poliA corta, mentre grazie alla PAB 2 diventa di 250 A°).
La poliA polimerasi ha molte caratteristiche che la rendono simile ad altri enzimi che
sintetizzano acidi nucleici, infatti possiede la struttura a mano che si chiude sul substrato, e
anche la chimica è simile alle altre polimerasi. Ha residui metallici nel sito attivo, uno di questi
è portato dal substrato entrante (in questo caso ATP) e all’interno del sito attivo c’è un residuo
di aspartato che coordina un altro ione metallico, che partecipa alla catalisi.
La poliA polimerasi ha una struttura che ricorda una mano che si chiude sul substrato; ci sono 3
domini:
•
Cerniera (è centrale)
•
Catalitico
•
Di legame con l’rna (ricorda gli RRM)
La PABP invece lega la coda di poliA non è appena viene sintetizzata attraverso due Rna
Reconditiong Motive (RRM) (beta-foglietti con le 2 alfa-eliche dall’altro lato). Inoltre la PABP ha
nella regione C-terminale un dominio composto da 4 alfa-eliche importanti per determinare le
interazioni proteina-proteina della PABP con altre proteine, come fattori di inizio della sintesi
proteica, soprattutto elF4G.
Quindi il nostro rna messaggero ha un cappuccio al 5’ (riconosciuto da elf4E) mentre la coda di
poli A è riconosciuta da PABP, che è in grado di legare elf4G che lega anche elf4E.
Perciò che cosa accade?
La circolarizzazione dell’rna messaggero è un punto di controllo di qualità cruciale perché l’rna
messaggero venga riconosciuto e il ribosoma decida di legarsi e leggere quell’m rna.
Alla PABP si lega anche PAIP, che interagisce sia con PABP sia con un altro fattore di inizio della
sintesti proteica (elf4A) promuovendo la circolarizzazione dell’m rna.
Quando una cellula non vuole più fare sintesi proteica, aumenta l’espressione di PAIP 2, che
inibisce l’azione di PAIP1 (inibendo cosi la circolarizzazione).
Il legame di PoliA polimerasi è importante per la formazione di una coda di PoliA: se poliA è
inibita, l’Mrna non è piu stabile, ma viene degradato rapidamente dall’esonucleasi.
La proteina U1A regola la sua stessa espressione genica, inibendo la PoliA polimerasi: essa è una
importante proteina che vedremo nel processo di splicing. Infatti esiste la SNARP U1 che tra le
proteine che possiede ha la U1A. Se la SNARP U1 non fa parte del complesso snarp ma è libera
nella cellula, è in eccesso rispetto alla small nuclear RNA (perciò non bisogna sintetizzarla).
Quando è libera, questa SNARP è in grado di interagire con il suo stesso m RNA, perché nel suo m
RNA c’è una regione che forma strutture a elica e ansa. Questa struttura particolare dell’m RNA
della proteina U1A può essere riconosciuta dalla proteina U1A stessa, quindi è reclutata vicino
all’m RNA, cosi può interagire con la poliA polimerasi, inibendola: cosi destabilizza il suo stesso
m RNA.
Il riconoscimento della sequenza terminale avviene perché, una volta che è riconosciuta la
sequenza di taglio, l’RNA che è stato tagliato è privo del cappuccio al 5’(perché lo stato di
fosforilazione della coda della polimerasi impedisce il reclutamento delle proteine che formano
il cap), quindi è aggredibile dalle RNAsi (RAT1 nel lievito, XRN2 nell’uomo). Queste nucleasi
scalzano l’RNA polimerasi una volta raggiunto e questo si stacca.






![Lezione 13 Codice genetico e sintesi proteica [modalità compatibilità]](http://s1.studylibit.com/store/data/000920742_1-3ab75c8c6018e2c240427969189fe9cd-300x300.png)