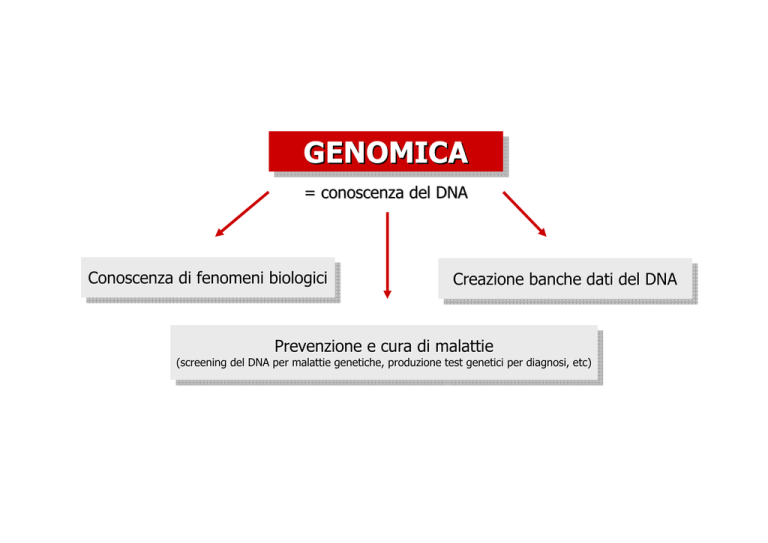
GENOMICA
GENOMICA
= conoscenza del DNA
Conoscenza
Conoscenzadidifenomeni
fenomenibiologici
biologici
Creazione
Creazionebanche
banchedati
datidel
delDNA
DNA
Prevenzione
Prevenzioneeecura
curadidimalattie
malattie
(screening del DNA per malattie genetiche,
, produzione test genetici per diagnosi,, etc)
(screening del DNA per malattiegenetiche
genetiche, produzione test genetici perdiagnosi
diagnosi, etc)
GENOMICA
GENOMICA
Analisi della sequenza di interi genomi
Genomica
Genomicacomparativa
comparativa
(somiglianze genetiche tra specie)
(somiglianze genetiche tra specie)
Progetto
ProgettoGenoma
GenomaUmano
Umano
Genomica
Genomicafunzionale
funzionale
(funzione dei geni)
)
(funzione deigeni
geni)
RIDURRE LA COMPLESSITA’ !
Librerie (o banche) genomiche
Librerie (o banche) cromosomiche
CGGCATTTGCGTATTCGGCC
TAGCTACTCGTACCCCTGAT
AGGTGTGCATTTAGGGCACT
GCAGGGCCTCGGCAATCCGC
CCCGGTGTGAATAAATCGTA
INGEGNERIA GENETICA
Insieme di tecnologie e strumenti molecolari per lo studio e
la manipolazione del DNA finalizzate:
1) a risolvere la sua complessità
2) al suo clonaggio
3) alla sua collezione in “biblioteche” molecolari
STRUMENTI DELL’INGEGNERIA GENETICA
ENZIMI DI RESTRIZIONE : consentono di tagliare il DNA in punti specifici, in
modo da separare il tratto di DNA desiderato, di legarlo al vettore prescelto, o
modificarlo in vitro, in modo conveniente alle proprie necessità.
VETTORI: consentono l’ingresso del gene scelto nella cellula ospite,
provvedono alla replicazione del DNA eterologo ed alla sua espressione.
ORGANISMI OSPITI: batteri, cellule animali e vegetali, lieviti. Sono dotati di
caratteristiche genetiche o biochimiche altamente specifiche che li rendono più o
meno adatti a determinate applicazioni pratiche o ad ospitare vettori.
Consentono di amplificare la sequenza di DNA clonata nel vettore.
Studiare il Genoma
Tagliare il DNA in punti specifici (enzimi di restrizione)
restrizione
Localizzare specifiche sequenze di DNA (Southern blotting e FISH)
FISH
Amplificare una sequenza di DNA generando quantità manipolabili
(PCR e applicazioni)
applicazioni
Mutare e/o unire frammenti di DNA per produrre sequenze desiderate
(mutagenesi e clonaggio genico)
genico
Trasferire nuove sequenze di DNA in cellule riceventi e selezione di
cloni positivi
Costruzione di “biblioteche” molecolari (genoteche o libraries)
TAGLIARE
TAGLIARE IL
IL DNA
DNA
1
2
3
Digestione vettore con lo
stesso enzima di restrizione
Sau3A
Ligazione
5’-GGATCC-3’
3’-CCTAGG-5’
5’-G
3’-CCTAG-5’
5’-GATCC-3’
G-5’
OSSERVARE
OSSERVARE II FRAMMENTI
FRAMMENTI DI
DI DNA
DNA
L’elettroforesi su gel permette di separare i frammenti di DNA in base
alle dimensioni
1-Il DNA, insieme ad
un colorante che
permette di seguirne
la “corsa”, si carica
sul gel di agarosio
3- Il DNA si può “osservare” grazie alla
presenza nel gel di bromuro di etidio, un
intercalante in grado di emettere fluorescenza
quando eccitato da raggi UV (emessi da un
trans-illuminatore)
2- L’apparato di elettroforesi si collega
ad un alimentatore e si applica una
corrente elettrica che permette la
migrazione del DNA verso il polo
positivo (elettrodo rosso in figura)
L’elettroforesi su gel consente
separare molecole di DNA
dimensioni diverse
di
di
Frammenti attesi dalla digestione con i
seguenti enzimi di restrizione:
Pst1
AatII
0
(2840bp)
Pst1
2840bp
AatII
900bp e 1940bp
XhoI
2840bp
XhoI
E da doppie digestioni?
AatII
Pst1+XhoI
700bp e 2140bp
AatII+XhoI
500bp, 400bp e 1940bp
Costruzione di una mappa di restrizione in un frammento di DNA
M
ctr
E
5
4,5
B
3
2
0,5
E/B
2,5
2
0,5
Digestione di molecole di DNA
relativamente piccole
Digestione di
DNA genomico
Come identificare la banda di interesse?
LOCALIZZARE
LOCALIZZARE II FRAMMENTI
FRAMMENTI DI
DI DNA
DNA
Una sonda è una sequenza di DNA o RNA (a singolo o doppio
filamento), complementare ad un DNA o RNA di interesse.
interesse
L’ibridazione tra la sequenza target e la sonda può essere rivelata
attraverso la radioattività (sonda marcata con isotopi radioattivi) o la
fluorescenza (sonda marcata con un fluoroforo).
+
Denaturazione
Ibridazione
Denaturazione (A) e rinaturazione (B) del DNA
(A)
La denaturazione a 95-100°C, seguita dal
rinaturazione delle molecole del DNA omologhe
(B)
raffreddamento,
permette
la
La rinaturazione tra molecole di DNA con totale omologia avviene a temperature
più alte (alta specificità) rispetto a quelle con omologia parziale (bassa specificità)
Una sonda di DNA (omologa o eterologa) può essere:
1- un cDNA parziale o full-length
2- un oligonucleotide
3- un amplificato da PCR
4- il DNA del gene
Una sonda di RNA
Identificazione di una sequenza per
ibridazione con una sonda
riconoscimento della sequenza d’interesse
SOUTHERN BLOTTING:
BLOTTING
come identificare un ago (gene)
in un pagliaio (genoma)
Southern blotting
(1970- Dr. Edward Southern)
Procedura
Procedura
––Isolamento
Isolamentodel
delDNA
DNAgenomico
genomico
––Digestione
Digestionecon
conenzimi
enzimididirestrizione
restrizione
specifici
specifici
––Separazione
Separazionemediante
medianteelettroforesi
elettroforesisu
sugel
gel
d’agarosio
dei
frammenti
di
DNA
d’agarosio dei frammenti di DNA
––Denaturazione
Denaturazionedel
delDNA
DNAper
perseparare
separarei i
filamenti
filamenticomplementari
complementari
––Trasferimento
Trasferimentodel
delDNA
DNAsu
sumembrana
membranaper
per
capillarità
capillarità
––Ibridazione
Ibridazionecon
conuna
unasonda
sondamarcata
marcata
–Autoradiografia
–Autoradiografia
Gel di agarosio colorato
con EtBr
Autoradiografia
Il Southern blotting permette di:
-determinare la presenza o assenza di specifici frammenti genici
- studiare la struttura e l’organizzazione di un gene
Applicazioni
Diagnosi di malattie genetiche ereditarie (es. anemia
falciforme, Corea di Huntington, etc) sia prenatale che
preclinica
Medicina forense (determinazione dell’”impronta” genetica)
Individuazione di agenti patogeni nelle infezioni
Analisi filogenetiche
Mappatura del genoma
A2A2
A1A2
A1A1
Una mutazione associata alla malattia (non necessariamente causale) può causare la perdita
o l’acquisizione di un sito di restrizione e quindi modificare il normale pannello di restrizione.
E’ il caso del gene della globina β mutata nell’anemia falciforme
(normale)
(mutata)
La mutazione (causa della
malattia) modifica un sito di
restrizione
sonda
sonda
Il Southern Blotting permette di
fare una diagnosi molecolare
della malattia
Il Southern blotting nello studio della variabilità genetica
RFLP (Restriction Fragment Lenght Polymorphism)
Quattro individui che presentano una diversa
lunghezza dei frammenti di restrizione nel
medesimo locus genico, rivelati per Southern
Blotting mediante la sonda indicata in figura.
Varianti del Southern Blotting:
Northern Blotting analisi di RNA
Western Blotting analisi di proteine
Eastern Blotting analisi di PTM
Northern Blotting
una variante del Southern Blotting per identificare un trascritto (RNA)
Procedura
Procedura
––Isolamento
Isolamentodell’RNA
dell’RNA
––Elettroforesi
Elettroforesidell’RNA
dell’RNAsu
sugel
geldidiagarosio
agarosio
––Trasferimento
dell’RNA
su
membrana
Trasferimento dell’RNA su membrana
––Ibridazione
Ibridazionecon
conuna
unasonda
sondamarcata
marcata
––Autoradiografia
Autoradiografia
Il Northern blotting permette di:
-determinare la dimensione e la quantità di specifici RNA
- studiare l’espressione genica
Applicazioni
Espressione di un trascritto in diversi tessuti
Come varia l’espressione di un trascritto
Caratterizzazione di diversi trascritti (es.isoforme di splicing)
Espressione di un trascritto in diversi tessuti
Es. Northern Blotting con mRNA estratti
da tessuti diversi.
Si ottengono diverse informazioni:
1- livello di espressione nei tessuti
esaminati
2- tipo di mRNA prodotto
Come varia l’espressione di un trascritto
trattamento
Controllo di caricamento
Gene
housekeeping
Gene
di interesse
Es. Northern Blotting in una stessa linea cellulare prima e dopo
trattamento con un farmaco
Caratterizzazione di isoforme di splicing
AMPLIFICARE
AMPLIFICARE IL
IL DNA
DNA
Clonaggio in vettori e amplificazione in cellule ospiti
(anche di sequenze non note)
CGGCATTTGCGTATTCGGCC
TAGCTACTCGTACCCCTGAT
AGGTGTGCATTTAGGGCACT
GCAGGGCCTCGGCAATCCGC
CCCGGTGTGAATAAATCGTA
CGGCATTTGCGTATTCGGCC
TAGCTACTCGTACCCCTGAT
AGGTGTGCATTTAGGGCACT
GCAGGGCCTCGGCAATCCGC
CCCGGTGTGAATAAATCGTA
CGGCATTTGCGTATTCGGCC
TAGCTACTCGTACCCCTGAT
AGGTGTGCATTTAGGGCACT
GCAGGGCCTCGGCAATCCGC
CCCGGTGTGAATAAATCGTA
CGGCATTTGCGTATTCGGCC
TAGCTACTCGTACCCCTGAT
AGGTGTGCATTTAGGGCACT
GCAGGGCCTCGGCAATCCGC
CCCGGTGTGAATAAATCGTA
CGGCATTTGCGTATTCGGCC
TAGCTACTCGTACCCCTGAT
AGGTGTGCATTTAGGGCACT
GCAGGGCCTCGGCAATCCGC
CCCGGTGTGAATAAATCGTA
CGGCATTTGCGTATTCGGCC
TAGCTACTCGTACCCCTGAT
AGGTGTGCATTTAGGGCACT
GCAGGGCCTCGGCAATCCGC
CCCGGTGTGAATAAATCGTA
Amplificazione di sequenze note mediante la reazione
a catena della DNA polimerasi o PCR
CGGCATTTGCGTATTCGGCC
TAGCTACTCGTACCCCTGAT
AGGTGTGCATTTAGGGCACT
GCAGGGCCTCGGCAATCCGC
CCCGGTGTGAATAAATCGTA
PCR (Polymerase Chain Reaction)
La reazione a catena della polimerasi
Mullis- Nobel 1993
È una metodica che consente di ottenere un numero
enorme di copie di una specifica sequenza di DNA
partendo da una piccola quantità di templato
Cicli
Ciclididi denaturazione
denaturazione
annealing
annealingdei
deiprimers
primers
allungamento
allungamento(polimerizzazione)
(polimerizzazione)
La Taq polimerasi (da Thermus aquaticus) è una DNA polimerasi resistente alle alte
temperature
Cicli
Ciclididi denaturazione
denaturazione
annealing
annealingdei
deiprimers
primers
allungamento
allungamento(polimerizzazione)
(polimerizzazione)
La Taq polimerasi (da Thermus aquaticus) è una DNA polimerasi resistente alle alte
temperature e per questo utilizzata nella PCR
http://www.youtube.com/watch?v=HMC7c2T8fVk
http://www.youtube.com/watch?v=eEcy9k_KsDI
Real-time PCR o PCR quantitativa
Il SYBR Green I emette fluorescenza solo dopo il
legame al DNA la quantità di fluorescenza è
proporzionale alla quantità totale di DNA a doppio
filamento presente.
La fluorescenza aumenta man mano che il target viene
amplificato
Vantaggi
Vantaggi della
della PCR:
PCR:
Amplificazione
Amplificazione di
di tracce
tracce minime
minime di
di DNA
DNA
Diagnosi
Diagnosi molecolare
molecolare diretta
diretta senza
senza l’uso
l’uso di
di sonde
sonde
radioattive
radioattive
Applicazioni della PCR:
Diagnosi di malattie genetiche ereditarie sia prenatale che preclinica
Amplificazione di DNA per clonaggio e sequenziamento
Medicina forense (determinazione dell’”impronta” genetica)
Individuazione di agenti patogeni nelle infezioni
DNA amplificato per PCR
DNA amplificato per PCR
Es. dell’anemia falciforme
La PCR sequita da digestione con
enzimi di restrizione (DdeI)
permette di fare una diagnosi
molecolare della malattia
RT= retrotrascrizione
PCR
PCR
Uso della PCR per rivelare la presenza di un genoma virale in un
campione di sangue
CLONARE
CLONARE IL
IL DNA
DNA
Fasi di un esperimento di clonaggio:
1) isolamento di un frammento di DNA (frammento di restrizione o
amplificato per PCR)
PCR
2) unione del filamento ad un vettore di clonaggio (tipi di vettori)
vettori
3) introduzione del DNA ricombinante nelle cellule ospiti (trasformazione
batterica)
batterica
4) identificazione delle cellule contenenti le molecole di DNA ricombinante
di interesse (selezione dei batteri trasformati)
trasformati
5) isolamento del DNA di interesse ed eventuale sequenziamento
Vettori di clonaggio
Permettono il trasferimento di DNA nella cellula ospite in
modo da farlo replicare e trasmetterlo alle cellule figlie.
Alcuni rimangono in forma episomale, altri si integrano nel
genoma dell’ospite
Elementi genetici extracromosomici
(cosmidi o plasmidi)
Tipi di vettore
Vettori virali (fagi o virus animali)
Caratteristiche di un vettore ideale
Ogni tipo di vettore possiede caratteristiche che lo rendono
adatto al tipo di applicazione di interesse
Plasmidi di piccole dimensioni, facili da isolare e purificare, con capacità di
replicazione autonoma, conferita anche da un’origine di replicazione.
Es. pBR322.
pBR322
Fagi ospitano frammenti di DNA più lunghi rispetto ai plasmidi.
es. fago lambda con genoma completamente sequenziato e fornito di
appropriate mutazioni, in modo da adattarlo alle necessità del clonaggio
(vengono eliminati alcuni geni del fago che sono sostituiti dal DNA esogeno).
Vettori di clonaggio
Vettore
dimensione dell’inserto (kb)
Plasmidi
0,1-15
Esempi
pUC18, pBR322
Fagi
8-22
λ
Cosmidi
32-45
pJB8
BAC
100-300
pBAC108L
YAC
1000-2000*
pYAC4
*2000kb= 2Mbp
NB: Il cromosoma più
più piccolo, il 21, ha circa 46 Mbp
Clonaggio di DNA in un vettore plasmidico
Sito di
taglio
Amp+
Trasformazione
Trasformazione
batterica
batterica
La trasformazione e
l’uso di marcatori
selezionabili
I batteri
I batteri
trasformati sono
trasformati sono
piastrati su
piastrati su
terreno
terrenoselettivo
selettivo
(+ampicillina)
)
+ampicillina
(+ampicillina)
Colonie
Colonie
resistenti
resistenti
all’
’ampicillina
all
all’ampicillina
NB
Uso di lacZ come
marcatore di clonaggio
Vettori derivati
da batteriofagi
Es. il fago λ
Selezione e amplificazione: infezione batterica
placche fagiche
Ciclo litico
Vettori cosmidici
Caratteristiche:
ospitano fino a circa 45 kb di DNA
origine replicazione plasmidica
uno o più siti di restrizione unici
uno o più marcatori selezionabili
uno o più siti cos*
*cos = estremità coesive del batteriofago; permettono l'impacchettamento del DNA nel
virione.
Cosmidi
Un cosmide è un plasmide, di
circa 5 Kpb, contenente un sito cos
di λ .
Come tutti i
vettori plasmidici
contiene:
una ori
un marcatore di resistenza
un sito unico di restrizione per
l’inserimento del DNA
Il cosmide ricombinante, per essere
un buon substrato per la reazione di
packaging deve avere dimensione tra
37-51 Kb.
Ciclo lisogenico
Si clona come un plasmide e si
propaga come un fago λ
Considerando la dimensione media di un vettore cosmidico, la dimensione di un inserto che può
essere clonato in un cosmide varia tra 32 e 45Kb, che rappresenta più di quanto è possibile
clonare nei vettori lambda (8-22Kb)
Cromosomi artificiali
Un’estensione logica nella produzione di vettori per il clonaggio di grossi frammenti
di DNA è quella di ricostruire un cromosoma a replicazione autonoma nel quale
possono essere inseriti frammenti di DNA esogeno.
I cromosomi naturali eucariotici per la loro stabilità e funzionalità hanno le seguenti
strutture:
- Telomeri (DNA e proteine all’estremità dei cromosomi, che hanno una funzione
protettiva)
- Centromeri (segmenti di DNA altamente ripetuti che sono essenziali per il controllo
e la segregazione cromosomica)
Cromosomi artificiali batterici (BAC):
100-300 kb di DNA
Cromosomi artificiali di lievito (YAC):
ospitano fino a 600-2000 kb di DNA
origine di replicazione di lievito
2 telomeri (stabilità)
1 centromero
Cromosomi artificiali batterici: BAC
I vettori BAC contengono:
- siti cos del fago λ
- marcatori di selezione (CAMR=
resistenza al cloramfenicolo)
- oriS e repE per la replicazione
del fattore F
- polylinker per il clonaggio di
inserti
I Cromosomi Artificiali Batterici (BAC)
sono vettori che derivano dal fattore
sessuale F
Cromosomi artificiali di lievito:YAC
I vettori YAC contengono:
- un centromero (CEN4)
- una sequenza a replicazione
autonoma (ARS1) che permette
la replicazione autonoma rispetto
alle origini di replicazione
cromosomiche
- telomeri (TEL) necessari per la
replicazione e il mantenimento
dei cromosomi
- marcatori di selezione in lievito
(ura3 e trp1)
- origine di replicazione (oriC) e
un marcatore selettivo batterico,
batterico
per la propagazione in E. coli
prima del clonaggio.
Trasformazione di cellule di lievito
e selezione in terreno minimo (ura- e trp-)
Vettori di espressione
Il gene esogeno può essere trascritto e tradotto.
Promotore
la trascrizione del
Es. vettore di espressione
A cosa serve clonare il
DNA?
Con il clonaggio del DNA
si possono isolare i geni
Genoteca genomica


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)



![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)





