Gene : Unità ereditabile
• I segmenti di DNA che
trasportano
l’informazione
genetica sono chiamati geni.
• Corrispondono normalmente
ad un tratto di DNA che
codifica per una proteina o
per una catena di RNA con
una funzione nell’organismo.
• Geni portano l’informazione
per la costruzione ed il
mantenimento delle cellule in
un organismo e la trasmettono
alla
progenie
(tratto
genetico).
Il gene vegetale e la regolazione dell’espressione genica
Promotore
Insieme di elementi in cis rispetto alla regione codificante che
vengono riconosciuti da fattori cellulari endogeni.
La corretta interazione tra i fattori e gli elementi del DNA
consentono:
-
la sintesi dell’mRNA
la corretta maturazione
il trasporto nel citosol
la traduzione e la sintesi della proteina codificata
Il promotore occupa da poche a numerose centinaia di bp a monte del
sito di inizio della trascrizione e consiste in regioni di DNA non trascritte
contenenti numerosi elementi di regolazione: elementi minimi del
promotore ed enhancer (regioni di regolazione a monte).
Elementi minimi: sequenze di DNA prossime al sito
di inizio della trascrizione.
La sequenza di 7 basi (TATAAAA) chiamata TATA box si trova a circa
-10/-30 rispetto all’inizio della trascrizione. Vi si lega il Fattore di
Trascrizione IID (TFIID) che è un complesso di numerose proteine
tra cui la TATA-binding protein (TBP), che lega la TATA e richiama i
vari elementi dell’RNA pol II. E’ presente in quasi tutti i geni
codificanti per proteine.
La CCAAT box è una sequenza conservata (GGNCAATCT) posta a
circa -30/-70 rispetto al sito di inizio della trascrizione. Vi si legano
altri fattori di trascrizione.
E’ importante la posizione, l’ordine e la distanza reciproca.
Garantiscono livelli minimi di trascrizione.
Si parla di enhancer quando le sequenze regolative svolgono la loro
funzione indipendentemente dalla posizione e dalla distanza dal sito di
inizio della trascrizione.
Gli enhancer sono sequenze nucleotidiche poste in cis che esplicano la
loro funzione aumentando notevolmente (fino a 200 volte) la frequenza
di trascrizione del gene che controllano.
Non esiste un’unica sequenza conservata, ma sono noti centinaia di
enhancer diversi (differenze qualitative e quantitative)
Agli enhancer si legano fattori di
attivazione della trascrizione che
velocizzano la sintesi dell’RNA.
Esistono anche repressori ai quali
si legano fattori di inibizione della
trascrizione.
Non è molto importante il loro
orientamento
né
la
loro
posizione rispetto al sito di
inizio della trascrizione.
Gli enhancer definiscono il tipo di promotore creando differenze:
-Quantitative maggiore o minore forza, più o meno espresso
-Qualitative dove viene espresso
Ne segue che esistono promotori forti o deboli, oppure promotori
costitutivi o inducibili.
Promotori costitutivi: determinano l’espressione di un gene in tutti i
tessuti, indipendentemente dallo sviluppo e da fattori ambientali
esterni.
Il promotore costitutivo più famoso e usato è il promotore 35S derivato dal
Cauliflower mosaic virus CaMV.
Lungo circa 350bp, è ricco in enhancer ed è divisibile in due domini:
dominio A: espressione nelle radici,
nel periciclo e nel meristema apicale
dominio B: porzioni aeree
(foglie, fusti, fasci vascolari).
E’ divisibile in molti subdomini.
Espressione complementare
Dominio B: insieme di più elementi
(subdomini B1-5).
Alcuni
subdomini
di
B
presi
singolarmente garantiscono l’espressione in un determinato tessuto della
pianta, ma la somma dei profili di
espressione dei singoli domini non
equivale al profilo di espressione del
dominio B completo effetto
combinatorio
Interazione sinergica tra elementi di B
e tra B ed A.
I due domini hanno un effetto sinergico
e sommatorio. Principalmente attivo
nelle dicotiledoni.
Altri promotori costitutivi sono sempre di origine virale o batterica
(nos, ocs) o ancora vegetali come il promotore del gene per
l’ubiquitina, ma sono fino a 50 volte meno forti.
Nelle monocotiledoni il promotore 35S è solo moderatamente forte, si
usano promotori specifici delle monocotiledoni come quelli del gene
dell’actina di riso, ubiquitina di mais spesso accoppiati al primo
introne dello stesso gene che funziona da enhancer.
Promotori inducibili: determinano l’espressione di un gene solo in
particolari tessuti o organi, in particolari momenti dello sviluppo, a
seguito di particolari stimoli interni od esterni.
Contengono elementi in cis riconosciuti da particolari fattori
trascrizionali presenti solo in determinati tessuti o in particolari
momenti.
Non sempre il livello di espressione è altissimo, ma esso può essere
molto specifico.
Promotori tessuto specifici:
seme-specifici (proteine di riserva)
tubero specifici (patatina)
tapetum dell’antera
Fattori ambientali:
luce, ferita, patogeni, caldo , freddo, secco, anaerobiosi.
Terminatori e siti di poliadenilazione
Richiesti per una corretta ed efficiente trascrizione consentono la
formazione di un’estremità 3’ corretta che influenza la stabilità del mRNA.
Consenso: AATAAA 10-30 nt a monte più un tratto ricco in GT nelle
vicinanze.
Si utilizzano di solito terminatori ocs e nos o dal CaMV di 200-500bp.
Introni
Regioni non tradotte, intersperse nell’mRNA.
Se inseriti al 5’ del gene si verifica un notevole aumento nel livello di
espressione, inteso come steady state, quindi provocano un aumento
nella stabilità del messaggero. Soprattutto nelle monocot.
Esempi: 1° introne del gene Adh di mais, o bronze1 o shrunken1.
Aumento fino a 100 volte rispetto al semplice promotore 35S.
Devono essere inseriti nel corretto orientamento nella regione 5’ del
cDNA.
Introni vegetali mancano del branch
site ma sono caratterizzati:
dicot: siti di splicing (AG/GUAAG e
UGCAG/G) e alta percentuale (60%) di
A/U
monocot: siti di splicing ma possono
anche avere basse % di A/U
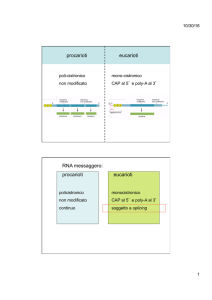
Regioni non tradotte
Le 5’-UTL influenzano grandemente l’efficienza di traduzione del
trascritto regolando l’attacco e lo scorrimento della subunità 40S del
ribosoma.
Se UTL ricca in strutture secondarie o in ATG spuri
bassa efficienza
Importante sequenza che circonda l’ATG
Consenso animali: GCC(A/G)CCAUGG, importante –3 serve purina
Vegetali: UAAACAAUGGCU, importante +4, ma meno stringente.
Codon usage
Codon usage:frequenza d’utilizzo di codoni sinonimi in un gene o un
genoma. L’importanza di questo parametro è dovuta all’osservazione che
il codon usage è diverso in organismi diversi.
Esprimendo geni di varie origini in cellule vegetali si possono avere
problemi di precoce terminazione della traduzione dovuti al diverso
codon usage e allo stallo del ribosoma in assenza del corretto tRNA.