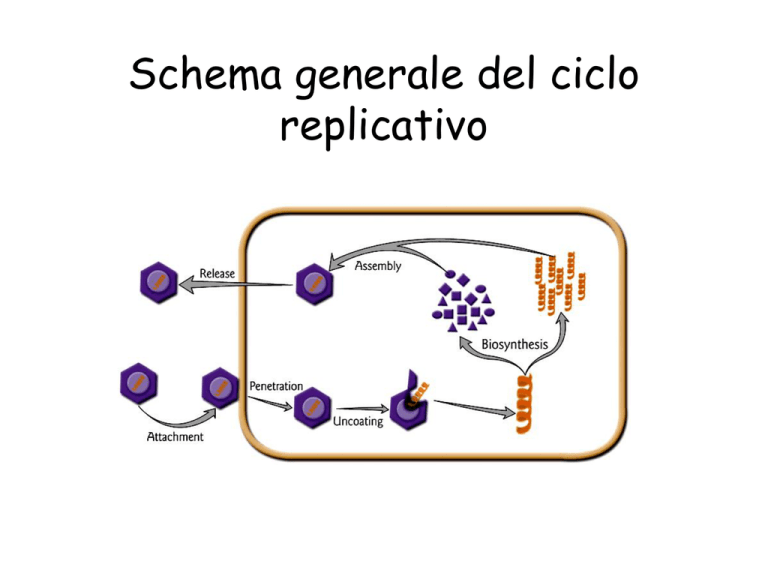
Schema generale del ciclo
replicativo
Classificazione secondo Baltimore
(suddivisione in classi di replicazione)
Tutti i virus devono produrre un mRNA che possa
essere tradotto dal macchinario traduzionale della
cellula. In questo sistema di classificazione, la
particolare via che dal genoma virale porta alla
produzione di mRNA definisce una specifica classe
virale basata sulla natura e sulla polarità dell’acido
nucleico.
Classificazione secondo Baltimore
(suddivisione in classi di replicazione)
Attenzione errore,
leggi +RNA
Figura 16.8 SCHEMA DEL FLUSSO DI EVENTI NELLA REPLICAZIONE DEI VIRUS ANIMALI A DNA DELLE CLASSI I, II E VII
SECONDO LA CLASSIFICAZIONE DI BALTIMORE.
a cura di Gianni Dehò ed Enrica Galli
Biologia dei microrganismi - II edizione
978-88-08-18686-7
2014 © CEA - Casa Editrice Ambrosiana
Classe VII: DNA doppio filamento con
intermedio a RNA
From Flint et al. Principles of Virology (2000), ASM Press
Intermedio di replicazione
+RNA
Genoma
Figura 16.9 SCHEMA DEL FLUSSO DEGLI EVENTI DELLA REPLICAZIONE DEI VIRUS ANIMALI A RNA DELLE CLASSI III, IV, V E
VI.
a cura di Gianni Dehò ed Enrica Galli
Biologia dei microrganismi - II edizione
978-88-08-18686-7
2014 © CEA - Casa Editrice Ambrosiana
Struttura e complessità dei
genomi virali
• Alcuni genomi di virus a DNA sono complessati ad
istoni cellulari a formare una struttura simile alla
cromatina all’interno dei virioni. Una volta all'interno
del nucleo della cellula ospite si comportano come
cromosomi satellite in miniatura seguendo i dettami
degli enzimi cellulari e del ciclo cellulare
• I primi mRNA poliadenilati al 3'osservati sono stati
quelli di Vaccinia virus
• Geni composti da introni non codificanti e esoni
codificanti, così come spliced mRNA, sono stati
scoperti per la prima volta in Adenovirus da Roberts
& Sharp in 1977.
Analisi dei genomi virali (DNA)
• Il genoma dei virus a DNA può essere
analizzato direttamente mediante
digestione con enzimi di restrizione
senza ricorrere al clonaggio molecolare
e questo approccio è stato applicato per
la prima volta per studiare il genoma a
DNA di SV40 nel 1971
Analisi dei genomi virali (RNA)
• L’analisi di sequenza di questi genomi non è
stata possibile fino a quando non è stato
identificato l’enzima trascrittasi inversa
(isolata dal virus della mieloblastosi aviaria,
1970), che permette di convertire RNA in
cDNA
Genomi a DNA, Herpesviridae
• Doppio filamento di DNA, lineare, le sue dimensioni
vanno da 120 kbp 220 kbp. Poiché tutti i virus
appartenenti a questa famiglia condividono le
funzioni base per una infezione produttiva, la
diversità nella grandezza dei genomi è dovuta alla
presenza di geni “dispensabili” coinvolti in aspetti
specifici della patogenesi di ciascun tipo di virus.
• Contiene tra i 60 e i 120 geni, a questo
corrispondono virioni complessi contenenti circa 35
proteine
Herpesviridae: virione
Genomi a DNA, Herpesviridae
Tre sottofamiglie
Alphaherpesvirinae:
Simplexvirus human herpesvirus 1 e 2 (HSV1, HSV2)
Varicellovirus human herpesvirus 3 (VZV)
Betaherpesvirinae:
Cytomegalovirus human herpesvirus 5 (CMV)
Muromegalovirus mouse cytomegalovirus 1
Roseolovirus human herpesvirus 6, 7 (HHV6, HHV7)
Gammaherpesvirinae:
Lymphocryptovirus human herpesvirus 4 (EBV)
Rhadinovirus human herpesvirus 8 ( HHV8 o KSHV)
Genomi a DNA, herpesviridae
HSV 1
I vari membri sono piuttosto diversi in termini di
sequenza genomica, ma condividono una struttura e
organizzazione comune: il genoma contiene due regioni
uniche (UL e US) fiancheggiate da ripetizioni invertite (TR
e IR), che determinano riarrangiamenti strutturali, tali
che questi genomi esistono come una mistura di 4 isomeri
funzionalmente equivalenti
From Flint et al. Principles of Virology (2000), ASM Press
Genomi a DNA, herpesviridae
•Codifica una varietà di enzimi coinvolti nel metabolismo degli
acidi nucleici, nella sintesi del DNA e nel processamento delle
proteine (protein kinasi)
HSV 1 - circa 84 ORF. E’ indicata la localizzazione approssimativa
dei geni e delle unità di trascrizione associate alla latenza. Geni
codificanti funzioni simili sono evidenziati con lo stesso colore. Geni
dispersi piuttosto che raggruppati
From Flint et al. Principles of Virology (2000), ASM Press
Genomi a DNA, herpesviridae
Origini di replicazione
HSV-1
OriL
EBV
OriS
OriS
Adenoviridae: virione
Genomi a DNA, Adenoviridae
• Doppio filamento lineare di 30-38 kbp, contenente 30-40
geni
• La sequenza terminale di ciascun filamento è una ripetizione
invertita di 100-140 bp (ITR), importante durante la
replicazione. Al 5’ di ogni filamento è associata una proteina
di 55 kDa nota come proteina terminale (TP)
• L’espressione genica è più complessa di quella degli
herpesvirus: i geni organizzati in cluster sono espressi a
partire da un ridotto numero di promotori; da ogni promotore
sono espresse più proteine (spliced mRNA e splicing
alternativo)
Genomi a DNA, Adenoviridae
From Cann Principles of molecular virology (2001). Academic Press
Genomi a DNA, Papillomaviridae
Genomi a DNA, Polyomaviridae
• Doppio filamento circolare di circa 5kbp
• Nel virione assume una struttura
superavvolta ed è associato a 4 istoni
cellulari H2A, H2B, H3 e H4
• Contiene 6 geni: essi sono posizionati in
entrambi i filamenti e spesso sono
sovrapposti
• L’origine di replicazione è circondata da
sequenze non codificanti che controllano la
trascrizione
Genomi a DNA, Polyomaviridae
From Cann Principles of molecular virology (2001). Academic Press
Genomi a DNA, Parvoviridae
• Lineare, singolo filamento circa 5kb. Molto spesso il filamento
impacchettato è quello –
• Anche i parvovirus replicazione-competente contengono solo due
geni: rep, proteine coinvolte nella replicazione, cap, proteine del
capside
• Le terminazioni genomiche hanno sequenze palindromiche di circa
115 nt, che formano strutture a forcina (hairpin) essenziali per
l’inizio della replicazione del genoma
From Cann Principles of molecular virology (2001). Academic Press
Genomi a DNA, Hepadnaviridae (HBV)
VII classe (RNA come stampo per sintesi di nuovi genomi)
Doppio filamento parziale, circolare
aperto, con associata la DNA
polimerasi RNA-dipendente
(trascrittasi inversa) al 5’ del
filamento (-)
Filamento (–) circa 3.5 kb, filamento
(+) variabile nelle diverse particelle,
1.7-2.8 kb
Il filamento (+) presenta un RNA con
cap al 5’
Alle terminazioni 5’ sono presenti delle
corte sequenze ripetute (DR1 e DR2),
importanti durante la
retrotrascrizione
From Flint et al. Principles of Virology (2000), ASM Press
Genomi a RNA (+)
Il genoma nudo di questi virus è infettivo
Picornavidae
• Dimensioni dalle 7.2 alle 8.5 kb, contiene una lunga UTR al 5’
(600-1200nt) importante per la traduzione, sequenze IRES
(Internal Ribosome Entry Site); UTR al 3’ più corta (50-100
nt) importante per la sintesi del filamento (-)
• Codifica una unica poliproteina
• Entrambe le estremità del genoma sono modificate: VPg al 5’
e poliadenilzione al 3’
From Cann Principles of molecular virology (2001). Academic Press
Genomi a RNA (+)
da 7.2 a 8.5 kb
10.5 kb
11.7 kb
30 kb
From Cann Principles of molecular virology (2001). Academic Press
Genomi a RNA (-)
• In questa classe sono presenti virus con genoma
costituito da una molecola continua
(Mononegavirales) o suddiviso in più segmenti.
• Nessuno di questi genomi è infettivo come RNA
purificato in quanto non è riconosciuto come
mRNA
• Tutti i virioni possiedono associata al genoma la
polimerasi virale che sarà utilizzata, all’inizio
dell’infezione, per produrre i messaggeri a
partire dallo stampo RNA(-)
Genomi a RNA (-), Rhabdoviridae
•
Circa 11kb. Contiene una
regione leader di 50nt al 3’ e
una breve UTR al 5’. E’
presente un segnale di
poliadenilazione alla fine di
ogni gene all’interno di corte
sequenze intergeniche tra i
5 geni
P
M
From Cann Principles of molecular virology (2001). Academic Press
Genomi a RNA (-), Rhabdoviridae
Genomi a RNA (-),
Orthomyxoviridae
I segmenti genomici
non sono
impacchettati come
acido nucleico nudo,
ma associati alla
nucleoproteina e
sono visibili al M.E.
come strutture
elicoidali
From Flint et al. Principles of Virology (2000), ASM Press
Genomi a RNA (-),
Orthomyxoviridae
• Genoma segmentato, 8 segmenti (virus
influenza A e B)
From Cann Principles of molecular virology (2001). Academic Press
Genomi a RNA (-),
Orthomyxoviridae
I segmenti hanno sequenze nucleotidiche comuni al 5’ e al 3’ importanti
per la replicazione: sono complementari tra di loro e all’interno del virione
i segmenti genomici sono ripiegati grazie all’appaiamento di basi presenti
alle estremità
Genomi a RNA a doppio filamento,
Reoviridae (Reovirus)
• Il genoma è costituito da
10 segmenti di RNA a
doppio filamento ed è
impachettato in un capside
icosaedrico costituito da 2
o 3 involucri concentrici. Il
virione è costituito dalle
proteine strutturali vere e
proprie che formano il
capside e da proteine non
strutturali meno
abbondanti che
comprendono la trascrittasi
virus-associata e tutti gli
enzimi necessari per la
produzione di mRNA virale,
inclusi quelli coinvolti nel
capping e nella metilazione
From Wagner and Hewlett Basic virology (2003) Blackwell Science Press
Retroviridae: virione
Genoma dei retrovirus
4 caratteristiche principali:
1) sono gli unici virus diploidi:
contengono due copie di
RNA(+) genomico
2) Sono gli unici virus che
richiedono uno specifico
RNA cellulare (tRNA) come
innesco per la polimerasi
From Flint et al. Principles of Virology (2000), ASM Press
Genoma dei
retrovirus
Retrotranscription
3) Sono gli unici virus ad
RNA(+) il cui genoma non
è utilizzato direttamente
come mRNA
4) Sono gli unici virus il
cui genoma è prodotto
attraverso la normale
trascrizione ad opera di
enzimi cellulari (RNA pol
II) a partire da DNA
From Cann Principles of molecular virology (2001). Academic Press
Genoma dei retrovirus
R è una corta sequenza che forma ripetizioni dirette ad entrambe le
estremità del genoma, importante durante il processo di
replicazione, segnale di poliadenilazione
U5 sequenza unica non codificante, prima parte del genoma ad essere
retrotrascritta, sito importante per l’integrazione
PBS 18nt complementari al 3’ del tRNA
Leader (90-500nt) non tradotta, contiene SD e y site
PPT (c.a. 10nt) sito di inizio del filamento + di DNA
U3 regione unica non codificante (200-1200nt) contenente elementi
promoter




![Lezione 15 Virus [modalità compatibilità]](http://s1.studylibit.com/store/data/000771737_1-84b1cca561c5813066d1b76125338a98-300x300.png)







