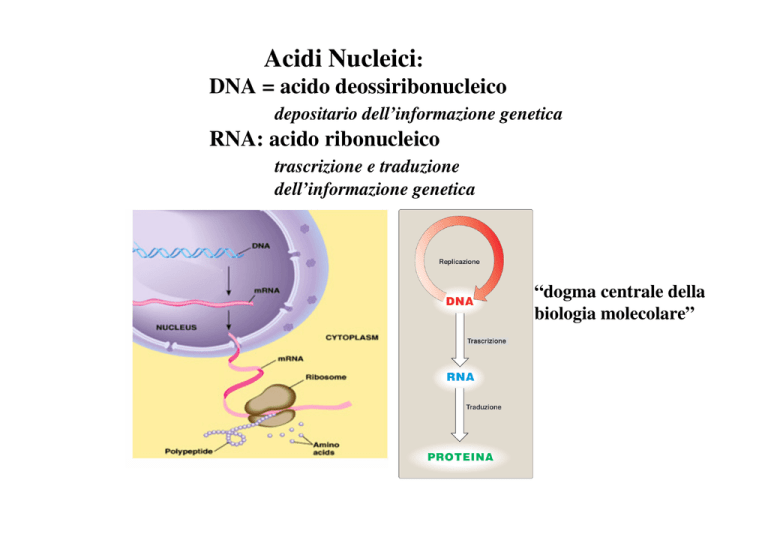
Acidi Nucleici:
DNA = acido deossiribonucleico
depositario dell’informazione genetica
RNA: acido ribonucleico
trascrizione e traduzione
dell’informazione genetica
“dogma centrale della
biologia molecolare”
(deossinucleotidi)
(nucleotidi)
Gli acidi nucleici sono polimeri
lineari di nucleotidi
Oligonucleotidi…
Polinucleotidi…
Appaiamento complementare delle basi:
A-T (2 legami H)
C-G (3 legami H)
Interazioni di van der
Waals tra coppie di
basi sovrapposte
(impilamento – stacking)
Per ogni molecola di DNA il contenuto di A è uguale al contenuto di T
e il contenuto di C è uguale al contenuto di G (regola di Chargaff)
10.5
Coppie di basi
per giro
- 34 Å Diametro
dell’elica
uniforme:
- 20 Å -
Filamenti
antiparalleli
Immagine di diffrazione dei
raggi X di una molecola di DNA
(R. Franklin & M. Wilkins)
Struttura a doppia elica destrorsa
del DNA (Watson e Crick, 1953)
• http://www.youtube.com/watch?v=ZGHkH
MoyC5I
Eliche destrorse
bp = base pair
Kb = migliaia di coppie di basi
Elica sinistrorsa
(favorita da alternanza
purine-pirimidine,
es: pCGCGCG….)
bp/giro
10.5
Diametro (Å)
20
Inclinazione basi 6°
11
26
20°
solco maggiore
più profondo
solco minore
meno profondo
12
18
7°
solco maggiore poco
profondo
solco minore
stretto e profondo
Il DNA a doppia elica può essere denaturato
La denaturazione si accompagna ad un
aumento dell’assorbanza a 260 nm:
effetto ipercromico
a
La denaturazione del DNA
è un processo reversibile
La riassociazione dei filamenti
“annealing” è rapida e avviene
in una sola tappa se la separazione
è incompleta
Se i filamenti sono completamente
separati la riassociazione avviene
in due tappe: 1) appaiamento di
un breve segmento complementare
(fase più lenta); 2) rinaturazione
completa (fase rapida)
La capacità di riassociazione di filamenti polinucleotidici secondo
la regola della complementarietà delle basi viene sfruttata in molte
tecniche di biologia molecolare
Si possono formare ibridi
DNA-DNA
DNA-RNA
RNA-RNA
Quanto DNA è contenuto in una cellula?...
bp = Base Pair (coppie di basi)
lungh DNA/lungh struttura ospitante
E. coli: 4,6x106 bp (1,7 mm)
850 (unica molecola
circolare)
Cellula umana: 3,2x109 bp (~2 m)
~4x105 (46 cromosomi)
Modalità di compattamento del DNA:
- Superavvolgimento
- associazione con proteine in strutture ordinate
Superavvolgimento
DNA rilassato
-Superavvolgimento negativo
(rotazione nella direzione opposta all’ avvolgimento dell’elica)
-Superavvolgimento positivo
(rotazione nella stessa direzione di avvolgimento dell’elica)
Il superavvolgimento
positivo compatta
il DNA e rende più difficile
la separazione dei filamenti
dell’elica
Il superavvolgimento
negativo deriva dal
parziale srotolamento
della doppia elica e
facilita la separazione
dei filamenti dell’elica
Il DNA è mantenuto in uno stato superavvolto perché le
catene non sono libere di ruotare su se stesse
(DNA circolare, nei procarioti – associazione con proteine,
negli eucarioti)
è richiesta la separazione dei filamenti dell’elica
La separazione dei filamenti dell’elica induce
superavvolgimento positivo
http://www.youtube.com/watch?v=BjcJu-o3YwY
I superavvolgimenti generati dalla separazione dei filamenti
dell’elica durante la replicazione o la trascrizione vengono
rimossi dalle DNA topoisomerasi
Come funzionano le TOPOISOMERASI?
TOPOISOMERASI DI TIPO I
- Monomeri
- Tagliano un singolo filamento
- Non consumano ATP
TOPOISOMERASI DI TIPO II
- Omodimeri (eucarioti)
eterotetrameri (batteri)
- Tagliano entrambi i filamenti
- richiedono ATP
Tutte le topoisomerasi formano un intermedio covalente enzima-DNA
Meccanismo d’azione delle topoisomerasi di tipo I
http://www.youtube.com/watch?v=
EYGrElVyHnU
In una cellula umana, ≈ 2 m di DNA sono impacchettati in
un nucleo di pochi μm di diametro!
DNA + proteine = cromatina
cromosomi
DNA nucleosomale:
146 bp + ∼ 60 bp (linker)
Istoni: proteine basiche
(ricche in Lys e Arg)
L’interazione con il DNA
è di tipo elettrostatico e
sequenza-indipendente
Livelli di organizzazione della cromatina
In una cellula umana, ≈ 2 m di DNA sono impacchettati in un nucleo di pochi µm di diametro!
• http://www.youtube.com/watch?v=3offBO3
sweg
Le modificazioni post-traduzionali
degli istoni regolano lo stato di
compattazione della cromatina
e quindi l’accessibilità del DNA
all’interazione con
fattori di regolazione
Il legame fosfodiestere può essere idrolizzato da nucleasi
(fosfodiesterasi)
-Ribonucleasi (RNAasi)
-Deossiribonucleasi (DNAasi)
-enzimi di restrizione = endonucleasi sequenza-specifiche
Il legame fosfodiestere può essere idrolizzato da
nucleasi
Ribonucleasi (RNAasi)
Deossiribonucleasi (DNAasi)
endonucleasi
enzimi di restrizione = endonucleasi sequenza-specifiche
Sequenze PALINDROMICHE
nei siti di taglio per gli enzimi di restrizione
Biosintesi del DNA: replicazione
Caratteristiche:
-Semiconservativa
-Bidirezionale
-Fedele
-Semidiscontinua
Principali enzimi coinvolti:
-DNA polimerasi
-DNA ligasi
-Primasi
-Elicasi
-Topoisomerasi
Nei procarioti la replicazione del DNA inizia in un unico sito
(origine della replicazione)
Negli eucarioti la replicazione inizia in più siti
Sia nei procarioti che negli eucarioti la replicazione è bidirezionale
Inizio della replicazione nei procarioti
La sintesi delle nuove molecole di DNA è catalizzata da
DNA polimerasi
LA DNA POLIMERARI CATALIZZA L’AGGIUNTA SEQUENZIALE DI UN
DEOSSIRIBONUCLEOTIDE ALL’ESTREMITA’ 3’-OH DI UNA CATENA
POLINUCLEOTIDICA ACCOPPIATA ADUN FILAMENTO STAMPO
(DNA)n residui +dNTP
(DNA)n+1+PPi
-Stampo
-3’-OH libero (innesco/catena in allungamento)
-Nucleosidi 5’-trifosfato (tutti e quattro i nucleotidi
devono essere presenti)
-Mg2+
La sintesi delle nuove molecole di DNA è catalizzata da DNA polimerasi
Reazione catalizzata dalla
DNA polimerasi
Allungamento della catena in direzione 5’ → 3’
Attività esonucleasica 3’
→
5’ per la correzione degli errori
Che cosa è richiesto per la replicazione del DNA….
-Stampo
-3’-OH libero (innesco/catena
in allungamento)
-Nucleosidi 5’-trifosfato (tutti
e quattro i nucleotidi devono
essere presenti)
-DNA polimerasi
-Mg2+
La replicazione è un processo molto accurato (replicazione fedele)
Frequenza di errore in E. coli: 1 errore ogni 109-1010 nucleotidi aggiunti
(1 errore ogni 1000-10000 replicazioni!)
Correzione degli errori:
attività ”proofreading” 3’
esonucleasica della DNA
Polimerasi I
5’
La DNA polimerasi
-Sintetizza DNA in direzione 5’
3’
-Richiede un “innesco” per la sua attività
Problema 1: poiché i due filamenti sono antiparalleli,
la crescita dei filamenti neosintetizzati deve avvenire
in direzioni opposte
Soluzione al problema 1:
-Sintesi continua nella direzione di avanzamento
della forcella di replicazione (filamento guida/catena veloce)
-Sintesi discontinua nella direzione di allontanamento dalla forcella di
replicazione (catena lenta: frammenti di Okazaki)
La replicazione del DNA è semidiscontinua
Problema 2: chi fornisce l’innesco?...
Soluzione:
DNA primasi: è una RNA polimerasi che
sintetizza un breve tratto di RNA (circa 10
nucleotidi) complementare allo stampo di
DNA (RNA primer).
La DNA primasi utilizza ribonucleosidi 5’trifosfato e catalizza la formazione del
legame fosfodiestere liberando PPi
La DNA polimerasi aggiunge poi
deossiribonucleotidi all’estremità 3’-OH
del RNA primer
La DNA polimerasi utilizza deossiribonucleotidi
5’-trifosfato e catalizza la formazione del
legame fosfodiestereo liberando PPi
La duplicazione del DNA nei Procarioti
La DNA polimerasi I (Pol I) è il principale enzima preposto alla duplicazione in E. coli.
Attività polimerasica 5’→ 3’ : sintesi DNA
Attività esonucleasica 3’→ 5’: correzione basi sbagliate inserite
Attività esonucleasica 5’→ 3’: riparazione DNA (rimozione basi in porzioni danneggiate del
DNA)
N
5’→3’ esonucleasica
3’→5’ esonucleasica
Polimerasi
C
Allungamento del primer di
RNA ad opera della Pol I
Sintesi di un nuovo RNA
primer
in
corrispondenza
dell’avanzare della forcina
Attività esonucleasica 5’→ 3’ rimozione frammenti di Okazaki
Il primer di RNA
all’estremità 5’ di un
frammento di Okazaki
viene eliminato grazie
all’attività
esonucleasica 5’→ 3’
della Poli I e sostituito
mediante la sua attività
polimerasica 5’→ 3’.
Il legame covalente è
ripristinato ad opera
della DNA ligasi
Rimozione del primer e
sostituzione con un tratto
di DNA da parte della DNA
polimerasi I
Okazaki
Fase di sintesi/allungamento
Limits of DNA polymerase
can only build onto 3′′ end of an
existing DNA strand
ents
m
g
a
r
f
ki
Okaza
3′′
5′′
3′′
5′′
5′′
5′′
3′′
ligase
growing
3′′
replication fork
5′′
5′′
3′′
Lagging strand
Leading strand
3′′
Lagging strand
Okazaki fragments
joined by ligase
5′′
3′′
DNA polymerase III
Leading strand
continuous synthesis
Attività esonucleasica 5’→ 3’
Riparazione DNA per rimozione di basi (10 basi) in porzioni
danneggiate del DNA dall’estremità 5’ di un DNA a singolo filamento
3’
5’
idrolisi
Quante DNA polimerasi?....replicazione
ricombinazione
riparazione
Processività = numero di
nucleotidi aggiunti prima
che la polimerasi si
dissoci
Velocità di replicazione
in E. coli =
circa 1000 nucleotidi/sec
Enzima principale della replicazione
riparazione
• DNA polymerase III
– 1000 nucleotidi/secondo!
– main DNA builder
Thomas Kornberg
1970
• DNA polymerase I
– 20 nucleotidi/second
– editing, repair & primer removal
DNA polymerase III
enzyme
Arthur Kornberg
1959
Saldatura dei frammenti di Okazaki
DNA LIGASI
La DNA ligasi catalizza l’unione di una catena di DNA la cui estremità libera
contiene un gruppo ossidrilico in posizione 3’ (3’-OH) con un’altra catena la cui
estremità libera contiene un gruppo fosforico in posizione 5’ (5’-P).
Reazione a più passggi
1. Attivazione del gruppo fosforico 5’ al
punto di interruzione
2. Formazione del legame fosfodiestereo
Reazione catalizzata dalla DNA ligasi:
1. Attivazione del gruppo fosforico 5’ al punto di interruzione:
1a) trasferimento di AMP ad un residuo di lisina dell’enzima
(nota: l’AMP può derivare dal NAD+ o dall’ATP)

![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)


![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)







