Clonare un gene significa isolarlo da un genoma ed inserirlo in un
vettore capace di replicarsi in un dato ospite (di solito E.coli o
lievito).
Esistono diversi tipi di vettori di clonaggio, ciascuno con diverse
caratteristiche.
La principale caratteristica che li contraddistingue é relativa alle
dimensioni dell'inserto di DNA che ogni vettore può accettare.
PLASMIDI
< 10 Kb
FAGI
< 23 kb
COSMIDI
PAC
30-46 kb
130-150 kb
BAC
< 300 kb
YAC
0.2-2 Mb
Plasmide
non integrativo
Episoma
Vettori Plasmidici
I vettori plasmidici sono derivati dai plasmidi batterici naturali. I
plasmidi sono degli elementi genetici extracromosomali che si
replicano autonomamente. Possono essere lineari o integrati nel
cromosoma batterico (episomi) ma, nella maggior parte dei casi,
sono molecole di DNA circolare.
Le caratteristiche comuni a qualsiasi vettore plasmidico sono:
• Origine di replicazione
• Marcatore selezionabile
• Siti di restrizione unici
Vettore
Plasmidico
Marcatori di Selezione
Tutti i vettori di clonaggio presentano almeno un marcatore selezionabile.
per selezionare le molecole ricombinanti. Inoltre, sotto un'appropriata
pressione selettiva, stabilizzano il plasmide.
I marcatori più utilizzati nei batteri sono i geni di resistenza agli antibiotici.
Per esempio il gene per la β-lattamasi codifica per un enzima capace di
idrolizzare l'anello lattamico degli antibiotici di tipo penicillinico. I batteri
che contengono un plasmide con questo gene quindi possono crescere in
terreni di coltura che contengono l'ampicillina.
Siti di restrizione
Per effettuare un clonaggio molecolare é necessario avere sempre almeno un
sito unico per un endonucleasi di restrizione, che non deve essere presente
in regioni cis essenziali (es. ori o promotori) o in geni che codificano per
funzioni essenziali (es. geni di resistenza).
Replicazione plasmidica
La caratteristica più importante dei plasmidi, quella di essere dei repliconi ,
cioè molecole capaci di replicazione autonoma, è conferita loro dalla presenza
di un origine di replicazione, chiamata ori
L origine di replicazione controlla:
• Il numero di copie
• La specificità d ospite
• I gruppi di incompatibilità I plasmidi si replicano con due modalità diverse.
• in maniera coordinata con la replicazione del cromosoma
batterico, generalmente quelli di grandi dimensioni, e si dicono a
controllo stringente. In genere a basso numero di copie, codificano
per proteine necessarie alla loro replicazione.
• in maniera indipendente dalla replicazione batterica, in genere di
piccole dimensioni, e si dicono a controllo rilassato. Ad alto
numero di copie, generalmente dipendono da proteine dell ospite
per la loro replicazione.
Plasmide
ori
numero di copie
PBR322 e derivati
PMB1
15-20
PUC e derivati
ColE1
500-700
pACYC e derivati
p15A
10-12
pSC101 e derivati
pSC10
1 -5
Un classico vettore plasmidico
Vettore episomale per la trasfezione
transiente di cellule di mammifero
MCS: Multiple Cloning Site
PCMV:
promotore del citomegalovirus
PA:
segnale di poliadenilazione
SV40 ori: sequenza di DNA corrispondente
all origine di replicazione di SV40
Neo:
gene per la selezione codificante
la neomicina fosfotransferasi e conferisce
alle cellule resistenza all antibiotico G418
Ori:
origine di replicazione di E.coli
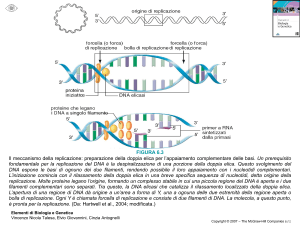
I Batteriofagi
I batteriofagi si presentano come placche di lisi su popolazioni batteriche
cresciute a confluenza su terreni solidi. Ogni placca rappresenta un clone. Tra i
batteriofagi più noti, il fago λ capace di integrarsi nel genoma batterico o il
batteriofago µ, che si integra a caso nel cromosoma batterico sfruttando
l'enzima transposasi. Il batteriofago λ può utilizzare due stili di vita all'interno
del batterio: il ciclo litico e il ciclo lisogenico
Nel ciclo litico il
batteriofago si replica,
si assembla in virione
maturo e lisa la cellula
ospite. Nel ciclo
lisogenico invece il
fago é capace di
integrare il proprio
DNA nel cromosoma
b a t t e r i c o ,
mantenendolo in uno
stato profagico
inattivo.
Il ciclo lisogenico
Il ciclo litico
In laboratorio, la replicazione e la crescita del fago λ avviene in capsule Petri. Il
fago viene mescolato con una coltura di E.coli ad alta densità, in una soluzione
di agar chiamata top agar e la soluzione viene versata sulla superficie di una
piastra di coltura ed incubata a 37°C. La crescita di λ produce la lisi delle
cellule batteriche e viene visualizzata come placche di lisi.
I vettori derivati dal fago λ
Il DNA di λ è una molecola lineare a doppio filamento di 48,5 Kb, caratterizzata
dalla presenza alle estremità 5 di due estremità coesive a singola elica di 12 nt,
chiamate siti cos (cohesive ends), che permettono la circolarizzazione del DNA
di λ dopo l infezione della cellula ospite. Il genoma di λ comprende circa 40 geni
che possono essere suddivisi in tre gruppi funzionali:
• La regione sinistra codifica per proteine strutturali della testa e della coda.
• La regione centrale contiene geni responsabili per la lisogenia. Gran parte di
questa regione non é essenziale per la crescita litica e può essere eliminata per
la costruzione di vettori.
• La regione destra contiene geni coinvolti nella replicazione del DNA e nel ciclo
litico.
Applicazioni biotecnologiche di λ
Il principale campo di applicazione dei batteriofagi, consiste nello sviluppo di
diversi tipi di vettori per biologia molecolare. Il più utilizzato è il fago λ, ma
anche M13, T3, T7 e f1.
Packaging in vitro
In una normale infezione litica, λ comincia a replicarsi, producendo
lunghi concatenameri, Questi vengono successivamente ridotti in
frammenti lineari, delimitati dalle estremità cos, e impacchettati
all interno di un capside vuoto. In seguito all assemblaggio con una
coda, si ricostituisce un virione maturo e infettivo. Mescolando il DNA
da inserire, con due colture di fagi difettivi, uno incapace di
introdurre il DNA nei capsidi vuoti e l altro incapace di produrre
capsidi vuoti, si riesce a produrre un packaging efficiente.
Cosmidi
Un cosmide è un plasmide, ~ 5 Kb, un ibrido tra plasmide e fago, che contiene:
un marker selettivo, un origine di replicazione, siti di clonaggio, sequenze
cos. La dimensione di un inserto che può essere clonato in un cosmide varia
tra 30 e 46Kb.
Invece di trasformare la miscela di ligazione per trasformazione, vista la
dimensione media di un cosmide ricombinante, si può utilizzare il packaging
in vitro. Il cosmide infatti possiede un sito cos è può essere considerato un
buon substrato per una reazione di packaging, purchè abbia dimensione
comprese tra 37 e 51 Kb.
Poiché i cosmidi sono privi di geni
di λ, quando il DNA si trova nelle
cellule di E.coli viene mantenuto
come plasmide
PAC
BAC
• stabili a basso numero di copie
• si basano sull uso del cromosoma
• basati sul batteriofago P1, per clonare
artificiale batterico.
frammenti di DNA <= 100 kb
• Derivano dal fattore sessuale F a
• contengono un sito pac, necessario
singola copia di E.coli e contengono
per l impaccamento del DNA
i siti cosN (di λ) e loxP (di P1), due
ricombinante nelle particelle virali, e
siti di clonaggio e vari siti di
due siti loxP
restrizione
• I siti loxP sono specifiche sequenze
•
riconosciute dalla ricombinasi fagica,
batteriche con buona efficienza di
prodotta dal gene cre, che portano alla
trasformazione
circolarizzazione del DNA quando
•
questo è iniettato all interno di una
frammenti di DNA <= 300kb
cellula ospite che esprima cre
• Utilizzati per produrre genoteche
•
Utilizzati per la produzione di
genoteche di lievito, topo, Drosophila,
uomo
Vengono introdotti in cellule
Permettono il clonaggio di
di taglia media pari a 125 kb.
I fosmidi sono simili ai cosmidi ma sono basati sul plasmide F
batterico. Il plasmide è a basso numero di copie, infatti l’ospite E. coli
ne contiene solo uno. I fosmidi hanno una taglia di circa 40 kb. I
fosmidi sono utilizzati per preparare librerie genomiche. Il basso
numero di copie comporta maggiore stabilità rispetto a plasmidi ad
alto numero di copie. Infatti i fosmidi vengono utilizzati per preparare
librerie di genomi complessi. Sono stati utilizzati anche per il
sequenziamento del genoma umano.
Vettori di clonaggio eucariotici:
vettori per lievito
I lieviti sono molto utilizzati come organismo ospite perchè, pur essendo unicellulari, sono
organismi eucariotici.
In generale esistono 2 tipi di vettori in lievito: i vettori d integrazione, che non possono
replicare e vengono mantenuti per integrazione nel cromosoma dell ospite e i vettori a
replicazione autonoma, che invece possono replicarsi autonomamente.
Vettori di integrazione
Un tipico vettore d integrazione, chiamato YIp (Yeast Integrative plasmid) è basato su un
vettore plasmidico entro il quale si clona la sequenza d interesse. Una volta trasformato in
lievito il plasmide verrà mantenuto solo dopo integrazione in un cromosoma mediato dalla
ricombinazione omologa tra un marcatore funzionale di lievito e il corrispondente
marcatore difettivo in un lievito mutante
Vettori autonomi
I vettori a replicazione autonoma assicurano alte frequenze di trasformazione e rese più
alte.
YEp (Yeast Episomal plasmid): basati su un plasmide endogeno di lievito di tipo episomale,
il plasmide 2 µ.
YCp (Yeast Centromere plasmid) e YAC (Yeast Artificial Chromosome): plasmidi con
sequenze ARS (Autonomous Replicating Sequence) come centromeri o telomeri
Yeast Integrative Plasmid
Yeast Replicative Plasmid
Yeast Episomal Plasmid
Yeast Centromere Plasmid
Una delle principali differenze tra i vettori plasmidici e quelli di lievito
risiede nei marcatori di selezione. Infatti, i lieviti sono molto meno resistenti
agli antibiotici e la selezione viene di solito effettuata sfruttando la
complementazione di mutazioni auxotrofiche nel ceppo di lievito ospite
Ap
ori
YEp
trp1
ori 2µ
Per esempio, un ceppo di Saccharomices cerevisiae contenente una
mutazione nel gene trp1 non sarà in grado di crescere su un terreno privo di
triptofano. Se, però, il vettore plasmidico porta un gene trp1 funzionale, i
trasformanti potranno crescere selettivamente su terreni privi di triptofano.
Tra i marcatori più comunemente impiegati, oltre a trp1, ura3 (uracile), leu2
(leucina) e his3 (istidina).
Inserimento di un gene in un vettore Yip
per ricombinazione omologa
Ap
ori
YIp
Inserto
ura3
Ricombinazione omologa
Ura3- difettivo
ura3
Ap
ori
Inserto
ura3
YAC
Nel caso di genomi grandi e complessi, come quelli dei mammiferi o delle
piante superiori, non è opportuno utilizzare genoteche cosmidiche o, ancor
meno fagiche o plasmidiche. Il numero di cloni necessari per coprire
statisticamente questi genomi sarebbe troppo grande.
Il metodo utilizzato quindi consiste nel suddividere il genoma in frammenti
molto più grandi, utilizzando appositi vettori come gli YAC (Yeast Artificial
Chromosome)
I vettori YAC possono accettare inserti grandi fino a 0,5-2Mb, anche se
l efficienza di trasformazione è molto bassa. Vengono mantenuti e propagati
in E.coli
Uno YAC è costituito essenzialmente da:
• un centromero di lievito
• due telomeri di lievito
• un origine di replicazione di lievito (ARS)
• uno o più siti unici per enzimi di restrizione
• un marcatore di selezione (di tipo auxotrofico)
Clonaggio in vettori YAC
Trasformazione in cellule di lievito Ade-, Ura-, Trpe selezione per colonie rosse, URA+, TRP+
Protocollo
1. Digerire parzialmente il DNA bersaglio con EcoRI e il
vettore YAC con EcoRI e BamHI
2. Separare i due bracci e rimuovere l eventuale
frammento stuffer
3. Ligare vettore YAC e inserto
4. Trasformare le cellule di lievito ade-, ura-, trp-,
selezionando per i 2 marcatori posizionati ciascuno su un
braccio diverso
5. Cellule ade- danno origine a colonie rosse, in presenza di
SUP4 si ha soppressione dell effetto della mutazione e
colonie bianche. Plasmidi YAC ricombinanti hanno
inattivato SUP4 e quindi danno origine a colonie di colore
rosso
5 dei sistemi di
clonaggio utilizzati
per costruire le
mappe per
sequenziare il
genoma umano
Il Primo Organismo Modello
Escherichia coli
Il Primo Organismo Modello eucariote
Saccharomyces cerevisiae
Trasformazione
di cellule procariotiche ed eucariotiche
L introduzione di DNA esogeno in una cellula viene generalmente
definita trasformazione.
La possibilità di trasferire DNA esogeno in un organismo modello è alla
base delle tecniche di ingegneria genetica. L efficienza di questo
passaggio è cruciale per garantire il successo di qualunque clonaggio.
Le tecniche di trasferimento genico permettono oggi di trasferire ad
alta efficenza, ed in modo controllato DNA ricombinante in una cellula
ospite capace di replicarlo.
Trasformazione: trasferimento di DNA in cellule ospiti
Trasfezione: trasferimento genico mediato da batteriofagi o virus
Trasferimento genico
mediante elettroporazione
Le cellule, in una ssospensione
acquosa ad alta concentrazione,
vengono incubate con il DNA e poste
in una piccola cella con 2 elettrodi
collegati ad uno speciale alimentatore.
Un breve impulso elettrico, emesso tra
i 2 elettrodi, induce l apertura di
piccoli fori sulla membrana cellulare.
Le cellule vengono poi diluite in
terreno liquido ed incubate in
agitazione per almeno un’ora
(recovery).
Le cellule possono essere poi
utilizzate per esperimenti in
transiente, o piastrate su terreno con
antibiotico per selezionare i cloni
positivi.
Espressione stabile
Espressione transiente
Trasformazione chimica
Recovery
Periodo di recupero in terreno liquido che permette al plasmide
di replicarsi nelle cellule in cui è entrato
Durante il recovery le cellule trasformate possono esprimere il
gene per la resistenza all’antibiotico
Efficienza di trasformazione
• Calcolare i mg totali di DNA usati per la trasformazione
• Calcolare il fattore di diluizione usato per piastrare i trasformanti
• Contare le colonie
• Moltiplicare n.colonie (CFU) x fattore diluizione
• Dividere per i mg di DNA utilizzato
Efficienza di trasformazione CFU/mg
Controlli di trasformazione:
Ligazione del solo vettore
Vettore chiuso a concentrazione nota
Vettore digerito
Selezione di vettori ricombinanti
A seguito di ligazione di un inserto con un vettore e introduzione di
questo costrutto in un ospite, si possono ottenere diversi tipi di
trasformanti:
• batteri vuoti
• batteri contenenti il solo vettore
• batteri contenenti il vettore ligato all'inserto
Come selezionare i cloni ricombinanti da quelli contenenti solo il
vettore???
Si può ricorrere a metodi generali e laboriosi come l'ibridazione su
colonia (colony hybridization) o un'analisi per digestione con
enzimi di restrizione (se i cloni trasformanti sono pochi), o la PCR
su colonia
Molti vettori di clonaggio sono costruiti per facilitare lo screening
dei cloni ricombinanti.
Specificità d ospite
Alcuni plasmidi sono in grado di replicare in un numero limitato di specie batteriche e
si dicono a specificità d'ospite limitata (narrow host range). Altri sono in grado di
replicarsi in una vasta gamma di specie batteriche e si dicono a largo spettro d'ospite
(broad host range).
I plasmidi a largo spettro d'ospite devono questa loro proprietà al possesso di alcuni
geni necessari per il riconoscimento dell'origine di replicazione. Dipendono meno,
quindi, dall'apparato replicativo dell'ospite batterico.
Gruppi di incompatibilità
Plasmidi con la stessa origine di replicazione (Inc) sono incompatibili tra loro.
I
plasmidi dipendono per la loro replicazione da componenti genetiche dell'ospite
batterico in grado di riconoscere l'origine di replicazione e iniziare la replicazione. Se
in uno stesso batterio entrano due plasmidi con la stessa origine di replicazione, questa
compete per componenti proteici comuni. Come risultato nel giro di poche generazioni
uno dei due plasmidi verrà perso. Possono invece coesistere plasmidi con origine di
replicazione diverse che appartengono a diversi gruppi di incompatibilità