COGNOME NOME
QUALIFICA
INDIRIZZO
TELEFONO
MAIL
WEB PAGE
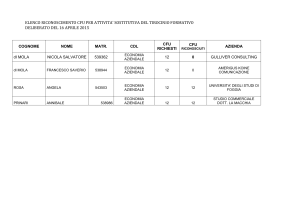
Rizzi Raffaella
Ricercatore confermato (INF/01-Informatica)
viale Sarca, 336/14, 20126 Milano
02 6448 7838
[email protected]
CARRIERA
ACCADEMICA
Da Dicembre 2008: ricercatore presso il Dipartimento di
Informatica Sistemistica e Comunicazione dell’Università degli
Studi di Milano-Bicocca (settore INF/01).
Da Febbraio 2007 a Dicembre 2008: contratto di Ricercatore
di chiara fama sul tema Progettazione, sviluppo e
realizzazione di un software per l’analisi computazionale
dello splicing alternativo di geni eucariotici (responsabile:
Dott. Giulio Pavesi) presso il Dipartimento di Scienze
Biomolecolari e Biotecnologie dell’Universita degli Studi di
Milano.
Da Aprile 2006 a Gennaio 2007: incarico di collaborazione
presso il Dipartimento di Informatica Sistemistica e
Comunicazione dell’Università degli Studi di Milano-Bicocca.
Da Aprile 2005 a Marzo 2006: assegnista di Ricerca
(responsabile: Prof. Piercarlo Fantucci) presso il Dipartimento
di Biotecnologie e Bioscienze dell’Università degli Studi di
Milano-Bicocca.
Da Aprile 2001 a Marzo 2005: assegnista di Ricerca in
Bioinformatica sul tema “Metodi per l’analisi e il confronto di
sequenze biologiche” (responsabile: Prof. Paola Bonizzoni)
presso l’Università degli Studi di Milano-Bicocca.
Da Gennaio 1998 a Marzo 2001: collaborazione con la sezione
di Bioinformatica (responsabile: Dott. Luciano Milanesi)
dell’Istituto di Tecnologie Biomediche Avanzate del CNR di
Segrate (Milano) finalizzata allo svolgimento della tesi di
dottorato.
AA 2006/2007
Corso di Informatica - CdL in Biotecnologie (I livello
e Vecchio Ordinamento), Facoltà di Scienze
Matematiche Fisiche e Naturali dell’Università degli
Studi di Milano-Bicocca.
AA 2007/2008
Corso di Informatica - CdL in Biotecnologie (I livello
e Vecchio Ordinamento), Facoltà di Scienze
Matematiche Fisiche e Naturali dell’Università degli
Studi di Milano-Bicocca.
Laboratorio di Bioinformatica: tecniche di base I
MOD (2 crediti)
–
CdL
Specialistica in
Bioinformatica, Facoltà di Scienze Matematiche
Fisiche e Naturali dell’Università degli Studi di
Milano-Bicocca (Docente: Prof. Giancarlo Mauri).
AA 2008/2009
Laboratorio per l’insegnamento di Bioinformatica:
DIDATTICA
ATTIVITA’ DI RICERCA
Tecniche di Base, modulo Bioinformatica: Tecniche
di Base I mod. - 2 CFU – CdL Specialistica in
Bioinformatica.
Esercitazione per l’insegnamento di Fondamenti
dell’Informatica, modulo Complessità - 1 CFU – CdL
Magistrale in Informatica.
AA 2009/2010
Affidamento
retribuito
dell'insegnamento
di
Informatica, modulo di Programmazione e modulo
di Basi di Dati – 6 CFU – CdL in Biotecnologie.
Laboratorio per l’insegnamento di Bioinformatica:
Tecniche di Base - 4 CFU – CdL Magistrale in
Bioinformatica.
Affidamento dell’insegnamento di Programmazione
- 4 CFU – CdL Magistrale in Bioinformatica.
AA 2010/2011
Laboratorio per l’insegnamento di Bioinformatica:
Tecniche di Base - 4 CFU
Affidamento dell’insegnamento di Programmazione
- 4 CFU – CdL Magistrale in Bioinformatica.
Esercitazione per l’insegnamento di Modelli e
Sistemi, modulo Disegno ed Analisi di Algoritmi - 1
CFU – CdL Magistrale in Informatica.
AA 2011/2012
Affidamento dell’insegnamento di Informatica - 8
CFU (di cui 3 CFU retribuiti) – CdL in Biotecnologie.
Esercitazione per l’insegnamento di Metodologie
Bioinformatiche - 1 CFU – CdL Magistrale in
Biotecnologie Industriali.
AA 2012/2013
Affidamento dell’insegnamento di Informatica - 8
CFU (di cui ? CFU retribuiti) – CdL in Biotecnologie.
Esercitazione per l’insegnamento di Metodologie
Bioinformatiche - 1 CFU – CdL Magistrale in
Biotecnologie Industriali.
Esercitazione per l’insegnamento di Bioinformatica –
2 CFU – CdL Magistrale in Informatica.
Il principale interesse di ricerca è il disegno di metodi
computazionali per la biologia molecolare, con particolare
interesse al problema di predizione della struttura genica e
delle isoforme potenzialmente espresse.
In particolare, RR ha lavorato al disegno, all’implementazione
e alla sperimentazione di algoritmi efficienti per predire, a
partire da sequenze EST e RNA-Seq, la struttura di un gene in
esoni e introni, e per predire i trascritti alternativi.
Ha lavorato al disegno e all’implementazione dei software
tool ASPic e PIntron, che si collocano nell’ambito del progetto
ASPIC (Alternative Splicing PredICtion), che ha condotto alla
realizzazione di ASPicDB, un database di alternative splicing
patterns (http://t.caspur.it/ASPicDB/).
- CdL M
PRINCIPALI
PUBBLICAZIONI
RELATIVE AGLI ULTIMI
CINQUE ANNI
T. Castrignanò, M. D’Antonio, et al. ASPicDB: a database
resource for alternative splicing analysis. Bioinformatics
(2008), 24(10):1300-1304.
P. Bonizzoni, G. Mauri, G. Pesole, E. Picardi, Y. Pirola, R.
Rizzi. Detecting alternative gene structures from spliced ESTs:
a computational approach. Journal of Computational Biology
(2009), 16(1):43-66.
P. Bonizzoni, G. Della Vedova, R. Dondi, Y. Pirola, R. Rizzi.
Minimum factorization agreement of spliced ESTs. Lectures
Notes in Bioinformatics (serie LNCS), Proceedings of 9th
Workshop on Algorithms in Bioinformatics WABI 2009,
Philadelphia 12-13 Settembre 2009, pp. 5724:1-12.
P. L. Martelli, M. DAntonio, P. Bonizzoni, T. Castrignanò, A.
M. D’Erchia, P. DOnorio De Meo, P. Fariselli, M. Finelli, F. L.,
M. Mangiulli, F. Mignone, G. Pavesi, E. Picardi, R. Rizzi, I.
Rossi, A. Valletti, A. Zauli, F. Zambelli, R. Casadio, G. Pesole.
ASPicDB: a database of annotated transcript and protein
variants generated by alternative splicing. Nucleic Acids
Research (2010), 39:D80-5.
Y. Pirola, R. Rizzi, E. Picardi, G. Pesole, G. Della Vedova, P.
Bonizzoni. PIntron: a fast method for detecting the gene
structure due to alternative splicing via maximal pairings of a
pattern and a text. BMC Bioinformatics (2012), 13(Suppl
5):S2.