CLASSIFICAZIONE DEI MICRORGANISMI
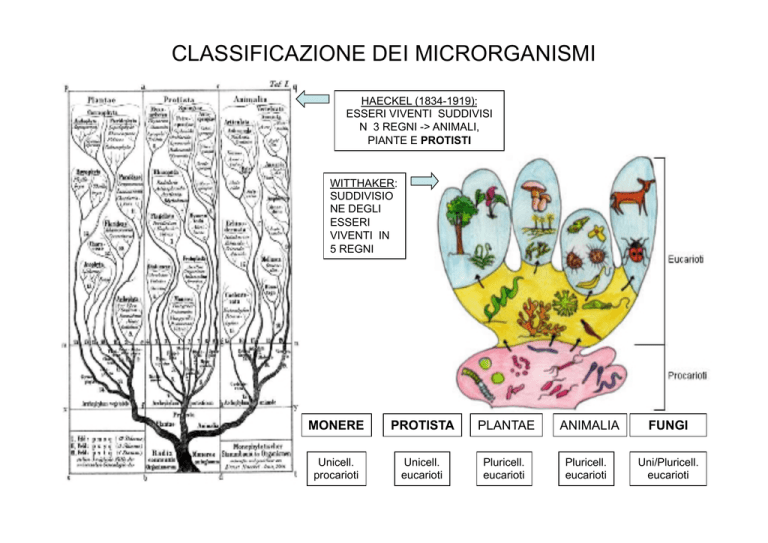
HAECKEL (1834-1919):
ESSERI VIVENTI SUDDIVISI
N 3 REGNI -> ANIMALI,
PIANTE E PROTISTI
WITTHAKER:
SUDDIVISIO
NE DEGLI
ESSERI
VIVENTI IN
5 REGNI
MONERE
PROTISTA
PLANTAE
Unicell.
procarioti
Unicell.
eucarioti
Pluricell.
eucarioti
ANIMALIA
Pluricell.
eucarioti
FUNGI
Uni/Pluricell.
eucarioti
CLASSIFICAZIONE E NOMENCLATURA DEI MICRORGANISMI
•
LE CARATTERISTICHE FENOTIPICHE FORNISCONO RELATIVAMENTE POCHE INFORMAZIONI
particolare importanza delle metodologie che si basano sul genotipo
•
•
COMPARAZIONE TRA SEQUENZE DI MACROMOLECOLE CON FUNZIONI OMOLOGHE IN DIFFERENTI SPECIE
COLLEZIONI DI MICRORGANISIMI E TYPE STRAINS
•
Bergey's Manual of Systematic Bacteriology
(caratteristiche fenotipiche impiegate per la tassonomia tradizionale e chiavi per l’identificazione di
microrganismi)
•
LA COMPOSIZIONE IN BASI E L’IBRIDAZIONE TRA ACIDI NUCLEICI
TASSONOMIA TRADIZIONALE E NUMERICA ->
Alcuni termini utili:
FILOGENESI: processo evolutivo degli organismi dal momento della loro comparsa sulla terra
fino ad oggi
TASSONOMIA: studio della classificazione e nomenclatura degli organismi viventi
SPECIE: unità principale della tassonomia, definita in base alle caratteristiche fenotipiche di
un’insieme di ceppi tra loro simili (per i batteri RNA 16S con omologia di sequenza >97%). PIÙ LUNGO È IL PERIODO DI TEMPO CHE SEPARA DUE ORGANISMI DA UN PROGENITORE COMUNE PIÙ
GRANDE SARÀ IL NUMERO DI DIFFERENZE NELLA SEQUENZA DI MACROMOLECOLE DI ANALOGA
FUNZIONE: METODI DI STUDIO BASATI SULL’ANALISI DEL RNA RIBOSOMALE
•
•
•
•
•
•
•
•
•
RNA RIBOSOMALE
molecole universalmente distribuite
funzione costante
facilmente purificabili facilmente sequenziabili (DNA primers e Reverse transcriptase) regioni che si sono evolute velocemente e altre che sono cambiate più lentamente → distanze
evolutive calcolabili in specie affini e non
costruzione di alberi filogenetici basati sulle sequenze di RNA 16S: gli organismi vengono
analizzati e situati in rami dell’albero genealogico distanziati gli uni da tutti gli altri in base
alle reciproche differenze di sequenza
identificazione di sequenze che sono uniche in particolari gruppi di microrganismi: quante più
sequenze saranno identificate tanto più facile sarà l’identificazione di microrganismi
“sconosciuti”
CONSEGUENZE
esistenza di Eubacteria e Archaea, tra loro evolutivamente distanti come, rispettivamente, dagli
Eucarya
gruppi diversi si sono evoluti a diverse velocità: lentamente gli Archaea, più velocemente gli
Eucarya
• origine endosimbiotica di mitocondri e cloroplasti
METODI DI IDENTIFICAZIONE E CARATTERIZZAZIONE
MOLECOLARE DI MICROGANISMI DI INTERESSE ENOLOGICO
IDENTIFICAZIONE:
ATTRIBUZIONE DI GENERE E SPECIE MEDIANTE
CONFRONTO CON INDIVIDUI GIÀ IDENTIFICATI
CARATTERIZZAZIONE:
BIOTIPIZZAZIONE, DIFFERENZIAZIONE DI CEPPI
NELL’AMBITO DELLA SPECIE DI APPARTENENZA
• conoscenza comunità microbica in uve, mosti, cantine
• individuazione di microganismi che agiscono in contemporanea e in successione nel corso delle
fermentazione
• valutazione del contributo di microganismi diversi alla fermentazione
• verifica della dominanza dei ceppi inoculati
• veriifca della presenza di contaminati
Carl Woese ---> 1977 scoperta degli Archea
---> 1989 utilizzo di RNA ribosomale per l’individuazione di
correlazioni filogenetiche
I geni codificanti per gli RNA ribosomali di diversi organismi vengono
sequenziati e le sequenze allineate e comparate --> maggiore è la
differenza nella sequenza degli RNA ribosomali di uno o più organismi e
maggiore è la loro distanza evolutiva
http://aulascienze.scuola.zanichelli.it/biologia-e-dintorni/2013/01/13/carl-woese-e-la-rivoluzione-degli-archaea/
Modificato da
L’ ESTRAZIONE DEL DNA
Il primo passo per molte tecniche molecolari e l’applicazione delle
biotecnologie è l’estrazione del DNA dal campione oggetto di analisi e/o
studio.
Il DNA ottenuto può essere valutato mediante corsa su un gel di agarosio e
può essere successivamente utilizzato per molteplici analisi molecolari.
BATTERI
GRAM +
GRAM –
(gram + hanno
spessa parete di
peptidoglicano:
protocollo di
estrazione
differente dai
gram- e simile
invece a quello
per i lieviti)
DA DOVE?
LIEVITI
FUNGHI
TESSUTO
VEGETALE
con
Kit di
estrazione
SUOLO
con
Kit di
estrazione
DNA e genoma, in riferimento ai procarioti
Estremità
5’
Estremità
3’
Brock - Biologia dei Microrganismi
STRUTTURA DEI LEGAMI TRA BASI PURINICHE (A, G)
PIRIMIDINICHE (T, C, U)
Brock - Biologia dei Microrganismi
Lastra radiografica
di sequenza
Esempio di cromatogramma inviato da un servizio di sequenziamento
RAPPRESENTAZIONE DELL’ALBERO FILOGENETICO DEGLI ORGANISMI
VIVENTI SECONDO LA COMPARAZIONE DELLE SEQUENZE DEGLI RNA
RIBOSOMALI
Modificato da
AMPLIFICAZIONE DI UNA
SEQUENZA
Reazione a catena della polimerasi
-
PCR - Polymerase chain reaction
•
•
•
•
•
•
K. B. Mullis, 1985
DNA polimerasi di Thermus aquaticus DNTPs desossinucleotidi trifosfati
Primers (oligonucleotidi complementari
alle regioni che fiancheggiano la sequenza
da amplificare
applicazioni in diagnostica, clonaggio,
sequenziamento, studi tassonomici
3 FASI: denaturing, denaturazione (94°C)
annealling, appaiamento (variabile
secondo primer, intorno 45-60°C
extension, allungamento, circa 75°C
Brock - Biologia dei Microrganismi
ELETTROFORESI SU GEL
L’elettroforesi in gel è il metodo standard utilizzato per separare e identificare
frammenti di DNA. Il gel può essere costituito da agarosio o da poliacrilamide.
La molecola del DNA è caricata negativamente e, quindi, in un campo elettrico
migrerà verso il polo positivo. La matrice porosa del gel ritarda la migrazione del
DNA, consentendo ai piccoli frammenti di spostarsi più velocemente rispetto ai
più grandi. Il risultato è che i frammenti di DNA si separeranno nella matrice in
funzione del peso molecolare. Più elevate concentrazioni si usano per separare le
molecole più piccole e, viceversa, concentrazioni più basse sono più adatte a
separare frammenti più grandi.
Il tampone di elettroforesi è una soluzione salina che serve sia a condurre la
corrente elettrica che a controllare il pH durante l’elettroforesi. Il tampone di
caricamento (colorante +addensante), che si aggiunge ai campioni prima
dell’elettroforesi, serve ad “appesantire” i campioni, permettendo la loro
permanenza nei pozzetti, e a fornire un marker visivo del progredire della corsa
elettroforetica.
Marcatori di peso molecolare (DNA marker)
IDENTIFICAZIONE DI SPECIE E CEPPO NEI PROCARIOTI
Determinazione della specie
•
•
•
ARDRA “Amplified ribosomal DNA restriction analysis” Amplificazione 16S
rDNA e successiva analisi di restrizione)
Amplificazione 23S rDNA
PCR specie-specifica
Determinazione del ceppo
•
•
RAPD (Random Amplified Polymorphic Dna)
AFLP (Amplified Fragment Length Polymorfism)
PCR specie-specifica
Utilizzazione di due primers che amplificano una regione specifica di un gene target 16
14
13
12
11
10
6
3
2
1
C
M
12 Oenococcus oeni
14 Lactobacillus sp.
Esempio di applicazioni:
•
Rapida identificazione di Oenococcus oeni primer On1 e On2 per l’enzima malolattico
(gene mle) specifico di Oenococcus oeni
•
Rilevamento di Oenococcus oeni direttamente in mosti e vini
RAPD
• i primers si possono legare alle sequenze bersaglio anche se non sono completamente
complementari e anche se la loro localizzazione nel genoma non è conosciuta • utilizzazione di un solo primer di sequenza arbitraria in condizioni di bassa stringenza
con innesco della reazione in più punti del genoma e ottenimento di profili intra speciespecifici
• se il legame PRIMER/SEQUENZA bersaglio avviene in posizione e orientamento che
permettono l’amplificazione si ottengono dei pattern caratteristici per ogni CEPPO
M
1
2
3
4
5
6
7
8
9
10
11 12
13 14
15 16
17 18
Profili RAPD di batteri malolattici con il primer DAF3
19
Brook, Biologia dei
microrganismi – 1
Microbiologia generale
19
Brook, Biologia dei
microrganismi – 1
Microbiologia generale
20
Brock - Biologia dei Microrganismi
DUPLICAZIONE
sintesi di una molecola di DNA a doppia elica identica al DNA duplex parentale
TRASCRIZIONE
processo enzimatico con cui l’informazione viene utilizzata per
specificare una sequenza complementare di basi in una catena di
mRNA
TRADUZIONE
processo attraverso cui l’informazione genetica presente in una
molecola di mRNA specifica la sequenza degli amminoacidi durante
la sintesi proteica
Brock - Biologia dei Microrganismi
DUPLICAZIONE (replication)
sintesi di una molecola di DNA a doppia elica identica al DNA duplex parentale
- anni ‘40 --> il DNA é la molecola depositaria del codice genetico
- 1953 --> Watson e Crick postulano il modello a doppia elica
- unicità delle proprietà fondamentali e dei meccanismi usati dagli
enzimi che la catalizzano in tutti gli organismi
Duplicazione
semiconservativa
ciascuna delle due catene funge da stampo per la sintesi
della seconda catena --> dopo ogni divisione cellulare la
metà del DNA é conservata intatta
#
la sintesi avviene in DIREZIONE 5’ --> 3’
#
la DNA POLIMERASI (1956, Arthur Kornberg) è responsabile della sintesi di una
delle catene di DNA utilizzando l’altra come matrice
#
la sintesi avviene producendo piccoli frammenti (frammenti di OKAZAKI) che poi
vengono uniti dall’enzima LIGASI
Brock - Biologia dei Microrganismi
TRASCRIZIONE (replication) E TRADUZIONE (translation)
Brock - Biologia dei Microrganismi
TRASCRIZIONE (transcription)
processo enzimatico con cui l’informazione viene utilizzata per specificare una
sequenza complementare di basi in una catena di mRNA
DNA --> RNA
mRNA = RNA messaggero
3 TIPI DI RNA
tRNA = RNA transfer
rRNA = RNA ribosomale
#
mRNA: molecola di RNA trascritta dal DNA; contiene l’informazione per dirigere
la sintesi di una specifica proteina
#
tRNA: molecola di RNA che trasporta amminoacidi al ribosoma durante la
TRADUZIONE; contiene ANTICODONI
#
rRNA: tipi di RNA che si trovano nei RIBOSOMI; alcuni partecipano attivamente
alla sintesi proteica
TRASCRIZIONE (transcription)
segue
- il processo di TRASCRIZIONE è SELETTIVO: solo particolari geni o
gruppi di geni vengono trascritti in una data circostanza
- ci sono nel DNA SEQUENZE REGOLATRICI che indicano l’INIZIO e
la FINE dei frammenti di DNA che devono essere trascritti
(diretta dal DNA)
RNA POLIMERASI
(diretta dal DNA)
necessita i 4 RIBONUCLEOTIDI 5’ FOSFATI (ATP, GTP, UTP,
CTP)
CONTIENE Zn
CATENA STAMPO (-)
CATENA NON STAMPO (+) = CATENA CODIFICANTE
5’
3’
TRADUZIONE (translation)
sintesi di una proteina usando l’informazione genetica della molecola di mRNA come stampo
CODONE
SEQUENZA di 3 BASI nel mRNA che codifica per un
AMMINOACIDO 4 BASI a gruppi di 3 possono dare 4 3 = 64 combinazioni
CODONE D’INIZIO
CODONI DI TERMINAZIONE
AUG
UAA UAG UGA (codoni STOP o codoni non senso)
IL CODICE GENETICO É DEGENERATO MA NON AMBIGUO
#
#
QUADRO DI LETTURA (reading frame)
no interruzioni, no sovrapposizioni, no intervalli
CODICE GENETICO
-
RICOMBINAZIONE GENETICA
processo per cui elementi genetici provenienti da genomi diversi
vengono ad essere riuniti in una stessa unità
scambio fisico di DNA tra elementi genetici
RECOMBINANT DNA (“DNA RICOMBINANTE”)
molecola di DNA contenente DNA proveniente da una o più origini
EUCARIOTI
# riproduzione sessuale PROCARIOTI
# trasformazione
# coniugazione
# trasduzione
TRASFORMAZIONE
introduzione di DNA esogeno in una cellula --> una molecola
di DNA libero viene incorporata in una cellula ricevente e determina
cambiamenti genetici
# Fred Griffith (1920) studi su Streptococcus pneumoniae # competenza
TRASDUZIONE
trasferimento dell’informazione genetica da una cellula ad
un’altra mediante un vettore virale
# Zinder e Lederberg (1952)
# trasduzione generalizzata e specializzata
# ciclo litico e ciclo lisogeno
CONIUGAZIONE
trasferimento di DNA da una cellula ad un’altra mediante
contatto diretto # Lederberg e Tatum (1946)
# plasmide F
# ceppi Hfr (high frequency of recombination)
F+
Brook, Biologia dei
microrganismi – 1
Microbiologia generale
33
HFR
LA CONIUGAZIONE BATTERICA RICHIEDE CONTATTO DIRETTO TRA CELLULA
DONATRICE CHE POSSIEDE IL PLASMIDE CONIUGATIVO E CELLULA RICEVENTE CHE
NON LO POSSIEDE.
IL PLASMIDE F (FERTILITÀ, 99159 BP) DI ESCHERICHIA COLI POSSIEDE:
- I GENI PER LA REPLICAZIONE DEL DNA
- ALCUNI ELEMENTI TRASPONIBILI CAPACI DI INTEGRARSI NEL CROMOSOMA
DELL’OSPITE
- LA REGIONE tra (TRA) RESPONSABILE DEL TRASFERIMENTO
CELLULE F+ -> PLASMIDE F NON INTEGRATO NEL CROMOSOMA
CELLULE HFR * -> PLASMIDE F INTEGRATO NEL CROMOSOMA
* HIGH FREQUENCY OF RECOMBINATION
LA PRESENZA DEL PLASMIDE F INDUCE:
- LA SINTESI DEL PILO F
- LA MOBILIZZAZIONE DEL DNA PER TRASFERIRLO AL RICEVENTE
- L’INCAPACITA DELLA CELLULA DI COMPORTARSI DA RICEVENTE
Brook, Biologia dei
microrganismi – 1
Microbiologia generale
35
LA CONIUGAZIONE BATTERICA RICHIEDE CONTATTO DIRETTO TRA CELLULA
DONATRICE CHE POSSIEDE IL PLASMIDE CONIUGATIVO E CELLULA RICEVENTE CHE
NON LO POSSIEDE.
IL PLASMIDE F (FERTILITÀ, 99159 BP) DI ESCHERICHIA COLI POSSIEDE:
- I GENI PER LA REPLICAZIONE DEL DNA
- ALCUNI ELEMENTI TRASPONIBILI CAPACI DI INTEGRARSI NEL CROMOSOMA
DELL’OSPITE
- LA REGIONE tra (TRA) RESPONSABILE DEL TRASFERIMENTO
CELLULE F+ -> PLASMIDE F NON INTEGRATO NEL CROMOSOMA
CELLULE HFR * -> PLASMIDE F INTEGRATO NEL CROMOSOMA
* HIGH FREQUENCY OF RECOMBINATION
LA PRESENZA DEL PLASMIDE F INDUCE:
- LA SINTESI DEL PILO F
- LA MOBILIZZAZIONE DEL DNA PER TRASFERIRLO AL RICEVENTE
- L’INCAPACITA DELLA CELLULA DI COMPORTARSI DA RICEVENTE
Brook, Biologia dei
microrganismi – 1
Microbiologia generale
37
LA CONIUGAZIONE BATTERICA RICHIEDE CONTATTO DIRETTO TRA CELLULA
DONATRICE CHE POSSIEDE IL PLASMIDE CONIUGATIVO E CELLULA RICEVENTE CHE
NON LO POSSIEDE.
IL PLASMIDE F (FERTILITÀ, 99159 BP) DI ESCHERICHIA COLI POSSIEDE:
- I GENI PER LA REPLICAZIONE DEL DNA
- ALCUNI ELEMENTI TRASPONIBILI CAPACI DI INTEGRARSI NEL CROMOSOMA
DELL’OSPITE
- LA REGIONE tra (TRA) RESPONSABILE DEL TRASFERIMENTO
CELLULE F+ -> PLASMIDE F NON INTEGRATO NEL CROMOSOMA
CELLULE HFR * -> PLASMIDE F INTEGRATO NEL CROMOSOMA
* HIGH FREQUENCY OF RECOMBINATION
LA PRESENZA DEL PLASMIDE F INDUCE:
- LA SINTESI DEL PILO F
- LA MOBILIZZAZIONE DEL DNA PER TRASFERIRLO AL RICEVENTE
- L’INCAPACITA DELLA CELLULA DI COMPORTARSI DA RICEVENTE
Brook, Biologia dei
microrganismi – 1
Microbiologia generale
39
Brook, Biologia dei
microrganismi – 1
Microbiologia generale
40
Brook, Biologia dei
microrganismi – 1
Microbiologia generale
41
MUTAZIONI
cambiamento del materiale ereditario di tipo casuale, improvviso, raro,
che spesso si manifesta a livello di fenotipo
- a
livello di DNA
- CASUALE
--> può interessare un punto qualsiasi del genoma (su uno o più cromosomi di funghi e
lieviti o sull’unico cromosoma dei batteri)
- RARO
--> bassa frequenza del fenomeno (1 mutante su 106-108 cellule)
- può essere trasmessa, a meno che non sia letale
- SPONTANEE e INDOTTE (mezzi fisici, chimici e biologici)
GENOMICHE
CROMOSOMICHE GENICHE O PUNTIFORMI
interessano l’intero genoma nel suo complesso
interessano un solo cromosoma
interessano un solo gene
GENETICA DEI MICRORGANISMI EUCARIOTICI
#genoma costituito da più cromosomi
#molecole di DNA lineari
#fase aploide (n ) e fase diploide (2n)
#meiosi
- due copie di ciascun gene
- allele
2n -->n
- prima divisione meiotica (segregazione in 2 cellule separate)
- seconda divisione meiotica (simile alla divisione mitotica)
- prodotto sono 4 gameti aploidi
- fusione dei gameti ---> zigote (2n)
#omozigote --> omologhi geneticamente identici --> # eterozigote --> omologhi geneticamente diversi -->
GENETICA DEI LIEVITI
# S. cerevisiae: genetica molecolare EUCARIOTA maggiormente conosciuta
# organismo unicellulare in cui ciascuna cellula si può comportare come un gamete
# tipo sessuale α e a
# formazione di spore (asco e ascospore in S. cerevisiae)
# 16 cromosomi (245-2200 kb)
# modificazione del tipo sessuale in cellule aplodi ---> diploidi da coltura pura di 1
tipo sessuale
# plasmidi
CICLO BIOLOGICO DI SACCHAROMYCES CEREVISIAE
! Fase aploide (n) e diploide (2n)
! Tipi sessuali a e α
! Ceppi omotallici
- la maggior parte dei Saccharomyces vinari
- SI inversione sessuale e autodiploidizzazione
- maggior parte del ciclo vitale in fase diploide (2n)
! Ceppi eterotallici
- ceppi di laboratorio
- NO inversione sessuale e autodiploidizzazione
- maggior parte del ciclo vitale in fase aploide (n)
Brook, Biologia dei
microrganismi – 1
Microbiologia generale
46
IL GENOMA DI S.CEREVISIAE
Lunghezza totale
DNA ribosomale
Elementi trasponibili
14.5 milioni di basi
1.1 milioni di basi
0.15 milioni di basi
Numero di cromosomi
Lunghezza
16
220 000-1 600 000 basi
Fasi aperte di lettura
(ORF> lOOaa)
densità
numero totale
trasponibili)
Geni con funzione ignota
una fase (gene) ogni 1800 basi
6800 geni (escluso DNA ribosomale e elementi
circa 2000
(da Goffeau e Vassarotti, 1994)
MILIORAMENTO GENETICO LIEVITI
S. cerevisiae, lievito omotallico (i ceppi di laboratorio possono essere eterotallici, non
vanno incontro al fenomeno di inversione sessuale e autodiploidizzazione) le cui
cellule rimangono allo stato diploide finché si moltiplicano e solo quando le
condizioni lo consentono sporificano trasformandosi in aschi:
a seguito del processo meiotico si formano 4 gameti
corrispondenti a 4 spore aploidi
le spore possono:
I) germinare e dare origine a colture denominate “da singole spore”; le
prime cellule che derivano dalla germinazione sono aploidi ma, per il fenomeno di
inversione sessuale possono fondere tra loro per formare cellule diploidi
(autodiploidizzazione); le colture da singole spore sono pertanto diploidi e omozigoti
perché derivano dalla fusione di due cellule uguali
II) comportarsi da gameti e coniugare tra di loro: la coniugazione di spore
provenienti da ceppi differenti origina ibridi eterozigoti
la costruzione di nuovi ceppi può avvenire mediante:
• miglioramento genetico utilizzando i metodi della genetica classica (ibridazione
intra e interspecifiche)
• fusione tra sferoplasti
• ingegneria genetica







![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)

![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)


