La struttura quaternaria
Considerazioni sulla struttura QUATERNARIA
•
•
•
•
•
•
•
•
•
•
Quasi tutte le proteine a massa > 100 kDa sono oligomeriche
La struttura quaternaria realizza economia a livello genico
Consente di rimpiazzare facilmente subunita’ danneggiate
Il folding di piccole catene polipeptidiche e’ meno delicato del folding di una grossa proteina
singola-catena
Attraverso la struttura quaternaria si possono creare siti attivi piu’ sofisticati e stabili dal punto di
vista strutturale (es. per il riconoscimento di grossi substrati)
Attraverso la struttura quaternaria si creano piu’ siti attivi nello stesso enzima
Attraverso la struttura quaternaria si puo’ produrre allosteria, coopertaivita’, controllo della attivita’
Le interfacce di aggergazione mimano il core della proteina a livelli differenti, a seconda della
necessita’ di raggiungere oligomeri particolarmente stabili, oppure facilmente dissociabili
Un arrangiamento oligomerico simmetrico implica che l’interfaccia di associazione in ogni
protomero esaurisca le proprie potenzialita’ associative al momento della sua associazione
all’oligomero.
OLIGOMERI ‘CHIUSI’ Simmetria ‘PUNTIFORME’
La struttura quaternaria
La superficie proteica si e’ evoluta per NON aggregarsi spontaneamente
ad altre proteine in maniera aspecifica
L’ultimo livello nella gerarchia strutturale delle proteine è rappresentato dalla
struttura quaternaria.
Struttura
supersecondaria
Domini
La struttura quaternaria
Le proteine che sono costituite da una sola catena polipeptidica sono chiamate
monomeriche.
Esiste un consistente numero di proteine costituite da un certo numero di
catene polipeptidiche identiche, chiamate subunità (o protomeri), che si
associano in modo specifico a formare una molecola multimerica. Si dice che
queste proteine hanno una struttura quaternaria. Le zone di contatto fra le
subunità sono costituite da catene laterali non polari e caratterizzate da
interazioni di van der Waals, legami idrogeno e talvolta ponti disolfuro
intercatena.
Le subunità possono funzionare in modo indipendente una dall’altra oppure in
modo cooperativo, così che la funzione di una subunità sia dipendente dallo
stato funzionale delle altre subunità.
Altre proteine sono costituite da catene polipeptidiche, e quindi da subunità,
diverse, ciascuna con una diversa funzione.
La struttura quaternaria
Sono svariate le ragioni per cui le proteine costituite da diverse subunità
(invece che da un’unica enorme catena polipeptidica) sono così comuni.
In particolare:
- eventuali difetti possono essere
riparati sostituendo la subunità
difettosa.
- Il sito di formazione della subunità
può essere diverso da quello di
assemblaggio della struttura finale e la
sola informazione genetica necessaria
è specificare le diverse subunità che si
devono assemblare.
La struttura quaternaria
- nel caso degli enzimi, aumentarne le
dimensioni attraverso l’associazione
di subunità identiche è più efficiente
che aumentare la lunghezza della
catena polipeptidica, poiché ogni
subunità ha un sito attivo.
Inoltre, la costituzione a subunità
fornisce le basi strutturali per la
regolazione della loro attività.
Le interfacce di aggregazione mimano il core della proteina a livelli differenti, a seconda della
necessita’ di raggiungere oligomeri particolarmente stabili, oppure facilmente dissociabili.
Un arrangiamento oligomerico simmetrico implica che l’interfaccia di associazione in ogni
protomero esaurisca le proprie potenzialita’ associative al momento della sua associazione
all’oligomero.
OLIGOMERI ‘CHIUSI’ Simmetria ‘PUNTIFORME’
protomeri.
1000-2000 Å2 di interfaccia tra
La struttura quaternaria
Nel formare i multimeri, le subunità si dispongono in modo simmetrico, cioè
occupano posizioni geometricamente equivalenti, secondo le seguenti
operazioni di simmetria rotazionale:
- simmetria ciclica, in cui le subunità
sono correlate da un singolo asse di
rotazione;
- simmetria diedra, caratterizzata dalla
composizione di un asse di rotazione di
ordine n con un asse di rotazione di
ordine
2
(che
si
intersecano
perpendicolarmente);
- altre simmetrie possibili sono quella
tetraedrica, ottaedrica e icosaedrica.
La struttura quaternaria
Alcune proteine multimeriche presentano simmetria elicoidale.
Le subunità chimicamente identiche dell’elica non sono strettamente
equivalenti, perché, per esempio, quelle alle estremità dell’elica hanno un
environment diverso rispetto a quelle nel mezzo.
Tuttavia l’intorno di tutte le subunità
nell’elica, tranne quelle vicino alle
estremità, sono sufficientemente simili,
per cui le subunità sono dette quasiequivalenti.
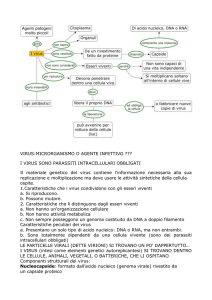
I virus
I virus
I virus sono parassiti costituiti da molecole di acido nucleico (DNA o RNA, a
doppia o singola elica) circondate da un rivestimento protettivo costituito da
proteine, chiamato capside. Alcuni virus hanno anche una membrana a doppio
strato lipidico, chiamato pericapside
o envelope, che riveste il capside.
Un acido nucleico non potrebbe mai
codificare una singola molecola
proteica tanto grande da racchiuderlo.
Il capside dei virus, quindi, è
costituito da innumerevoli copie di
una o alcune catene polipeptidiche.
I virus hanno forme e dimensioni
diverse: in particolare, il capside può
essere sferico o a bastoncello.
I virus
La conoscenza in dettaglio (struttura tridimensionale a livello atomico) del
capside dei virus potrebbe essere importante per progettare farmaci antivirali.
Per esempio, l’entrata dei virus nelle cellule dei mammiferi dipende dal
riconoscimento specifico, da parte del capside virale, di proteine o carboidrati
(recettori) che si trovano sulla superficie delle cellule bersaglio. Questa
interazione gioca un ruolo cruciale e composti che interferiscono con questa
interazione dovrebbero bloccare l’infezione.
I siti di legame virale sulla superficie delle cellule ospite sono anche i target
degli anticorpi, per cui il sistema immunitario dell’ospite ha con tutta
probabilità giocato un ruolo importante nell’evoluzione dei virus animali. La
conoscenza, quindi, delle regioni di superficie di questi virus dovrebbe aiutare
a capire meglio gli adattamenti strutturali che i virus hanno evoluto per eludere
le difese dell’ospite.
I virus
L’esistenza di tutti i virus dipende dalla loro
abilità di infettare le cellule, inducendole a
generare altre particelle di virus.
Le cellule infette generalmente muoiono
durante questo processo.
Il ciclo di infezione dei virus si può così
schematizzare:
- i virus rilasciano il loro acido nucleico
(ss/ds-DNA, ss/ds-RNA) nella cellula ospite,
spesso insieme a proteine virali;
I virus
- la macchina biosintetica della cellula ospite
viene utilizzata per la replicazione,
trascrizione e traduzione dei geni virali, a
spese dell’espressione dei geni cellulari;
- le nuove particelle di virus generate lasciano
la cellula infettata per infettare nuove cellule.
I virus
Immagini al
microscopio elettronico
I virus
Le proteine che costituiscono il capside dei virus si dispongono in modo
simmetrico o quasi-simmetrico.
Sono note due possibili disposizioni (assemblaggi di natura quaternaria,
quindi):
- virus con capside elicoidale, in cui le subunità proteiche si associano
a formare un bastoncello elicoidale;
- virus con capside sferico, in cui le subunità proteiche si aggregano a formare
un poliedro.
Considerato l’elevato numero di subunità proteiche (fino ad alcune centinaia)
coinvolte nella formazione del capside dei virus, essi rappresentano
l’esempio più complesso di organizzazione delle proteine in
struttura quaternaria.
Virus con capside elicoidale
Un esempio di virus con capside elicoidale è dato dal virus del mosaico del
tabacco (TMV).
TMV ha una forma a bastoncello, è lungo circa 3000
180 Å e ha una massa di 40 milioni di dalton.
Il suo capside è costituito da circa 2130 copie
identiche di una proteina (158 aminoacidi),
disposte a formare un’elica destrorsa cava
(diametro cavità circa 40 Å), caratterizzata da
n = 16.3 subunità per giro, p = 23 Å.
TMV contiene una molecola di RNA a singola
elica che si avvolge nella cavità interna seguendo i
giri dell’elica di proteine, in modo tale che 3
nucleotidi interagiscano con una subunità proteica.
Å, ha un diametro di
Virus con capside elicoidale
Lo stato di aggregazione delle proteine del capside di TMV dipende dal pH e
dalla forza ionica.
- pH basico, bassa forza ionica:
le proteine formano complessi di solo
alcune subunità;
- pH basico, alta forza ionica:
le subunità si associano a formare
dischi a doppio strato (17 subunità per
disco);
Virus con capside elicoidale
- pH neutro, bassa forza ionica:
le subunità formano corte eliche di
poco più di 2 giri, chiamate
protoeliche;
- pH acido, bassa forza ionica:
le protoeliche si impilano in modo
imperfetto e alla fine si fondono a
formare
un lungo
bastoncello
elicoidale.
Virus con capside elicoidale
Immagini al microscopio
elettronico per seguire
l’andamento dell’aggregazione
delle proteine del capside di
TMV, passando da pH = 7 a
pH = 5, in condizioni di bassa
forza ionica.
Virus con capside elicoidale
L’analisi cristallografica a raggi X ha mostrato che ciascuna subunità del
capside del virus MTV è costituita da un bundle di 4 eliche, disposto in modo
radiale rispetto all’asse dell’elica del virus.
asse elica virus
Structure of the TMV protein disk in cross
section showing its polypeptide chains as ribbon
diagrams.
Assembly of TMV. (a) The process begins by the insertion
of the hairpin loop formed by the initiation sequence of
the viral RNA into the protohelix’s central cavity.
Assembly of TMV. (b) The RNA then intercalates
between the layers of the protohelix, thereby
ordering the disordered loop and trapping the
RNA.
Page 1378
Assembly of TMV. (c) Elongation proceeds by the
stepwise addition of protohelices to the “top” of the
viral rod.
Virus con capside sferico
Un altro modo in cui subunità equivalenti si dispongono a costituire il capside
dei virus è quello di formare un poliedro sferico, secondo due principi base:
- specificità: le subunità devono riconoscersi con precisione per formare la
giusta interfaccia di interazioni non-covalenti, visto che i virus si assemblano
spontaneamente dai loro componenti individuali;
- economia genetica: il capside è costituito da molte copie di alcuni tipi di
subunità.
Questi due principi implicano la simmetria: pattern di legame ripetuti e
specifici di unità costituenti identiche portano ad una struttura finale
simmetrica.
Virus con capside sferico
Come disporre in modo simmetrico oggetti identici in modo tale da costruire
una sfera?
Esiste solo un numero limitato di modi per farlo e fra questi la simmetria
icosaedrica è la più alta possibile, permettendo al maggior numero di oggetti
identici di formare una sfera.
Lo studio del capside dei virus sferici con il microscopio elettronico e con la
cristallografia a raggi X ha indicato che la simmetria icosaedrica sta alla base
della loro architettura.
Virus con capside sferico
L’icosaedro è un oggetto approssimativamente sferico costituito da 20 triangoli
equilateri identici. La sua simmetria è descritta da tipi diversi di rotazioni, che
lo portano a
coincidere con
se stesso.
12 vertici
(asse di rotazione
di ordine 5)
20 facce
(asse di rotazione
di ordine 3)
30 spigoli
(asse di rotazione
di ordine 2)
Kin exercises/Viral-coat
Virus con capside sferico
L’icosaedro presenta 12 5 = 60 (o 20 3, o 30 2) posizioni equivalenti.
Ogni oggetto che presenta simmetria è costituito da parti più piccole, identiche
e messe in relazione una con l’altra da operazioni di simmetria; tali parti
vengono chiamate “unità connesse da simmetria” o “unità asimmetriche”.
Le subunità proteiche che costituiscono il capside dei virus sferici sono unità
asimmetriche: il numero minimo di subunità proteiche necessarie a formare il
capside con simmetria icosaedrica è quindi uguale alle posizioni equivalenti
dell’icosaedro.
Le proprietà di simmetria dell’icosaedro non si limitano alla superficie, ma si
estendono all’intero volume. Un’unità asimmetrica è quindi una parte di questo
volume: è un cuneo che parte dalla superficie, fino al centro dell’icosaedro.
Virus con capside sferico
L’icosaedro ha 60 unità asimmetriche, per cui il numero minimo di subunità
proteiche richiesto per formare il capside di un virus a simmetria icosaedrica è
60.
Ciascuna unità asimmetrica può contenere una o più catene polipeptidiche.
X-Ray structure of satellite tobacco mosaic virus
(STMV). The virion is shown in cutaway view along
its icosahedral (T-1) 5-fold axis.
From: Biochemistry – Voet & Voet (2004) J. Wiley and Sons Inc.
Virus con capside sferico
In un capside sferico costituito solo da 60 subunità proteiche ben poco spazio è
lasciato alla molecola di DNA/RNA.
I virus autosufficienti hanno genomi molto lunghi, che codificano per enzimi
necessari alla replicazione del loro acido nucleico virale, oltre che per le
proteine strutturali del capside. Questi genomi necessitano ampi spazi
all’interno del capside e quindi un capside più grande.
Come si può costruire un capside più grande mantenendo la simmetria
icosaedrica?
L’aumento delle dimensioni di ciascuna subunità porterebbe ad un piccolo
guadagno nelle dimensioni del capside ma ad un notevole suo ispessimento.
Il solo modo possibile è aumentare il numero delle subunità, cioè ciascuna
unità asimmetrica dell’icosaedro deve contenere più di una subunità (che
possono essere identiche o diverse).
Virus con capside sferico
Il numero totale di subunità costituenti il capside deve essere un multiplo intero
di 60.
Donald Caspar e Aaron Klug nel 1962 hanno dimostrato che solo alcuni
multipli interi (1, 3, 4, 7, …) di 60 (subunita’ proteiche) sono possibili per
mantenere la specificità delle interazioni all’interno della simmetria
icosaedrica, in modo tale che i contatti proteina-proteina siano simili.
Questi multipli interi sono chiamati numeri di triangolazione T.
I valori di T permessi sono dati dalla seguente formula:
T = h2 + hk + k2
[h, k interi positivi]
Nel caso di T = 1 le subunità proteiche si impaccano a formare l’icosaedro in
modo strettamente equivalente.
Nel caso di T 1 le subunità si possono impaccare con pattern di legame solo
leggermente diversi, in modo quasi equivalente.
Virus con capside sferico: T = 1
I virus satellite, che sono non autosufficienti, poiché non codificano da soli tutte
le funzioni richieste per la loro replicazione, hanno il capside sferico costituito
soltanto da 60 catene proteiche (numero di triangolazione T = 1).
Un esempio è il virus satellite della necrosi del tabacco, che è anche uno dei
più piccoli finora conosciuti. Esso ha un diametro di 180 Å e per replicarsi ha
bisogno dell’aiuto del virus della necrosi del tabacco.
Il suo RNA codifica
per una sola catena
polipeptidica
(195 aminoacidi)
per unità
asimmetrica
dell’icosaedro.
Virus con capside sferico: T = 3
Nel caso di T = 3 (180 subunità), ciascuna unità asimmetrica contiene 3
subunità proteiche (A, B e C) con un diverso environment.
Le subunità A interagiscono intorno all’asse di rotazione di ordine 5, mentre le
subunità B e C si alternano intorno all’asse di rotazione di ordine 3.
Ci sono, quindi, 6 subunità (3 B e 3 C)
disposte in modo pseudo-simmetrico intorno
all’asse di rotazione di ordine 3, che diventa
quindi anche un asse di pseudo-simmetria di
ordine 6.
La disposizione delle subunità intorno
all’asse di simmetria di ordine 5 e a quello di
pseudo-simmetria di ordine 6 dovrebbe
essere molto simile, con piccole alterazioni
delle modalità di impaccamento.
T=3
Virus con capside sferico: T = 3
Un esempio di capside di virus sferico con T = 3 è dato dal virus di una
malattia del pomodoro (tomato bushy stunt virus). Questo virus contiene 180
catene polipeptidiche chimicamente identiche (386 aminoacidi) per un
diametro totale di 330 Å.
Ciascuna subunità è costituita da un dominio R, una regione di loop (a) che
connette R con il dominio S,
a formare il guscio virale,
A
e da un dominio P che si
B
C
proietta fuori dalla superficie.
I domini P ed S sono collegati
da una cerniera (h).
Le catene polipeptidiche
identiche hanno diverse
strutture tridimensionali
quando formano le 3
subunità A, B e C.
Radial organization of TBSV indicating the
distribution of its protein and RNA
components.
From: Biochemistry – Voet & Voet (2004) J. Wiley and Sons Inc.
Page 1381
X-Ray structure of the tomato bushy stunt
virus (TBSV) coat protein subunit.
From: Biochemistry – Voet & Voet (2004) J. Wiley and Sons Inc.
Virus con capside sferico
Il fatto che i virus sferici vegetali e alcuni di quelli animali abbiano lo stesso
modo di disporre le subunità proteiche per costruire l’icosaedro, fa ipotizzare
che essi siano correlati dal punto di vista evoluzionistico e abbiano un ancestore
comune.
Questa ipotesi è supportata dall’analisi del fold delle subunità proteiche
costituenti il capside.
L’analisi cristallografica
ai raggi X ha mostrato
che tutte le proteine
costituenti il capside
dei virus sferici
assumono la struttura
-antiparallela con
topologia jelly roll.
Virus con
capside
sferico
In generale si osserva
che i loop ad una
estremità del barile
sono sempre corti,
mentre quelli
all’estremità opposta
sono molto lunghi e di
dimensioni variabili.
Tali strutture
proteiche si possono
schematizzare con un
cuneo.
Virus satellite
della necrosi del tabacco
Subunità VP2 virus
raffreddore
Subunità VP1 virus
polio
Virus con capside sferico
Un’eccezione alla regola è rappresentata dal fold delle proteine che
costituiscono il capside del batteriofago MS2 (T = 3).
In questo caso le proteine hanno la struttura
di un foglietto antiparallelo con topologia
up and down.
Virus con capside sferico
Molti farmaci sono efficaci perché si legano a siti recettoriali specifici,
bloccando la funzione fisiologica della proteina.
Studi di progettazione di farmaci sono stati condotti sul virus del raffreddore.
Nel capside del virus del raffreddore, come in tutti i picornavirus, ci sono
zone di canyon (profonde 25 Å e larghe da 12 a 30 Å) che circondano le
protuberanze, formate dalle proteine VP1.
Virus con capside sferico
I canyon formano il sito di legame per
il recettore del virus dell’influenza
sulla superficie della cellula ospite.
In particolare, il composto antivirale
si lega a VP1, inserendosi in una
cavità idrofobica sotto il pavimento
del canyon.
Virus con capside sferico: T = 4
Nel caso di T = 4 (240 subunità), ciascuna unità asimmetrica contiene 4
subunità proteiche (A, B, C e D) con un diverso environment.
Le subunità A interagiscono intorno
all’asse di rotazione di ordine 5,
le subunità D intorno all’asse di
rotazione di ordine 3,
mentre le subunità B e C sono
disposte in modo tale che 2 copie di
ciascuna
interagiscano
intorno
all’asse di ordine 2, oltre a 2
subunità D.
T=4
From: Biochemistry – Voet & Voet (2004) J. Wiley and Sons Inc.
Structure of the PBCV-1 capsid. A quasi-atomic
model based on fitting the X-ray structure of Vp54
to the cryo-EM–based image of the capsid.