Capitolo 19
Risposte a domande interne al capitolo
19.1 (p. 595)
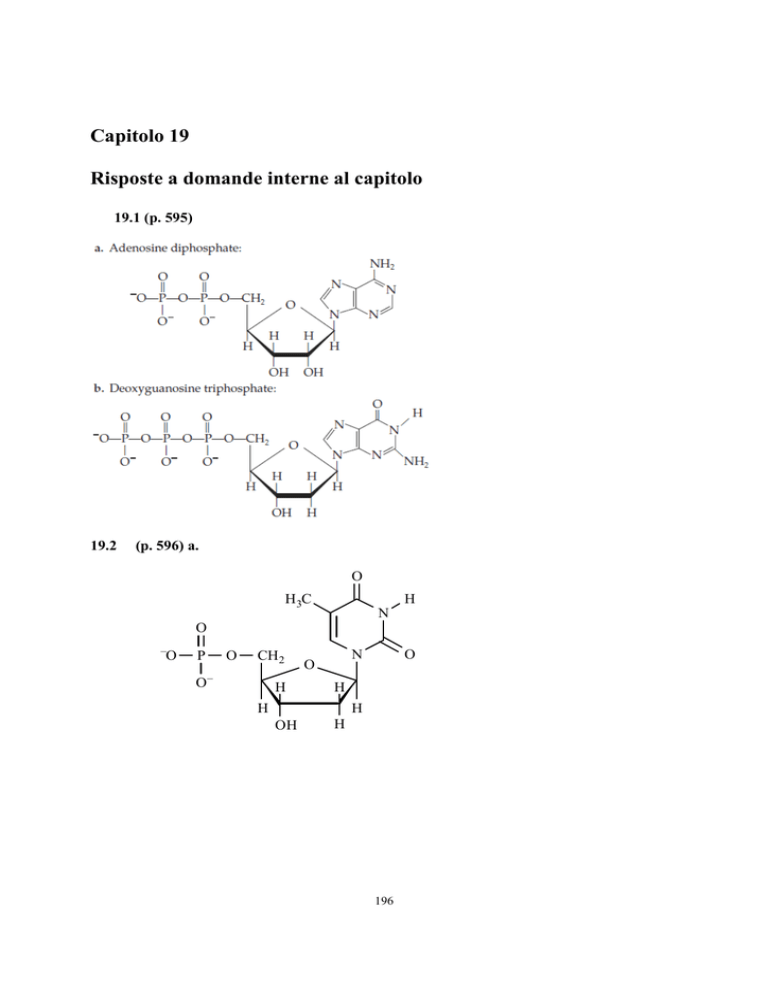
19.2
(p. 596) a.
O
H3C
H
N
O
–
O
P
O
CH2
O–
H
N
O
O
H
H
H
OH
H
196
b.
NH2
N
O
–
O
O
P
O
O–
P
O
O
O–
P
O
CH2
O–
N
O
H
H
OH
H
OH
H
O
c.
O
H
N
O
–
O
P
O
CH2
O–
N
O
H
H
OH
H
OH
H
O
19.3 (p. 610) L’RNA polimerasi riconosce il sito promotore per un gene, separa i filamenti di
DNA e catalizza la polimerizzazione di un filamento di RNA complementare al
filamento di DNA che porta il codice genetico di una proteina. Riconosce un sito
di terminazione alla fine del gene e rilascia la molecola di RNA.
19.4 (p. 610) La sequenza del promotore di un gene, riconosciuta dalla RNA polimerasi,
segnala il sito di inizio e la direzione di trascrizione.
19.5 (p. 613) Il codice genetico si dice degenerato perché diverse triplette di codoni possono
codificare per un singolo aminoacido.
197
19.6 (p. 613) Il codice genetico è detto resistente alla mutazione perché un cambiamento nella
terza posizione di molti codoni non cambia l’identità dell’aminoacido codificato.
19.7 (p. 618) Le basi azotate dei codoni sono complementari a quelle degli anticodoni. Come
risultato, essi sono in grado di legarsi tramite ponti idrogeno secondo le regole
dell’accoppiamento delle basi.
19.8 (p. 618) Se la sequenza di un codone dell’mRNA è 5’-AUG-3 ‘, la sequenza
dell’anticodone sarebbe 5’-CAU-3’.
19.9 (p. 621) Il sito P ribosomiale contiene il peptidil tRNA durante la sintesi proteica. Il
peptidil tRNA è il tRNA che porta la catena peptidica in crescita. L’unica
eccezione si ha durante l’inizio della traduzione quando il sito-P lega il tRNA
iniziatore.
19.10 (p. 621) Il sito A ribosomiale è la tasca sulla subunità minore ribosomiale che ospita
l’aminoacil tRNA durante la fase di allungamento della traduzione. La catena
peptidica crescente viene trasferita dal peptidil tRNA nel sito P all’aminoacido
attaccato all’amminoacil tRNA nel sito A.
19.11 (p. 623) La normale sequenza di mRNA, AUG-CCC-GAC-UUU, codifica la sequenza
peptidica metionina-prolina-aspartato-fenilalanina. La sequenza di mRNA
mutante,
AUG-CGC-GAC-UUU, codifica la
sequenza
mutante
peptide
metionina-arginina-aspartato-fenilalanina. Questa non è una mutazione silente
poiché un aminoacido idrofobo (prolina) è stato sostituito da un amminoacido a
carica positiva (arginina).
19.12 (p. 623) La sequenza di mRNA AUG-CCC-GAC-UUU codifica per il tetrapeptide
metionina -prolina-aspartato-fenilalanina. La sequenza di mRNA mutata AUG-
198
CCG-GAC-UUU codifica per lo stesso tetrapeptide. Questa è una mutazione
silente. La mutazione nella sequenza nucleotidica del gene è nella terza posizione
del codone. In questo caso non si altera la sequenza di aminoacidi codificati dal
mRNA.
Risposte a Domande ed Esercizi di fine capitolo
19.1
Un’ammina eterociclica è un composto che contiene un azoto in almeno una posizione
dello scheletro dell’anello.
19.2
Le purine e pirimidine, che sono basi azotate, sono ammine eterocicliche.
19.3
L’atomo N-9 della purina forma il legame N-glicosidico con il C-1 dei cinque atomi di
carbonio dello zucchero. La struttura generale dell’anello purinico viene mostrata di
seguito:
19.4
a. La struttura ad anello di una pirimidina:
4
3
5
N
2
N
6
1
b. L’azoto in posizione 1 forma il legame N-glicosidico con lo zucchero a 5 atomi di
199
carbonio.
19.5
I due filamenti del DNA a doppia elica sono detti antiparalleli perché corrono in
direzioni opposte. Un filamento progredisce nella direzione 5 ′ → 3 ′, e il filamento
opposto progredisce nella direzione 3 ′ → 5 ′.
19.6
Il DNA è composto dallo zucchero 2’-desossiribosio, le purine adenina e guanina, le
pirimidine citosina e timina. È generalmente una molecola elicoidale a doppio
filamento. L’RNA è composto dallo zucchero ribosio, le purine adenina e guanina,le
pirimidine citosina e uracile. L’RNA è generalmente a singolo filamento.
19.7
La doppia elica del DNA è larga 2 nm. Le basi azotate sono impilate a una distanza di
0,34 nm l’una dall’altra. Un giro completo della spirale è di 3,4 nm o 10 paia di basi.
19.8
19.9
Tre legami idrogeno sono formati tra la citosina e la guanina.
200
H
H
Cytosine
N
O
Guanine
N
N
H
N
N
H
H
O
H
O
N
O
CH2
N
N
H
H2
C
H
O
O
H
H
O
H
H
H
O
H
H
19.10 Il cromosoma procariotico è una molecola di DNA circolare superavvolto (l’elica è
avvolta su se stessa).
19.11 Il cromosoma degli eucarioti ha più livelli di struttura. Il primo livello è il nucleosoma,
che consiste di un filamento di DNA avvolto attorno a un piccolo disco di istoni. Questa
stringa di perle viene poi avvolta in una struttura più ampia, chiamata fibra, a 30 nm,
avvolte in fibre 200 nm. Ci potrebbero essere molti livelli di struttura che non sono
ancora ben compresi.
19.12 Il termine replicazione semiconservativa del DNA si riferisce al fatto che ogni filamento
di DNA parentale funge da stampo per la sintesi di un filamento figlio. Come risultato,
ciascuna delle molecole di DNA figlio è costituita da un filamento del DNA parentale
originale e da un filamento di DNA di nuova sintesi.
19.13 Nella replicazione del DNA semiconservativa, ogni molecola di DNA figlia consiste di
201
un filamento di DNA parentale e di un filamento figlio neo sintetizzato:
5’AGCTAAAGGGCCTTCTCGACTA 3’
3’TCGATTTCCCGGAAGAGCTGAT 5’
↓
5’AGCTAAAGGGCCTTCTCGACTA 3’
3’TCGATTTCCCGGAAGAGCTGAT 5’
+
5’AGCTAAAGGGCCTTCTCGACTA 3’
3’TCGATTTCCCGGAAGAGCTGAT 5’
19.14 La DNA polimerasi III deve leggere un filamento di DNA stampo e catalizzare la
polimerizzazione di un filamento nuovo, ma deve anche rileggere il filamento
neosintetizzato per correggere eventuali errori, rimuovendo il nucleotide non correttamente
inserito e aggiungendo quello giusto.
19.15 a. La DNA polimerasi è detta modello-diretta, perché è governata dalle regole di
accoppiamento delle basi, cioè adenine con timina e guanina con citosina. In questo modo
la DNA polimerasi legge il filamento parentale, il modello, e catalizza la polimerizzazione
di un filamento figlio di DNA complementare.
b. La replicazione del DNA è un processo di auto-correzione a causa della capacità di
correzione della DNA polimerasi III. Durante l’allungamento della catena, la DNA
polimerasi III rileva gli errori sul filamento di DNA che è stato appena sintetizzato. I
nucleotidi errati vengono rimossi e sostituiti con quelli corretti, complementari al filamento
202
parentale.
19.16 3′-TACGGGCTCGACTAACTAGTCT-5′
19.17 L’origine di replicazione di una molecola di DNA è una sequenza unica sulla molecola di
DNA in cui inizia la replicazione.
19.18 L’enzima elicasi separa i filamenti di DNA all’origine di replicazione, in modo che le
proteine coinvolte nella replicazione possano interagire con le coppie di basi azotate.
19.19 Poiché i filamenti di DNA sono antiparalleli e a causa della necessità di un primer di
RNA per la replicazione del DNA, i meccanismi per la replica dei due filamenti di una
molecola di DNA sono diversi. Un filamento, chiamato filamento principale, viene
replicato in continuazione. Il filamento opposto, chiamato filamento ritardato, viene
replicato in maniera discontinua. Per il filamento continuo, un singolo primer di RNA
viene prodotto all’origine di replicazione e la DNA polimerasi III catalizza
continuamente l’aggiunta di nucleotidi nella direzione 5 ‘- 3’, iniziando con l’aggiunta
del primo nucleotide al primer di RNA.
Sul filamento ritardato molti primer di RNA vengono prodotti man mano che la forcella
di replicazione avanza. La DNA polimerasi III catalizza la sintesi di DNA da ciascuno
dei singoli primers. Senza nessun altro processo, il risultato sarebbe una molecola
frammentata e con diversi frammenti di RNA inclusi nel filamento figlio. La tappa finale
della sintesi del DNA sul filamento ritardato include la rimozione dei primers, la
riparazione delle lacune lasciate dai primers stessi e l’unione dei diversi frammenti in un
unico filamento di DNA.
19.20 DNA→RNA→PROTEINE
203
19.21 La sequenza nucleotidica del DNA contiene il codice genetico per la produzione di
proteine e le sequenze nucleotidiche trascritte per produrre RNA transfer e RNA
ribosomiale. Queste informazioni vengono trasmesse da generazione in generazione dalla
replicazione del DNA. Tre specie di RNA sono prodotte dalla trascrizione del DNA e
interagiscono tra loro per tradurre il codice genetico in proteine. L’RNA messaggero
porta il codice per la proteina; l’RNA transfer decodifica il messaggio, mentre l’RNA
ribosomiale è un componente funzionale e strutturale del ribosoma, la piattaforma su cui
avviene la sintesi proteica. Le proteine effettuano la maggior parte del lavoro cellulare e
sono componenti strutturali dell’organismo.
19.22 Gli anticodoni si trovano sulle molecole di RNA transfer.
19.23 I codoni si trovano sul RNA messaggero.
19.24 3’-AUGCCCGUAUCCGGAAUUUCGAUCGAA-5’
19.25 Se un RNA avesse la sequenza nucleotidica 5’-AUGCCAUAACGAUACCCAGUC-3 ‘,
il DNA trascritto avrebbe la seguente sequenza:
3’-TACGGTATTGCTATGGGTCTG-5’
Questa sequenza di DNA potrebbe essere più propriamente scritta nella forma seguente:
5’-GTCTGGGTATCGTTATGGCAT-3’
19.26 Lo splicing dell’RNA è il processo che permette la rimozione delle sequenze non
codificanti (introni) del trascritto primario di un mRNA eucariotico e delle sequenze
204
codificanti proteine (esoni) mescolate tra loro.
19.27 RNA messaggero, RNA transfer e RNA ribosomiale.
19.28 L’RNA messaggero porta il codice genetico di una proteina. L’RNA transfer decodifica
l’informazione contenuta nel mRNA e porta l’aminoacido corretto al sito di sintesi
proteica. L’RNA ribosomiale è un componente strutturale e funzionale del ribosoma, la
piattaforma sulla quale vengono prodotte proteine.
19.29 Gli spliceosomi sono complessi ribonucleoproteici di piccole dimensioni che svolgono lo
splicing dell’RNA.
19.30 Le snRNP, o piccole ribonucleoproteine, sono i componenti dello spliceosoma. Le
porzioni di RNA delle snRNP sono complementari alle sequenze coinvolte nello splicing.
Attraverso legami idrogeno al confine delle zone di splicing o sequenze introniche, le
snRNP riconoscono e riuniscono le sequenze coinvolte nelle reazioni di splicing.
19.31 La coda di poli (A) è un tratto di 100-190 adenosine polimerizzate sull’estremità
terminale 3 ′ di un mRNA, da parte dell’enzima poli (A) polimerasi.
19.32 La coda di poli (A) protegge l’estremità 3’ del mRNA dalla degradazione da esonucleasi.
19.33 La struttura a cappuccio è composta dal nucleotide 7-metilguanosina legato all’estremità
5′ di un mRNA da un ponte trifosfato 5’-5′. Generalmente i primi due nucleotidi di
mRNA sono metilati.
19.34 I codoni che costituiscono il codice genetico sono 64.
19.35 Nel codice genetico ogni parola del codice consiste di tre nucleotidi.
19.36 Il quadro di lettura di un gene è l’insieme sequenziale delle triplette di codoni che portano
il codice genetico per la struttura primaria di una proteina.
19.37 Metionina e triptofano
205
19.38 Serina, arginina, e leucina sono codificate da sei codoni ciascuna.
19.39 I ribosomi svolgono la funzione di piattaforma per la sintesi proteica. Inoltre catalizzano
la formazione dei legami peptidici.
19.40 Il sito aminoacil tRNA (sito A) e il sito peptidil tRNA (sito P) sono i due siti di legame
per il tRNA sul ribosoma.
19.41 La sequenza di nucleotidi di DNA in un gene viene trascritta per produrre una sequenza
di nucleotidi di RNA complementari in un RNA messaggero (mRNA). Nel processo di
traduzione la sequenza di mRNA viene letta sequenzialmente in parole di tre nucleotidi
(codoni) per produrre una proteina. Ogni codone aggiunge un particolare aminoacido alla
catena peptidica in crescita. Se uno dei codoni è stato modificato mediante mutazione, si
può aggiungere l’aminoacido errato alla catena peptidica in crescita. Ciò può causare un
ripiegamento improprio della proteina e la perdita della funzione biologica.
19.42 Se il mRNA ha la seguente sequenza nucleotidica:
5’-AUG-UGU-AGU-GAC-CAA-CCG-AUU-UCA-CUG-UGA-3’
La proteina avrà la seguente sequenza amminoacidica:
met-cys-ser-asp-gln-pro-ile-ser-leu
L’ultimo codone, UGA, è un codone di stop.
19.43 Un aminoacido è legato a una milecola di tRNA in una molecola di amoacil-tRNA con
un legame estere.
206
19.44 Una mutazione puntiforme è la sostituzione di una coppia di nucleotidi con un’altra in un
gene.
19.45 Una delezione è una mutazione che provoca la perdita di uno o più nucleotidi da una
molecola di DNA. Una mutazione di inserimento comporta l’aggiunta di uno o più
nucleotidi ad una molecola di DNA
19.46 Alcune mutazioni sono silenti perché la variazione nella sequenza nucleotidica non altera
la sequenza aminoacidica della proteina. Questo può succedere perché ci sono aminoacidi
codificati da più codoni.
19.47 Una mutazione puntiforme è più probabile che sia una mutazione silente perché
influenzerà solo un singolo codone. In molti casi, una mutazione puntiforme in un
codone, in particolare se è nella terza posizione, non modifica l’aminoacido incorporato
nella proteina. D’altra parte, una delezione di uno o due nucleotidi cambierà il quadro di
lettura del gene, il che modificherà la sequenza aminoacidica codificata da tale gene da
quel punto alla fine della proteina. Sebbene la delezione di tre nucleotidi non cambi il
quadro di lettura, il risultato sarà la perdita di un aminoacido dal peptide.
19.48 La luce UV provoca la formazione di dimeri di pirimidina, il legame covalente di due
basi pirimidiniche adiacenti. Le mutazioni si verificano quando il sistema di riparazione
del danno UV commette un errore. Questo provoca una variazione nella sequenza
nucleotidica del DNA.
19.49 Un cancerogeno è un composto che causa il cancro. I tumori sono causati da mutazioni
nei geni responsabili del controllo della divisione cellulare. I cancerogeni causano danni
al DNA che determinano variazioni nella sequenza nucleotidica del gene, pertanto sono
anche mutageni.
207
19.50 Un enzima di restrizione è un enzima batterico che “taglia” lo scheletro zucchero-fosfato
del DNA a una sequenza nucleotidica specifica.
19.51 Un enzima di restrizione è un enzima che taglia il DNA in una specifica sequenza
bersaglio. Gli enzimi di restrizione sono usati per tagliare il DNA bersaglio e il DNA
vettore, lasciando entrambi con lo stesso tipo di “estremità appiccicose”. Le “estremità
appiccicose” di DNA bersaglio e vettore possono ibridizzare. Il DNA bersaglio e vettore
sono poi uniti covalentemente utilizzando l’enzima DNA ligasi.
19.52 Un marker selezionabile è un tratto genetico che può essere utilizzato per rilevare la
presenza di un plasmide in un batterio. Molti plasmidi hanno come marker selezionabili
geni resistenti agli antibiotici. I batteri contenenti il plasmide saranno in grado di crescere
in presenza dell’antibiotico, quelli senza il plasmide moriranno.
19.53 Un vettore di clonazione è una molecola di DNA auto-replicante (plasmide o fago) in cui
il DNA estraneo può essere clonato. Una volta all’interno della cellula, il vettore e il
frammento di DNA clonato sarà replicato per produrre un gran numero di copie del DNA
clonato.
19.54 Insulina umana, interferone, ormone della crescita umana e il fattore VIII umano della
coagulazione del sangue
19.55 L’obiettivo finale dell’ingegneria genetica potrebbe essere la sostituzione di un gene
mutante con una copia normale, e quindi l’eliminazione della malattia genetica. Alcuni
temono che l’ingegneria genetica possa essere utilizzata per cercare di produrre una
popolazione di esseri umani con specifiche caratteristiche di intelligenza e aspetto fisico.
19.56 Dopo dodici cicli di reazione a catena della polimerasi, si avranno 4.096 copie di DNA
bersaglio.
208
19.57 Il Progetto Genoma Umano identificava e mappava tutti i geni del genoma umano e
determinava le sequenze di DNA complete dei tre miliardi di coppie di nucleotidi.
19.58 I benefici potenziali delle informazioni acquisite nel Progetto Genoma Umano includono
una migliore comprensione, il trattamento e la prevenzione di alcune malattie genetiche.
19.59 Una libreria genomica è un insieme di cloni che rappresentano tutte le sequenze di DNA
nel genoma di un organismo.
19.60 Chromosome walking è una tecnica per l’analisi del genoma che richiede librerie di cloni
che si sovrappongono. Ogni volta che un frammento di DNA è sequenziato, tali dati sono
utilizzati per identificare cloni sovrapposti, in modo che le sequenze di DNA prossime al
frammento possano essere identificate. Questo processo procede “camminando” lungo il
cromosoma in due direzioni fino a quando l’intera sequenza è clonata, mappata e
sequenziata.
19.61 Un dideossinucleotide ha atomi di idrogeno anziché gruppi idrossilici legati ai carboni 2′
e 3 ′ dello zucchero a 5 atomi di carbonio.
19.62 Poiché un dideossinucleotide non ha alcun gruppo 3’-idrossile, non può formare un
legame fosfoestereo con un altro nucleotide. Si comporta quindi da terminatore della
catena, poiché nessuna ulteriore polimerizzazione può verificarsi.
19.63 Le sequenze che questi tratti di DNA hanno in comune sono evidenziate in grassetto.
a. 5′ AGCTCCTGATTTCATACAGTTTCTACTACCTACTA 3′
b. 5′ AGACATTCTATCTACCTAGACTATGTTCAGAA 3′
c. 5′ TTCAGAACTCATTCAGACCTACTACTATACCTTGG GAGCTCCT 3′
d. 5′ACCTACTAGACTATACTACTACTAAGGGGACTATT CCAGACTT 3′
L’estremità 5 ′ della sequenza (a) è identica all’estremità 3 ′ della sequenza (c).
209
L’estremità 3 ′ della sequenza (a) è identico all’estremità 5 ′ della sequenza (d).
L’estremità 3 ′ della sequenza (b) è identica all’estremità 5 ′ della sequenza (c). Da 5 ′ a
3′, le sequenze formeranno la seguente mappa:
b
c
a
d
210








![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)

![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)

