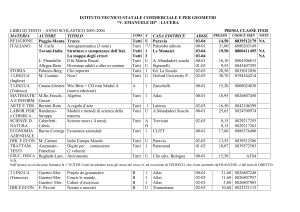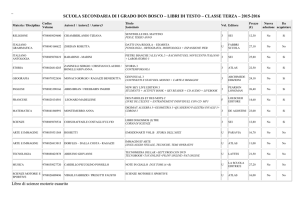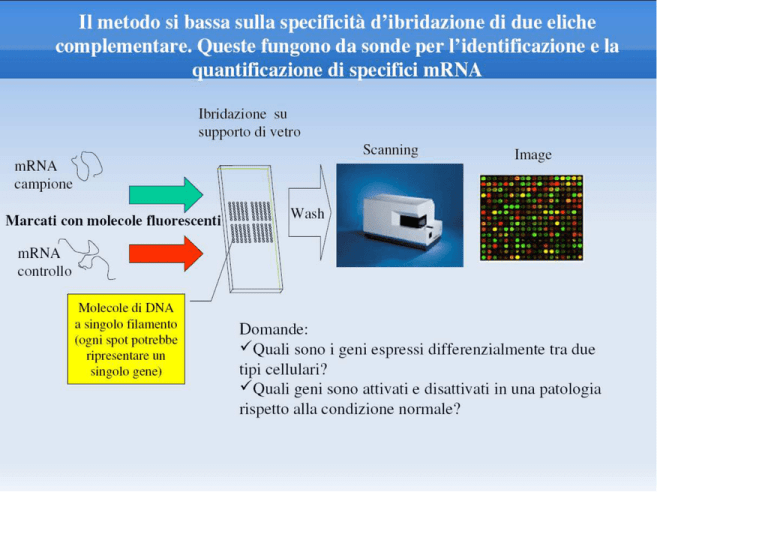
1 color vs 2 colors MicroArrays
1 color vs 2 colors MicroArrays
1 color (combimatrix)
2 colors (affymetrix)
+ Design custom
+ Stripping
+ Economicità
+ Design standard
+ Ibridazione
contemporanee
+ Analisi standard
- Analisi onerosa
- Sample disomogenei
- Design complesso
- Design fisso
- Costo
- Stripping
tocopherol
ArrayExpress repository query
ArrayExpress Atlas
Atlas per ArrayExpress è un database
arricchito semanticamente per la meta-analisi,
basato su statistiche globali su un sottoinsieme
di dati di AE appositamente selezionato.
Permette di esplorare in quali condizioni un gene
è espresso o quali geni sono differenzialmente
espressi in una particolare condizione, tessuto,
tipo di cellula, ecc.
ArrayExpress Atlas
ArrayExpress Atlas
ArrayExpress Atlas
ArrayExpress Atlas
ArrayExpress Atlas
ArrayExpress Atlas
ArrayExpress Atlas
ArrayExpress Atlas
ArrayExpress Atlas
ArrayExpress Atlas
ArrayExpress Atlas
ArrayExpress Atlas
ArrayExpress Atlas
ArrayExpress Atlas
Si possono fare ricerche sia per nome del gene che per
descrizione.
- Nel caso di più geni, si ottiene una heat-map combinata
- Si provi con "Violaxanthin de-epoxidase" (notare
autocompletamento) e
Lhcb in Arabidopsis thaliana
ArrayExpress Atlas
(per condizione)
Si possono fare ricerche per condizioni (es
cancer in Homo sapiens)
- In automatico i termini inerenti sono utilizzati
per la ricerca
ArrayExpress Atlas
(per condizione)
ArrayExpress Atlas
ricerca avanzata
Dal menu ‘Experimental factor’ , seleziona ‘disease state’. Il filtro selezionato
verrà aggiunto alla voce ‘Find genes matching all the following conditions’.
Restringere la ricerca ai/al gene/i che ‘is up in’ ed al tipo ‘hepatocellular
carcinoma’ nel text box a destra (auto-completamento disponibile).
Dal
menu
‘Experimental
factor’,
seleziona
‘organism
part’.
Restringere ora la ricerca ai/al gene/i che sono ‘down in’ e di tipo ‘liver’ nella
casella di testo.
Infine ancora dal menu ‘Experimental factor’ , seleziona ‘disease state’ e
restringi la ricerca al/ai gene/i che sono ‘is down in’ e di tipo ‘normal’ .
Clicca Search
- Tra i geni trovati annotare I valori di concordanza e discordanza
nell’espressione nel “nervous system”
Eliminiamo una colonna
Analisi dei dati di Microarray
Livelli di analisi
• Indentificare geni differenzialmente espressi di
interesse(fold-change)
• Cercare similarità tra gruppi di geni
• Identificare relazioni tra i geni identificati
• Connettere i dati a quelli di trascrittomica, proteomica,
ecc
Tipi di analisi
• Statistica (t-test, correzione), clustering, GO, ecc
Tecniche di Clustering
(“raggruppamento”)
Obiettivi
• Scoprire strutture/andamenti nei dati
• Raggruppare i geni in modo che quelli
appartenentei a un gruppo siano tra essi più
simili di quanto non siano rispetto a quelli di
un altro gruppo
Passo 1: decidere il tipo di
clustering
Clustering Gerarchico
• Singoli geni sono raggruppati in nodi a loro volta
raggruppati tra loro in base al criterio di similarità
• Il processo continua finchè tutti i geni sono raggruppati
(albero gerarchico)
Flat Clustering (e.g, K-means)
• Vettori di riferimento sono inizializzati casualmente
• I geni sono partizionati nel vettore a essi più simile
• Ogni vettore è ricalcolato come media dei geni contenuti
• I passi sono ripetuti fino alla convergenza (i vettori non
cambiano per iterazioni consecutive)
Qual’è il più adatto?
Confronto tra gruppi
• Tipo di trattamento
• Normale vs. Trattato
• Mutante vs. WildType
Serie temporali
• Ciclo cellulare
• Risposta al dosaggio
Passo 2: Scegliere una misura
• La distanza è una misura che corrisponde al
grado di similarità tra profili di espressione
• Ci sono molti modi di definire una distanza
• Ciascuno ha diverse caratteristiche e
permette di evidenziare differenti proprietà
dei dati
Passo 2: Scegliere una misura
• Distanza euclidea, la più breve distanza tra
2 punti (restituisce i due geni più simili)
• Distanza di Manhattan, la distanza tra due
punti misurata come lunghezza deli assi di
angoli retti (evidenzia i geni “esterni” perchè
ritorna na misura maggiore di quella
euclidea)
• Correlazione di Pearson, misura la
similitudine tra le “forme” dei profili di
espressione (1 simili, 0 non simili, -1 diversi)
• Ecc ecc
Passo 3: Scegliere un algoritmo di
clustering
L’algoritmo di clustering esprime le regole usate per decidere
quali punti di ciascun gruppo considerare nel valutare
l’appartenenza ai diversi cluster.
Single linkage cluster (basato sulla distanza più breve tra oggetti)
Average linkage cluster (basato sulla distanza media tra oggetti)
Complete linkage cluster (basato sulla distanza maggiore tra oggetti)
Comunque...
• I risultati del clustering sono molto sensibili al
metodo e ai parametri usati
• In assenza di informazioni biologiche è molto
difficile capire quale metodo di clustering
usare
• I risultati di 2 metodi di clustering possono
essere molto diversi
Può essere utile confrontare metodi di clustering
diversi
•
•
•
•
•
•
Gene Ontology è un vocabolario per descrivere la
biologia di un prodotto genico in un qualsiasi organismo
Ci sono 3 tipi distinti di “dizionari” o ontologie che
descrivono:
- la funzione molecolare di un prodotto genico
- il processo biologico in cui il prodotto genico partecipa
- e i componenti cellulari in cui il prodotto genico può
essere trovato
I termini di GO possono essere utili per identificare
relazioni tra grandi classi di geni con profili di
espressione simili come quelli raggruppati in cluster.
ArrayExpress Atlas