LE BASI AZOTATE
PURINE
PIRIMIDINE
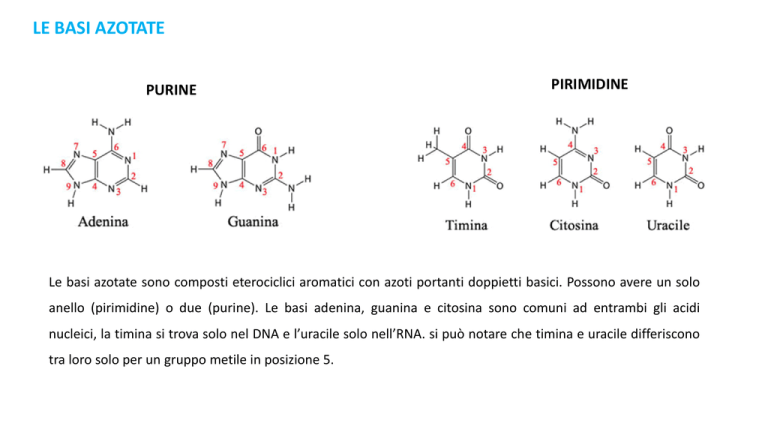
Le basi azotate sono composti eterociclici aromatici con azoti portanti doppietti basici. Possono avere un solo
anello (pirimidine) o due (purine). Le basi adenina, guanina e citosina sono comuni ad entrambi gli acidi
nucleici, la timina si trova solo nel DNA e l’uracile solo nell’RNA. si può notare che timina e uracile differiscono
tra loro solo per un gruppo metile in posizione 5.
Gli zuccheri che entrano a far parte della struttura degli acidi nucleici sono i seguenti due aldopentosi:
H
1’
O
H
C
2’
3’
4’
O
C
5’
OH
OH
OH
4’
3’
1’
2’
H
H
H
OH
H
OH
CH2 OH
CH2 OH
5’
ribosio
2’-deossiribosio
Il ribosio entra nella composizione dell’RNA mentre il deossiribosio del DNA. Nella numerazione delle posizioni
degli zuccheri si inserisce un apice (2’-) per distinguerla dalla numerazione delle posizioni delle basi azotate.
NUCLEOSIDI E NUCLEOTIDI
N H2
N
N H2
N
N
N
O
N
HO
–
N
O
HO
O
P
–
OH
un NUCLEOSIDE, costituito da uno
zucchero (in questo caso ribosio) e da
una base azotata (qui l’adenina), legati
mediante un legame β-N9-glicosidico.
Questo nucleoside è detto ADENOSINA.
N
O
N
O
O
HO
OH
un NUCLEOTIDE, costituito da uno zucchero e da una base
azotata, legati mediante un legame β-N9-glicosidico, con
l’aggiunta di un estere dell’acido fosforico sull’OH in
posizione 5’ del ribosio.
Questo nucleotide è detto ADENOSINA-5’-MONOFOSFATO.
ATP: IL MAGAZZINO DELL’ENERGIA
N H2
N
N
O
–
O
P
–
N
O
N
O
O
AMP
HO
ADP
OH
questi equilibri servono alle cellule per immagazzinare
energia là dove è disponibile (reazioni di fosforilazione,
endoergoniche, traggono l’energia necessaria dai processi
metabolici) e usarla dov’è richiesta (idrolisi, esoergonica)
ATP
GLI ACIDI NUCLEICI
Sono polimeri di nucleotidi, legati tra loro tramite legami 5’-3’:
N H2
N
N
O
O
in questo modo si ha una catena di
P
–
N
O
N
unità di zucchero monofosforilate a cui
O
O
sono legate le diverse basi azotate che
NH 2
O
–
O
P
costituiscono il codice genetico.
N
O
N
O
O
HO
O
Le basi azotate possono formare, tra loro, legami idrogeno specifici
che permettono il riconoscimento e l’appaiamento fra guanina e
citosina e tra adenina e timina (o uracile, che ha una struttura simile).
Ciò permette l’aggregazione della doppia elica
del DNA e la trascrizione del DNA in RNA.
DNA
struttura secondaria
Le interazioni specifiche si hanno sempre
tra una purina e una pirimidina, così che la
distanza sia quella ottimale.
Tra A e C, come tra T e G, potrebbe
formarsi un solo legame idrogeno.
Nelle fasi di separazione tra le catene della
doppia elica, la % di coppie CG è determinante
per l’entità dell’energia richiesta, dato che è la
coppia più fortemente legata.
struttura del B-DNA
doppia elica destrorsa
con filamenti
antiparalleli
il B-DNA è la forma più
comune e più stabile
un giro completo ogni 10 coppie di basi
un nucleotide
vi sono anche altre strutture per il DNA, indicate come A-DNA o Z-DNA, in cui l’elica
è avvolta in maniera più stretta o più rilassata o diversamente orientata.
Il DNA è contenuto in un NUCLEO delimitato da una membrana nucleare.
Nel nucleo il DNA (che è un acido) è avvolto intorno a proteine basiche (ISTONI) alle
quali è legato da forze elettrostatiche. Questo complesso DNA-istoni è chiamato
NUCLEOSOMA. Diversi nucleosomi costituiscono la CROMATINA.
ISTONI
NUCLEOSOMA
cromatina
La cromatina si avvolge in fibre di diametro 30 nm e queste in fibre da 200 nm,
che poi danno luogo ai CROMOSOMI (diametro 1 m).
un gene è una porzione di
DNA che codifica per una
proteina o un RNA. I geni
umani sono organizzati in
23 coppie di cromosomi.
REPLICAZIONE DEL DNA
replicazione
semiconservativa
la DNA polimerasi
allunga i filamenti
la DNA elicasi svolge
la doppia elica
la DNA primasi
sintetizza l’innesco
le proteine ssb rendono
lo stampo accessibile per
la duplicazione
1. la proteina ELICASI separa i due filamenti
2. le proteine single-strand binding (ssb) si legano ai singoli filamenti per impedirne la
riassociazione
3. l’enzima topoisomerasi impedisce eccessive tensioni
4. la primasi catalizza la formazione di un frammento di RNA da 10-12 nucleotidi
(primer o innesco) da cui inizia la replicazione
5. la DNA polimerasi catalizza la replicazione allungando il primer
La replicazione è un fenomeno bidirezionale ma la sintesi avviene in modo continuo solo
lungo il filamento 3’ – 5’, detto filamento guida, e quindi avviene sempre in direzione 5’ – 3’.
Sull’altro filamento, 5’ – 3’, la sintesi è discontinua (filamento ritardato o lagging) perché deve
sempre procedere in direzione 5’ – 3’, e procede per frammenti detti frammenti di Okazaki
Al termine, i primer
dovranno essere rimossi
e i frammenti di DNA
ricongiunti ad opera di
una DNA ligasi.
mRNA trasporta informazioni da DNA
ai ribosomi ed è complementare ad
un gene del DNA
rRNA è un componente
strutturale e funzionale dei
ribosomi, dove avviene la
sintesi delle proteine
tRNA traduce il codice di
mRNA in una sequenza di
amminoacidi
TRASCRIZIONE
La RNA polimerasi crea un legame stabile con il DNA e riconosce la sequenza promotore.
L’azione della RNA polimerasi è quella di aggiungere unità ribonucleotidiche al terminale 3’ OH libero. Essa sintetizza
in direzione 5’ 3’ avendo come stampo l’elica del DNA in direzione 3’ 5’ e quindi mRNA che ne risulta sarà copia
dell’elica non stampo che è la catena codificante. In pratica mRNA sarà una copia dell’elica 5’ 3’ con U al posto di T
e il ribosio al posto del deossiribosio.
Per permettere la trascrizione la doppia elica deve aprirsi temporaneamente formando una bolla di trascrizione
che scorre verso destra nel verso 5’ 3’, provocando lo srotolamento di altri tratti di DNA, mentre quello
trascritto, a sinistra, si riavvolge. La sintesi di RNA procede fino a un punto in cui la RNA polimerasi incontra
sequenze sul DNA che favoriscono la sua dissociazione e la fine della sintesi.
MODIFICAZIONI POST TRASCRIZIONALI
Aggiunta di 7-metilguanosina all’estremità 5’ del trascritto
Aggiunta di una coda di poli (A) all’estremità 3’ del trascritto
Splicing o rimozione degli introni (sequenze non codificanti)
t-RNA
forma legami idrogeno con
il CODONE complementare
situato sul mRNA
struttura tridimensionale del tRNA
il corretto tRNA si lega all’enzima.
L’amminoacido si lega covalentemente
al tRNA. L’AMP viene rilasciato.
Dato che vi sono 4 diverse basi azotate e 20 amminoacidi, il codice che permette di tradurre
l’informazione genetica del DNA in istruzioni per la sintesi di proteine richiede che ogni
amminoacido debba essere identificato da una tripletta di basi: se il codice prevedesse 2 basi
si potrebbero codificare 42 = 16 amminoacidi, mentre con un codice a 3 basi si possono avere
43 = 64 diverse triplette. Dato che questo numero è sovrabbondante, il codice è DEGENERATO,
ossia ogni amminoacido può essere codificato da più di una tripletta. Tuttavia, ogni tripletta
codifica un solo amminoacido (il codice è NON AMBIGUO). Vi sono anche triplette che non
codificano per alcun amminoacido, ma danno il segnale di STOP, ossia di termine della catena
di amminoacidi. Il segnale di START invece corrisponde alla tripletta AUG, che è anche l’unica
che codifica per l’amminoacido metionina. Il codice genetico è UNIVERSALE, ossia è comune a
tutti gli esseri viventi. L’informazione viaggia a senso unico, DNA RNA proteine.
Degenerazione del codice per gli
amminoacidi
START
Ala
Arg
Asn
Asp
Cys
Gln
Glu
Gly
His
Ile
Leu
Lys
Met
Phe
Pro
Ser
Thr
Trp
Tyr
Val
4
6
2
2
2
2
2
4
2
3
6
2
1
2
4
6
4
1
2
4





![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)






