DNA – REPLICAZIONE (1)
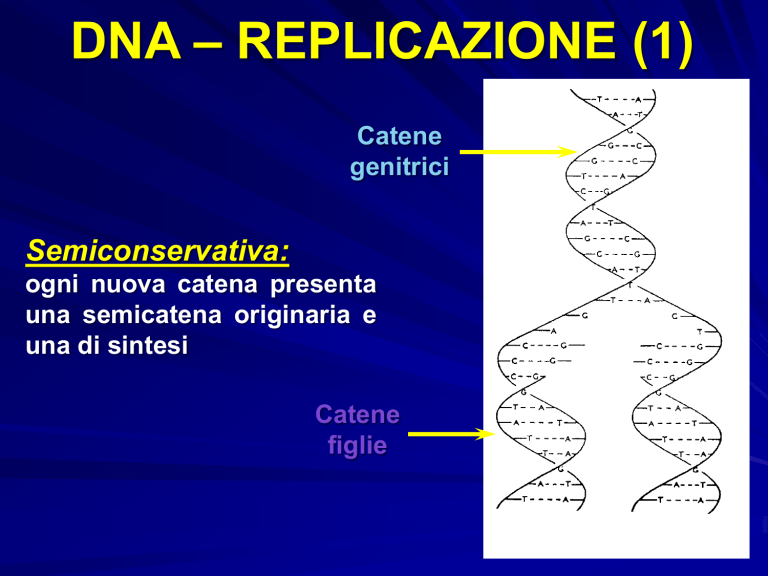
Catene
genitrici
Semiconservativa:
ogni nuova catena presenta
una semicatena originaria e
una di sintesi
Catene
figlie
DNA – REPLICAZIONE (2)
processo complesso che richiede l’ intervento di
moltissimi enzimi e di energia
le due semieliche del DNA vengono srotolate ad dalle
DNA elicasi
la singola elica viene stabilizzata finché non viene
copiata, ad opera delle proteine che legano il singolo
filamento (SSBP)
nella regione adiacente a quella srotolata, si crea un
superavvolgimento, per cui intervengono le topoisomerasi
(I e II) che operano i tagli e poi risaldano i filamenti
DNA – REPLICAZIONE (3)
DNA – REPLICAZIONE (4)
DNA – REPLICAZIONE (5)
le DNA polimerasi aggiungono nucleotidi al 3’ di una
catena polinucleotidica preesistente
il nuovo filamento cresce sempre dal 5’ al 3’
DNA – REPLICAZIONE (6)
nel punto di inizio della
replicazione è necessario un
innesco, costituito da un RNA
primer (5-14 nucleotidi) che viene
sintetizzato dalla RNA primasi
DNA – REPLICAZIONE (7)
5’
3’
Punti di origine della replicazione (circa ogni 150 kb)
3’
5’
Replicazione bidirezionale
Bolla di replicazione
Fusione delle bolle
5’
3’
5’
3’
Cromosomi figli
3’
5’
3’
5’
DNA – REPLICAZIONE (8)
Replicazione semidiscontinua
I due filamenti di DNA hanno una polarità opposta 5’ 3’ e 3’ 5’.
La DNA polimerasi sintetizza DNA solo nella direzione 5’ 3’.
Il DNA è replicato sui due filamenti stampo in direzioni opposte:
Filamento leader: sintetizzato da 5’ a 3’ nella direzione del
movimento della forcella di replicazione, la replicazione è continua e
richiede un solo RNA d’innesco
Filamento tardivo: sintetizzato da 5’ a 3’ nella direzione opposta
del movimento della forcella di replicazione, la replicazione è
discontinua e richiede molti RNA d’innesco
DNA – REPLICAZIONE (8)
5’
3’
filamento leader si replica in modo
Movimento
forcella di
replicazione
3’
continuo
filamento tardivo si replica in modo
discontinuo (frammenti di Okazaki)
3’
5’
5’
TRASMISSIONE DELL’INFORMAZIONE GENICA
DNA
RNA
PROTEINA
principio
di
collinearità
TRASCRIZIONE (1)
processo mediante il quale le informazioni contenute nel
DNA vengono trascritte enzimaticamente in una molecola
complementare di RNA
gene: unità di trascrizione costituita da un segmento di
DNA che codifica per una molecola di RNA e dalle
sequenze necessarie alla sua trascrizione
direzione 5’
3’
RNA polimerasi DNA-dipendente
promotore
- specifica regione del filamento stampo localizzata 20-30 nucleotidi a
monte del punto di inizio della trascrizione
- riconosciuto dai fattori di trascrizione
TRASCRIZIONE (2)
TRASCRIZIONE (3)
filamento senso
contiene la sequenza di DNA corrispondente al relativo
RNA
filamento antisenso
filamento stampo complementare alla catena di RNA in
crescita
TRASCRIZIONE (4)
3 fasi:
1. iniziazione
2. allungamento
3. terminazione
TRASCRIZIONE (5)
RNA (1)
acido nucleico che partecipa ai processi di:
espressione dei geni
biosintesi delle proteine
DNA
RNA
RNA (2)
H2O
NH3
sostituzione di un gruppo amminico con
un gruppo carbonilico
RNA (3)
struttura primaria: sequenza di basi in una molecola di RNA
struttura secondaria: struttura a doppia elica che si forma quando
regioni delle molecole di RNA si ripiegano su se stesse
struttura terziaria: conformazione a più elevati livelli di complessità
RNA (4)
RNA messaggero
(mRNA)
2%
RNA ribosomiale
(rRNA)
16%
RNA transfer
>80%
(tRNA)
mRNA
tipo di RNA che codifica e porta informazioni durante la
trascrizione dal DNA ai siti della sintesi proteica, per
essere sottoposto alla traduzione
Ciclo vitale dell’mRNA
1. trascrizione
2. modificazioni (pre-mRNA eucariotico)
splicing
cappuccio 5’
poliadenilazione
3. trasporto
4. degradazione
pre-mRNA
mRNA (1)
Cappuccio 5’
nucleotide guaninico metilato
in
posizione
7
aggiunto
all’estremità 5’ del pre-mRNA
inedito legame 5’-5’
funzioni:
- riconoscimento da parte del
ribosoma
- protezione dall’RNasi
mRNA (2)
Coda poliadenilata
lunga sequenza di nucleotidi adenina aggiunta al 3' del premRNA
funzioni:
- aumenta la stabilità dell’mRNA
- terminazione della trascrizione
- trasporto dell’mRNA
- traduzione
mRNA (3)
Regioni codificanti
composte da codoni che vengono decodificati e tradotti in
proteine dai ribosomi
cominciano con un codone di inizio
terminano con tre possibili codoni di stop
Regioni non codificanti (3’ UTR, 5’ UTR)
sezioni dell'RNA:
- posizionate prima del codone d'inizio/dopo il codone di stop
- non vengono tradotte
funzioni:
- stabilizzazione dell'mRNA
- localizzazione citoplasmatica dell'mRNA
- efficienza traduzionale
mRNA (4)
Introni
sequenze di un gene che vengono trascritte in RNA, ma non
vengono tradotte in proteine
presenti quasi esclusivamente nei geni eucariotici
variano per numero (0-60) e per lunghezza (200-50000)
mRNA (5)
mRNA (6)
tRNA (1)
piccola catena di RNA (74-95 nucleotidi) che trasferisce un
aa specifico ad una catena polipeptidica in crescita al sito
ribosomiale della sintesi proteica durante la traduzione
STRUTTURA
sito accettore (CCA)
braccio TΨC
braccio variabile
braccio dell’AC
braccio D (diidrouridina)
numerose basi modificate
tRNA (2)
BRACCIO DELL’AntiCodon (AC)
AC: costituito da 3 nucleotidi (tripletta)
ogni tripletta di uno specifico AC può appaiarsi ad
uno o più codoni per uno stesso aa (appaiamento incerto)
appaiamento accurato per le prime due basi mentre
per la terza può esistere una tolleranza agli
appaiamenti "sbagliati“
es. glicina (GGU, GGC, GGA, GGG)
rRNA
tipologia più abbondante di RNA presente nelle cellule
componente più conservato in tutte le cellule
fornisce un sistema per la decodifica dell’mRNA in aa
interagisce con il tRNA durante la sintesi proteica
RIBOSOMA
RIBOSOMA
DIFFERENZE NELLA TRASCRIZIONE
TRA EUCARIOTI E PROCARIOTI (1)
EUCARIOTI
1. 3 diverse RNA pol
PROCARIOTI
1. un unico tipo di RNA pol
2. l’mRNA viene maturato, 2. l’mRNA viene tradotto
trasportato nel ct e poi
mentre la trascrizione è
tradotto
in corso
3. i
geni
contengono 3. i geni non sono interrotti
introni ed esoni
4. mRNA monocistronici
4. mRNA policistronici
DIFFERENZE NELLA TRASCRIZIONE
TRA EUCARIOTI E PROCARIOTI (2)












