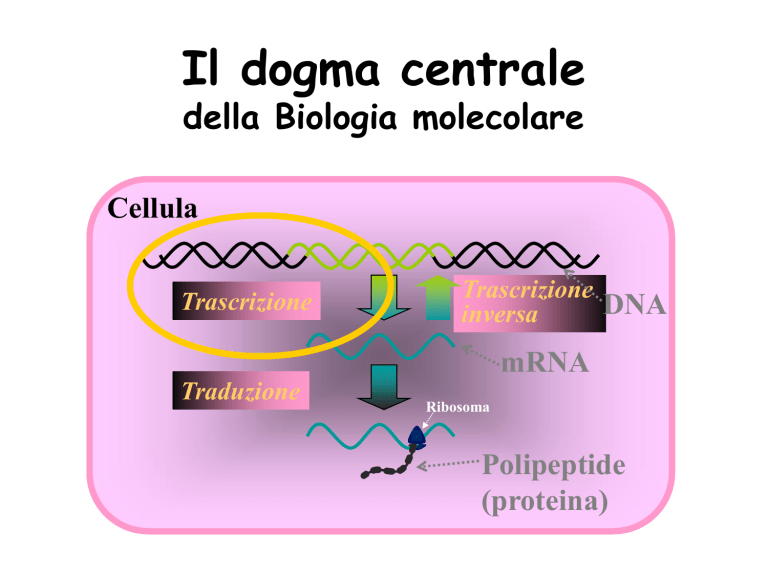
Il dogma centrale
della Biologia molecolare
Cellula
Trascrizione
Traduzione
Trascrizione
DNA
inversa
mRNA
Ribosoma
Polipeptide
(proteina)
ESPRESSIONE DEL GENOMA
L’Espressione del genoma è specifica del tipo di cellula e del suo stato.
Essa richiede la sintesi di molecole di RNA (trascrizione) che
eventualmente dirigeranno la sintesi di proteine (traduzione).
-l’RNA polimerasi non ha bisogno di un primer ma l’inizio avviene sole
su certe sequenze
-l’RNA sintetizzato non rimane appaiato allo stampo + enzimi possono
trascrivere lo stesso gene contemporaneamente
ESPRESSIONE DEL GENOMA
la trascrizione è meno accurata della replicazione
(1mismatch/1000 nt, assenza di meccanismi di riparazione).
Ogni errore che insorge durante la replicazione può essere
catastrofico e diventa permanente nel genoma di quell’individuo
e può essere trasmesso alla progenie
La trascrizione produce solo copie transienti di ciascuna regione
e danneggia solo un singolo trascritto tra i diversi che sono
prodotti
La trascrizione copia solo alcune porzioni del genoma e produce
anche mille copie di ciascuna regione
La replicazione deve copiare tutto il genoma una sola volta
IL PROTAGONISTA PRINCIPALE DELLA TRASCRIZIONE E’ L’ENZIMA…….
……… RNA polimerasi DNA dipendente
La struttura 3D dell’RNA polimerasi ricorda una chela
La scelta della regione da trascrivere e………………
………..in quale quantità
è un processo altamente regolato
una cellula batterica trascrive un gruppo diverso di
geni a seconda del terreno di cultura in cui sta crescendo
una cellula muscolare e un neurone trascrivono
gruppi diversi di geni (differenziamento cellulare)
LA SCELTA DELLA REGIONE DA TRASCRIVERE NON È CASUALE
Ciascuna regione contiene uno o più geni
Appositi segnali sul DNA indicano alla
RNA Polimerasi dove cominciare e
dove finire
Ci sono sequenze di DNA che
dirigono l’avvio della trascrizione
Ci sono sequenze di DNA che
decretano il termine della trascrizione
PROMOTORE
sequenza di nucleotidi che indica il punto di INIZIO
TERMINATORE
sequenza di nucleotidi che indica il punto di TERMINE
Si definisce UNITÀ DI TRASCRIZIONE
il tratto di DNA compreso fra il sito di
inizio e quello di termine per l’RNA
Polimerasi
Può, nei procarioti, comprendere più
di un gene
Il sito di inizio indica la posizione
sul DNA corrispondente alla prima
base incorporata nell’RNA
Il termine A MONTE identifica
sequenze che precedono l’unità di
trascrizione
IL PROMOTORE BATTERICO È
A MONTE DELL’UNITÀ DI
TRASCRIZIONE
Il termine A VALLE identifica sequenze nella stessa
direzione dell’espressione
IL TERMINATORE È A VALLE DEL CODONE DI INIZIO
SEQUENZE CONSERVATE
Una sequenza conservata è la sequenza che rappresenta le basi più spesso
trovate in una data successione ed in una particolare posizione dove
successione e posizione non sono casuali.
Tale sequenza può essere definita come una sequenza consenso
Una sequenza consenso è dedotta dall’allineamento di tutti gli esempi noti.
Una sequenza consenso può essere definito come un putativo sito di
riconoscimento sul DNA
PROMOTORE
Un promotore è definito dalla presenza di sequenze consenso in una
localizzazione specifica.
INSIEME DEI SEGNALI MOLECOLARI PRESENTI SUL
DNA RICONOSCIUTI DALL’RNA POLIMERSI
PROMOTORE
La regione -35
La regione -10
La distanza che separa le regioni -35 e -10
Start point
Startpoint
Lo startpoint (+1) è di solito (>90%
delle volte) una purina. Spesso è la
base centrale della sequenza CAT,
ma la conservazione della tripletta non
è frequente
La sequenza -10
regione di 6 bp riconoscibile in quasi tutti i promotori.
Il centro dell’esamero è generalmente circa 10 bp a
monte dello startpoint, ma la distanza varia da -18 a -9.
L’esamero è spesso chiamato Pribnow box.
Pribnow box
Il consenso è TATAAT, e può essere indicato
nella forma
T80A95T45A60A50T96
dove il pedice indica la percentuale
dell’occorrenza della base più comune, che
varia dal 45 al 96%.
La basi più importanti sembrano essere le più
conservate: la TA iniziale e la T finale.
La sequenza -35
Un altro esamero conservato è centrato
~35 bp a monte dello startpoint.
È chiamata la sequenza -35.
Il consenso è TTGACA
in forma più dettagliata
T82T84G78A65C54A45
Distanza –10 / -35
• La distanza che separa i siti
• –35 e –10 è tra 16-18 bp nel 90%
dei promotori
• La sequenza nella regione non è
importante, ma
la distanza è critica.
Orientazione della RNA pol
• I promotori sono
vettoriali e indicano
quale dei due
filamenti deve essere
usato come stampo
ELEMENTI A MONTE DELLA REGIONE - 35
Siti Fis
Elemento Up
UP
PROMOTORE ESSENZIALE (minimo)
Molti promotori possiedono altre sequenze “consenso”
poste a monte della sequenza -35 (elementi UP e siti FIS)
• Elementi presenti in promotori di geni la cui espressione è altamente
regolata (promotori ribosomali)
• Sito di legame x proteine in grado di regolare l’ espressione genica
IL PROTAGONISTA PRINCIPALE DELLA TRASCRIZIONE E’ L’ENZIMA…….
……… RNA polimerasi DNA dipendente
L'RNA polimerasi (DNA-dipendente) è un’enzima appartenente alla classe delle
transferasi che catalizza la seguente reazione:
Nucleoside trifosfato + RNAn ⇄ RNAn+1 + pirofosfato
Le RNA Polimerasi
I Batteri posseggono una RNA polimerasi
• relativamente semplice
Gli Archaea posseggono una RNA polimerasi …….ma
• è più complessa
• ha caratteristiche funzionali simili a quelle degli Eucaria
gli Eucaroti hanno tre RNA polimerasi
• Pol I : RNA ribosomali
• Pol II : RNA messaggeri
• Pol III: tRNA e piccoli RNA
Nella cellula eucariotica esistono altre RNA polimerasi : l’RNA polimerasi
mitocondriale e l’RNA polimerasi dei cloroplasti
LE RNA POLIMERASI EUCARIOTICHE
La RNA Pol I, localizzata nel nucleolo, trascrive il gene per il precursore
degli RNA ribosomiali (40% dei trascritti totali)
La RNA Pol II è responsabile della trascrizione della maggior parte dei
geni, inclusi quelli codificanti proteine, e alcuni piccoli RNA nucleari (U1,
U2, U4, U5)
La RNA Pol III trascrive i geni per i tRNA, alcuni piccoli RNA nucleari (es.
U6) e l’RNA ribosomiale 5S
RNA polimerasi di E.coli è un’enzima
costituito da piu’ subunità
β
oloenzima
(155 kD)
costituiscono il core catalitico
β’ (160 kD)
La subunità α
(40 kD) è richiesta per l’assemblaggio
del core dell’enzima, per il riconoscimento del promotore e
per l’interazione con effettori (attivatori o repressori).
La subunità σ (32-90 kD)
Esistono diversi tipi di σ
specifiche per gruppi diversi di
geni da trascrivere
La subunità ω
contribuisce alla
stabilità del complesso proteico.
La presenza di σ fa
aumentare l’affinità
dell’enzima x il DNA
SUBUNITÀ alpha
La subunità alpha mediante il dominio C terminale
riconosce e lega l’elemento UP e questo determina un
legame molto stretto tra polimerasi e promotore
ELEMENTO UP
Sequenza ricca di A-T.
Interagisce con la subunità α
della RNA polimerasi.
Si trova tipicamente nei
promotori che sono molto
espressi, quali quelli dei
geni per gli rRNA.
SUBUNITÀ sigma
SIGMA CONTROLLA IL RICONOSCIMENTO DEL PROMOTORE
La subunità sigma contiene regioni che riconoscono in modo specifico gli
elementi a -35 e a -10
due eliche della regione 4 di sigma
prendono contatto con la regione -35
mediante un dominio di legame al DNA
helix-turn-helix
solco maggiore
L’ elica coinvolta nel
riconoscimento della regione -10
interagisce con le basi del filamento
non stampo in maniera da
stabilizzare il DNA aperto
Fattori σ
I promotori hanno tutti la stessa dimensione e localizzazione relativa allo
start-point, ed hanno sequenze conservate (–35 e –10).
MA
Esistono promotori con differenti sequenze consensus
E. coli ha diversi fattori sigma che
riconoscono promotori con
differenti consensus
L’espressione di differenti fattori
sigma è indotta da differenti
condizioni ambientali
La reazione di trascrizione ha tre passaggi
La sintesi dell’RNA, come quasi
tutte le reazioni biologiche di
polimerizzazione, avviene in tre
fasi:
Inizio
Allungamento
Termine
L’INIZIO DELLA TRASCRIZIONE
LA RNA POLIMERASI NON RICHIEDE UN PRIMER
La trascrizione non richiede l’intervento di un primer, ma è necessaria una
interazione molto specifica con il ribonucleotide di inizio, che normalmente è
una A.
a) l’oloenzima si lega debolmente al DNA più
volte alla ricerca di un promotore
b) l’oloenzima ha trovato un
promotore e si lega debolmente ad
esso a formare un complesso chiuso
c) l’oloenzima si è legato saldamente al
DNA denaturandone localmente i due
filamenti e formando un complesso
aperto (bolla di trascrizione)
Affinità dell’RNA pol al DNA
Il nucleo (core) dell’RNA polimerasi ha un’affinità intrinseca per il
DNA basata su interazioni fra l’enzima (basico) e il DNA (acido) e
si lega stabilmente a siti di legame aspecifici sul DNA.
L’associazione del core al fattore sigma rende molto labile il
legame a siti aspecifici e consente all’enzima di riconoscere siti
specifici con elevata affinità
L’RNA POLIMERASI CIRCONDA LA BOLLA
L’RNA Polimerasi, quando si lega al promotore, separa i due filamenti del DNA
in una “BOLLA” transitoria e utilizza uno dei filamenti come stampo per
dirigere la sintesi di una sequenza complementare di RNA
I processi di appaiamento e
aggiunta delle basi
avvengono all’interno della
bolla
La sintesi procede in
direzione 5’
3’
Il 3’OH dell’RNA nascente
forma il legame
fosfodiestereo con il
nucleoside 5’ trifosfato in
arrivo allungando la catena.
LA TRASCRIZIONE AVVIENE PER APPAIAMENTO DELLE BASI ALL’INTERNO
DELLA ”BOLLA” DI DNA NON APPAIATO
DURANTE LA TRASCRIZIONE LA BOLLA SI
MANTIENE ALL’INTERNO DELL’RNA
POLIMERASI
L’ENZIMA SVOLGE E RIAVVOLGE IL DNA
L’ENZIMA SINTETIZZA L’RNA
La bolla si sposta insieme all’RNA
polimerasi man mano che l’enzima si
muove lungo il DNA e la catena
dell’RNA si allunga
La lunghezza della bolla è di circa 1214 bp e la lunghezza dell’ibrido DNARNA al suo interno è di circa 8-9 bp
LA BOLLA DI TRASCRIZIONE SI SPOSTA LUNGO IL DNA
I processi di appaiamento e aggiunta
delle basi avvengono all’interno della
bolla e sono catalizzati e controllati
dall’enzima
La sintesi dell’RNA parte dall’estremità 5’
e procede in direzione dell’estremità 3’
Il gruppo 3’OH dell’ultimo nucleotide
aggiunto alla catena reagisce con il
nucleotide 5’ trifosfato in arrivo che si
aggiunge alla catena.
IL SITO ATTIVO CONTIENE LA BOLLA DI TRASCRIZIONE
Una parete proteica
costringe il DNA a
curvarsi all’interno
del sito attivo della
RNA polimerasi
LA RNA POLIMERASI MANTIENE APERTO IL DNA
L’INIZIO DELLA TRASCRIZIONE
Comprende più fasi
1) La RNA polimerasi si lega al DNA in
un complesso chiuso
2) Il complesso chiuso si converte nel
complesso aperto
3) La polimerasi incorpora i primi 9 o
10 nucleotidi nella catena nascente di
DNA
4) La polimerasi rilascia il suo fattore
sigma e comincia la fase di
allungamento
TRASCRITTI ABORTIVI
Dopo l’ingresso del ribonucleotide di inizio nel sito attivo, segue una fase
denominata inizio abortivo, nella quale la polimerasi sintetizza brevi
frammenti di RNA (<10 nt) che vengono rilasciati senza che la polimerasi si
dissoci dallo stampo, sul quale inizia poi la sintesi di un nuovo frammento.
La regione 3.2 di sigma
ostruisce il canale di uscita
dell’ RNA
Il ciclo di σ
Appena la RNA polimerasi, oloenzima, ha superato
la fase abortiva e inizia a spostarsi lungo lo
stampo allungando l’RNA, si dissocia il fattore σ
che raggiunge il core di una nuova polimerasi per
iniziare la sintesi di un nuovo RNA
ESPRESSIONE DEL GENOMA
Molte copie di RNA possono essere prodotte a partire da un singolo gene
poichè l’RNA sintetizzato non rimane appaiato allo stampo e più molecole di
RNA polimerasi possono trascriverlo in un determinato momento.
I trascritti all’estremità 3’ del gene sono più lunghi poichè la direzione
della trascrizione è da sinistra a destra.
estremità 5’del gene
estremità 3’del gene
FORZA DI UN PROMOTORE
Per forza di un promotore s’intende il numero di trascritti a cui esso può
dare inizio in un dato tempo.
La forza di un promotore è collegata alla affinità di legame con la
polimerasi ed alla capacità di evadere dal promotore
I MODULATORI TRASCRIZIONALI
I batteri devono rispondere repentinamente ai
cambiamenti delle condizioni ambientali per cui l’attivita’
trascrizionale deve essere velocemente attivata o repressa
L’espressione genica e’ regolata da proteine regolative che
possono attivare o reprimere la trascrizione
(attivatori o repressori)
Gli attivatori e i repressori sono proteine
in grado di legarsi al DNA
GLI ATTIVATORI AGISCONO SECONDO DUE
MODALITA’:
INTERAZIONE PROTEINA-PROTEINA
CLASSE I
Si legano a monte della -35 e ancorano la RNAP interagendo con il
dominio α-CTD facilitando il riconoscimento del promotore da parte
dell’enzima
CLASSE II
Si legano sempre a monte della – 35 ma interagiscono con la subunità σ
dell’enzima
GLI ATTIVATORI AGISCONO SECONDO DUE
MODALITA’:
VARIAZIONE CONFORMAZIONALE
Legano il promotore tra la regione -10 e -35 facilitando il riconoscimento
del promotore da parte dell’RNA pol
I REPRESSORI AGISCONO SECONDO TRE
MODALITA’:
•Ingombro sterico
Il repressore lega la regione compresa tra le sequenze - 10 e - 35
impedendo il riconoscimento del promotore
•Inducendo la
formazione di un
loop
Più molecole di repressore legano il DNA ( in regioni anche lontane dal
promotore) interagiscono tra di loro impedendo il legame dell’RNA pol
con la regione del promotore
I REPRESSORI AGISCONO SECONDO TRE
MODALITA’:
• Per modulazione
di un attivatore
Il repressore lega l’attivatore impedendone l’interazione con la RNA pol
CARATTERISTICHE GENERALI DELLE RNA POLIMERASI
Sintetizza RNA in direzione 5’
b=blue
b’ = viola
= giallo e verde
3’
Si muove srotolando l’elica del DNA (Elicasi +
Topoisomerasi)
Non necessita di un primer ma l’inizio avviene solo
su specifiche regioni (promotori) attività
primasica
Aggiunge nucleotidi usando un filamento di stampo
Utilizza ribonucleosidi 5’-trifosfato (ATP, GTP, UTP e
CTP) e richiede Mg++
Una finestra di 9 nt DNA-RNA si muove lungo il DNA
Ogni nucleotide è selezionato in base alle regole
della complementarietà A:U e G:C e alla geometria
della coppia di basi
ALLUNGAMENTO
è il passaggio nella reazione di sintesi della macromolecola
quando la catena nucleotidica si allunga per l’aggiunta di subunità.
Dopo diversi tentativi, quando la RNA polimerasi riesce a sintetizzare
un frammento più lungo di 10 nt, inizia la fase di allungamento e si ha
poi il distacco del fattore .
L’allungamento è catalizzato dal core enzimatico e procede alla max
velocità di 40 basi/sec. L’allungamento si arresta in presenza del
nucleotide scorretto.
TERMINE DELLA TRASCRIZIONE
è il passaggio che termina la sintesi della macromolecola
bloccando l’addizione delle subunità, e generalmente causando
la dissoluzione dell’apparato di sintesi
Tutte le sequenze necessarie per la terminazione sono nella regione
trascritta
Alcune sequenze, dette terminatori
inducono il rilascio della RNA polimerasi
e del trascritto.
La trascrizione termina ed il core
enzimatico si dissocia dal DNA in
corrispondenza dei terminatori
I terminatori batterici sono di due tipi:
rho-indipendenti, e rho-dipendenti.
Formazione di un RNA hairpin
TERMINAZIONE DELLA TRASCRIZIONE
I terminatori batterici sono di due tipi: i) rho-indipendenti, e ii) rho-dipendenti.
I terminatori rho-indipendenti, detti anche terminatori intrinseci, sono costituiti da
una corta sequenza palindromica (ca. 20 nt) cui segue una sequenza di 8 coppie
A:T.
Il terminatore è una struttura a
forcina seguita da una serie di
residui U. In genere vi è una
regione ricca di GC nei pressi
della base dello stelo.
La delezione dei residui U
all’estremità
3’
non
provoca più la fine della
trascrizione
L’interferenza provocata dalla struttura stem-loop unita
alla debolezza del legame A:U favorisce il distacco del
trascritto dallo stampo.
La struttura stem-loop
assunta dall’RNA
provoca la rottura del
complesso di
allungamento
Viene aperto a forza il
canale di uscita
dell’RNA?
Si interrompono le
interazioni tra l’RNA e lo
stampo?
La proteina Rho
La proteina Rho, è una proteina essenziale per E.coli. Ha una struttura
omoesamerica (monomero di 46 kDa) e assume una forma ad anello,
contiene un dominio di legame al ssRNA e un dominio di legame all’ATP. Si
lega all’RNA nascente ed ha attività elicasica.
La terminazione Rho-dipendente
(1/3)
Nel secondo caso la terminazione richiede il fattore proteico rho
(che lega siti specifici detti rut).
Rho Utilization Site
La terminazione non può
avvenire se rut viene
tradotto
Proteina
Ribosoma
Rho lega il sito rut
sull’mRNA e si sposta
lungo il messaggero
inseguendo la RNA
polimerasi
La terminazione Rho-dipendente
Il movimento di Rho
richiede l’idrolisi
dell’ATP
(2/3)
La RNA polimerasi si ferma
su un terminatore;
Rho raggiunge la polimerasi.
La terminazione Rho-dipendente
(3/3)
L’attività elicasica di
rho causa la
dissociazione dell’ibrido
RNA-DNA e richiede
ATP.
Molte copie di RNA possono essere prodotte a partire da
un singolo gene poichè più molecole di RNA polimerasi
possono trascriverlo in un determinato momento.
I trascritti all’estremità 3’ del gene sono più lunghi poichè
la direzione della trascrizione è da sinistra a destra.
estremità 5’del gene
estremità 3’del gene
RNA Polimerasi
La RNA Polimerasi svolge le seguenti funzioni:
1. Riconoscimento della regione promotrice
2. Svolgimento della doppia elica del DNA
(Elicasi + Topoisomerasi)
3. RNA Priming (Primasi)
4. RNA Polimerizzazione
5. Riconoscimento del terminatore












