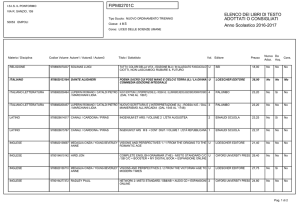
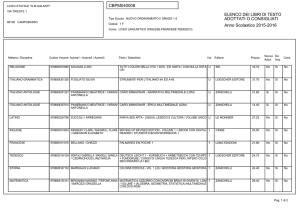
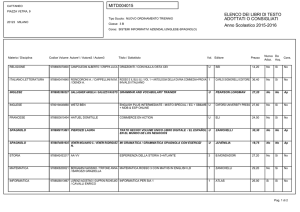
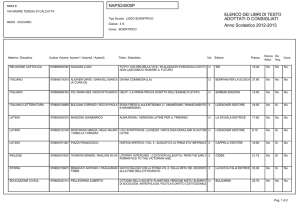
Lezioni di
biotecnologie
2
Lezione 2
Analisi del DNA
e delle proteine
3
© Zanichelli editore, 2014
Analizzare DNA e proteine
Per le applicazioni delle biotecnologie
è di fondamentale importanza:
1. essere in grado di identificare con precisione
le sequenze di DNA di interesse;
2. analizzare il contenuto della loro informazione
per stabilirne la funzione;
3. identificare i prodotti proteici corrispondenti;
4. saper localizzare geni e proteine all’interno
delle cellule.
Esistono tecniche in grado di rispondere a tutte
queste esigenze.
4
© Zanichelli editore, 2014
Leggere il DNA
• Un aspetto fondamentale delle biotecnologie è la
possibilità di poter leggere la sequenza del DNA, in
modo da identificare con certezza la presenza dei geni o
delle regioni regolative che ci interessa clonare.
• La lettura della sequenza del DNA ha costituito una
tappa fondamentale nella nascita delle biotecnologie.
• Il procedimento attraverso cui si identificano le singole
basi che compongono una molecola di DNA è detto
sequenziamento.
5
© Zanichelli editore, 2014
Il moderno sequenziamento (I)
Per il sequenziamento sono necessari:
• il filamento di DNA stampo (di cui vogliamo leggere la
sequenza);
• un innesco complementare a una regione dello
stampo (per l’inizio della reazione di DNA polimerasi);
• la DNA polimerasi;
• una miscela di 4 nucleotidi (dNTPs);
• i 4 dideossinucleotidi (ddNTPs) fluorescenti.
6
© Zanichelli editore, 2014
Il moderno sequenziamento (II)
La DNA polimerasi viene incubata in presenza del DNA,
dei 4 dNTPs e di uno specifico ddNTP, posto a un
rapporto molare con il corrispondente dNTP (per esempio
ddATP verso dATP), tale per cui venga incorporato solo a
una determinata frequenza.
Se la concentrazione di ddATP fosse troppo alta, infatti,
tutte le molecole di DNA si arresterebbero alla prima T.
In questo modo, invece, solo una frazione del DNA
sintetizzato sarà bloccato ogni volta che sullo stampo
è presente una T.
7
© Zanichelli editore, 2014
Il moderno sequenziamento (IV)
Facendo quattro
reazioni
separate, una per
ogni ddNTP, sarà
possibile
identificare ogni
base presente
sullo stampo,
basandosi
sull’incorporazione
del ddNTP
complementare.
8
© Zanichelli editore, 2014
Sequenziamento del DNA per PCR (I)
Nella metodica classica di Sanger era necessaria
una quantità rilevante di DNA per poter rilevare
i frammenti sintetizzati.
Oggi è possibile sequenziare quantità minime di DNA
grazie all’uso della PCR, in cui il campione di partenza
viene prima amplificato e successivamente vengono
aggiunti i ddNTPs fluorescenti.
I moderni sequenziatori automatici utilizzano
l’accoppiamento tra PCR e metodo Sanger.
I frammenti vengono separati in microtubi
di poliacrilammide e contemporaneamente letti
dal raggio laser.
9
© Zanichelli editore, 2014
Sequenziamento del DNA per PCR (II)
Un sequenziatore automatico Immagine:
Konrad Förstner via Wikimedia
Commons
Traccia della sequenza
generata dal
sequenziatore
10
© Zanichelli editore, 2014
Analisi delle sequenze
Nel 2003 è stato completato il sequenziamento del
genoma umano. Ma anche il genoma di molti altri
organismi è stato completamente sequenziato.
La sequenza del DNA genomico è costituita da una serie
ininterrotta di milioni o miliardi di lettere (ciascuna
corrispondente ad una base A,T,C,G) e quindi la sua
lettura dall’occhio umano non consente di estrarre alcuna
informazione utile.
Per poter decodificare l’informazione contenuta nella
sequenza del DNA sono necessari strumenti
bioinformatici.
11
© Zanichelli editore, 2014
Analisi delle sequenze: fase di lettura
Il livello di analisi più semplice è l’individuazione di
geni, ovvero di sequenze codificanti.
Dato che ogni amminoacido è codificato da una tripletta
(3 nucleotidi), ciascuna sequenza di DNA può essere
letta in tre modi diversi (a partire cioè dalla prima,
seconda o terza base) o fasi di lettura.
I programmi informatici effettuano la lettura a triplette
su ciascuna delle tre possibili fasi di lettura e identificano
quella in grado di codificare per un prodotto proteico
(generalmente le fasi sbagliate mostrano codoni di stop
dopo pochi amminoacidi e quindi non sono geni).
12
© Zanichelli editore, 2014
Analisi delle sequenze: omologie (I)
Tutte le sequenze accumulate negli anni sono conservate
in banche dati liberamente accessibili.
Una nuova sequenza può così essere confrontata con
quelle note. I programmi sono in grado di evidenziare
le sequenze comuni e il grado di somiglianza.
Sequenze identiche avranno la medesima funzione.
Sequenze che si assomigliano identificheranno
probabilmente funzioni simili in organismi evolutivamente
distanti.
Grazie all’analisi comparativa delle sequenze è
possibile identificare geni simili in organismi diversi,
costruire alberi genealogici e stabilire relazioni evolutive
tra i geni.
13
© Zanichelli editore, 2014
Analisi delle sequenze: omologie (II)
Mappa circolare
che compara il genoma del
cromosoma 1 umano con
l’intero genoma del topo: le
linee colorate collegano i geni
ortologhi, ovvero geni simili per
struttura tra le due specie.
14
© Zanichelli editore, 2014
Localizzazione del DNA nella cellula
Oggi è possibile evidenziare la posizione di una qualsiasi
sequenza di DNA sul genoma cellulare in vivo.
Viene utilizzata la tecnica FISH (fluorescence in situ
hybridization) o ibridazione fluorescente in situ, che
sfrutta la capacità delle sequenze complementari di DNA
di appaiarsi.
15
© Zanichelli editore, 2014
FISH: applicazioni
Grazie alla FISH è stato possibile mappare la disposizione
spaziale dei cromosomi umani nel nucleo interfasico
delle cellule.
Immagine: Bolzer et al.
(2005), PloS Biology, via
Wikimedia Commons
16
© Zanichelli editore, 2014
Western Blotting
È possibile identificare la presenza di una specifica
proteina all’interno di una miscela complessa
utilizzando la tecnica detta Western Blotting.
La tecnica si divide in 3 fasi:
1. Separazione delle proteine su gel di poliacrilammide
2. Trasferimento delle proteine su membrana
3. Rilevazione con anticorpi specifici per la proteina di
interesse.
17
© Zanichelli editore, 2014
Western Blotting: il gel
La miscela di proteine viene caricata su di un gel di
policrilammide.
Questo è un polimero dell’acrilammide, la cui struttura
tridimensionale è formata da fibre. Il suo potere risolutivo
è superiore a quello dell’agarosio, in virtù della maggiore
densità delle maglie.
L’acrilammide liquida viene miscelata con opportuni
reagenti che ne causano la polimerizzazione a
temperatura ambiente.
18
© Zanichelli editore, 2014
Western Blotting: separazione (I)
La poliacrilammide viene fatta
polimerizzare in verticale tra due
vetri in presenza di una
mascherina (detta «pettine») che
forma in cima al gel delle tasche
(i pozzetti) in cui sarà depositato
il campione. Lo spessore del gel
è di circa 1,5 mm.
Al gel viene applicato un campo
elettrico e le proteine vengono
separate sulla base del loro peso
molecolare, in modo analogo ai
frammenti di DNA sul gel di
agarosio.
Immagine: Mark Sommerfeld via
Wikimedia Commons
19
© Zanichelli editore, 2014
Western Blotting: separazione (II)
La separazione avviene facendo migrare le proteine dal
polo negativo a quello positivo, ma le proteine non
hanno una carica uniforme negativa e spesso hanno
forme diverse (globulari, filamentose) che potrebbero
influenzare la corsa.
20
© Zanichelli editore, 2014
Western Blotting: separazione (III)
Le proteine separate sul gel possono essere visualizzate
con diversi coloranti (per esempio il blu di Coomassie).
Elettroforesi su gel di
poliacrilammide con
proteine colorate dal blu
di Coomassie
21
© Zanichelli editore, 2014
Western Blotting: trasferimento (I)
Il gel contenente le proteine (non colorato) viene messo
in contatto con una membrana di nitrocellulosa, una
sostanza carica positivamente che tratterrà le proteine
cariche negativamente.
Il gel e la membrana sono messi tra due fogli di carta
assorbente e inseriti in un apparecchio che applica un
campo elettrico, facendo migrare le proteine dal gel alla
membrana.
22
© Zanichelli editore, 2014
Western Blot: trasferimento (II)
Immagine: Bensaccount via Wikimedia Commons
23
© Zanichelli editore, 2014
Western Blotting: trasferimento (II)
Le proteine fissate sulla membrana possono essere
evidenziate con diverse colorazioni reversibili (per
esempio rosso Ponceau) per controllare l’avvenuto
trasferimento.
Immagine: Argymeg via
Wikimedia Commons
24
© Zanichelli editore, 2014
Western Blotting: rivelazione
Grazie a questa tecnica
è possibile identificare
la presenza di una
singola proteina tra
migliaia di proteine
diverse.
25
© Zanichelli editore, 2014
Identificazione di proteine in cellula (I)
Utilizzando una tecnica analoga
alla FISH, è possibile
visualizzare una proteina
all’interno della cellula.
La tecnica di
immunofluorescenza si basa
sull’utilizzo di un anticorpo
primario specifico per
la proteina di interesse e di
un anticorpo secondario in
grado di legare quello primario
e coniugato ad un gruppo
fluorescente.
26
© Zanichelli editore, 2014
Identificazione di proteine in cellula (II)
Le cellule vengono fissate su di un vetrino e incubate con
l’anticorpo primario. Successivamente sono poste
in contatto con l’anticorpo secondario. Infine vengono
osservate al microscopio con illuminazione laser. Il segnale
fluorescente mostra la posizione della proteina nella cellula.
È possibile usare anticorpi con fluorescenze di colore
diverso per evidenziare più proteine contemporaneamente.
27
© Zanichelli editore, 2014