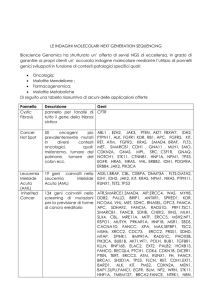
UNI EN ISO 9001:2008, UNI EN ISO 13485:2004
IDENTIFICATIONE DELLA MUTAZIONE TP53
(Analisi con sequanziamento del gene)
AMPLI-TP53 somatic mutations
Cat. n.1.435seq
TP53 è il gene mutato più comunemente associato al cancro come il glioblastoma, il carcinoma renale a cellule chiare, il cancro a cellule
squamose della testa e del collo, il grave carcinoma ovarico, il cancro polmonare a cellule non piccole, il cancro gastrico, il carcinoma
epatocellulare, il colangiocarcinoma, il cancro del seno e della prostata (mediamente 25-60%). Nelle neoplasie ematopoietiche le
mutazioni TP53 sono meno frequenti (10-15%) ma sono comunque tra i 10 geni mutati più significativamente presenti.
I primi studi sulle mutazioni TP53 si sono concentrati sui tumori con delezioni alleliche al sito TP53 (17p13.1) ed hanno mostrato che
le mutazioni risultanti in sostituzioni di un singolo aminoacido erano frequenti sull’allele rimasto intatto. Inoltre è stato notato che i
tumori aventi entrambi gli alleli parentali mostrano frequentemente mutazioni puntiformi su un allele suggerendo una sorta di effetto
dominante della mutazione.
Di tutte le mutazioni puntiformi del gene TP53 solo 12 mutazioni sono altamente ricorrenti. Queste mutazioni rappresentano i così detti
“hotspots” del DNA mutazionale.
Fino ad ora la raccomandazione accettata per l’analisi molecolare del TP53 è suggerita per la leucemia linfatica cronica (CLL) in cui la
mutazione somatica TP53 è predittiva di una scarsa risposta alle terapie convenzionali. Le mutazioni rappresentano la forma più comune
del TP53 inattivato nel CLL e sono frequentemente accompagnate (70% dei casi) dalla perdita del secondo allele a causa della delezione
17p13. L’implicazione di queste osservazioni molecolari è raccomandare lo studio sia della presenza sia della delezione 17p13, grazie
alla tecnica FISH, e della mutazione TP53, grazie al sequenziamento del gene. L’importanza clinica delle anormalità del TP53 nel CLL
è strettamente correlata alla prognosi infausta, dovuta a questa lesione genetica, e alla sua associazione con la chemioresistenza
(chemioterapia e immunochemioterapia) e dunque con la resistenza sia alle terapie convenzionali che alle nuove terapie mirate.
Il kit per le mutazioni somatiche del TP53 segue le amplificazioni degli esoni 2,3,4,5,6,7,8,9 del gene TP53 a ulteriori analisi eseguite
tramite sequenziamento del DNA.
Principio del metodo:
A) estrazione del DNA genomico
B) amplificazione
C) rilevazione su gel di agarosio
D) sequenziamento del DNA
REAGENTI E CONSERVAZIONE
AMPLIFICATION
Mix PCR EX 2-3
Mix PCR EX 4
Mix PCR EX 5-6
Mix PCR EX 7
Mix PCR EX 8-9
Sterile H2O
Taq Polymerase (5U/µl)
Applicabilità: su DNA genomico purificato da campione di
sangue
intero
o
tessuto
Tests:40
Stabilità: fino a 18 mesi se correttamente conservato (gel di
agarosio, se protetto dalla luce, può essere conservato un
anno
a
temperatura
ambiente)
Attrezzatura necessaria: tubi da 1.5ml con tappo a vite; tubi
da 15ml in polipropilene; vassoio per tubi raffreddato; punte
sterili (barriera areosol); vetro Pasteur; tubi no-oil.
Reagenti non inclusi nel kit: acqua sterile bidistillata ,
cloroformio, etanolo 70%, etanolo 100%, reagenti per
sequenziamento.
Strumentazione richiesta: microcentrifuga con rotore fisso
per tubi da 2ml (13-15.000g); pipette pre-PCR e post-PCR; (0.5
– 20 µl, 10 – 100 µl, 20 – 200 µl, 200 – 1000 µl);
termociclatore programmabile, cappa Class II Bio Hazard
Biological Safety, sistema per elettroforesi con alimentazione,
transilluminatore UV, sistema fotodocumentale, sequenziatore
DNA
Tutti i materiali devono essere sterili, DNase e RNase free e
monouso.
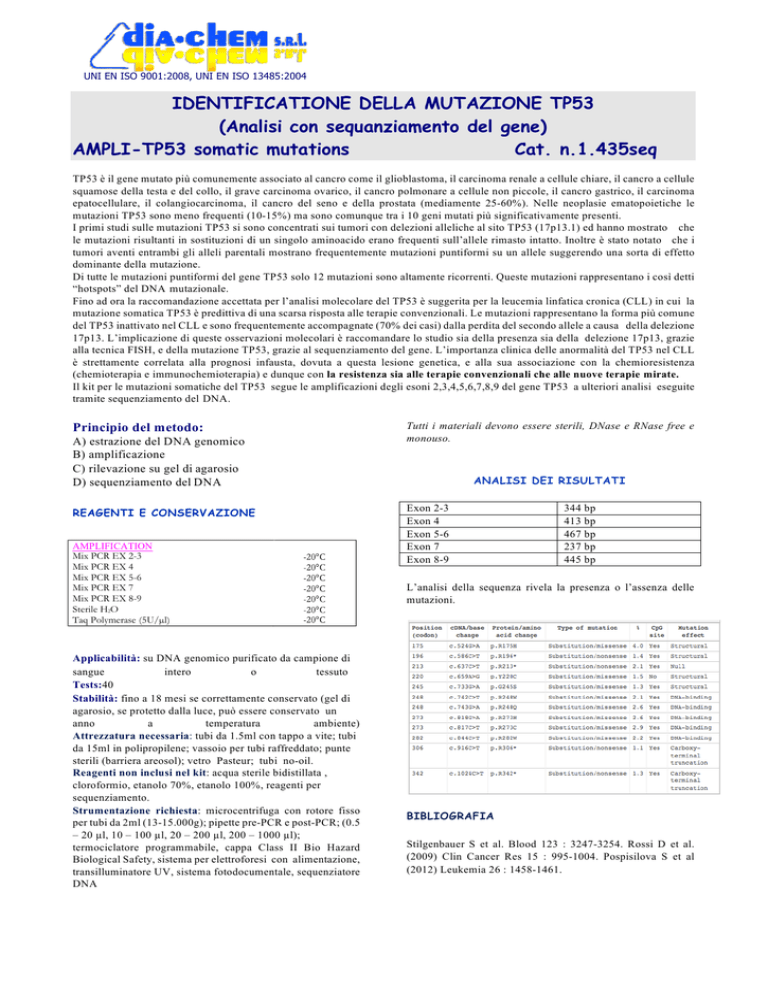
ANALISI DEI RISULTATI
Exon 2-3
Exon 4
Exon 5-6
Exon 7
Exon 8-9
344 bp
413 bp
467 bp
237 bp
445 bp
L’analisi della sequenza rivela la presenza o l’assenza delle
mutazioni.
BIBLIOGRAFIA
Stilgenbauer S et al. Blood 123 : 3247-3254. Rossi D et al.
(2009) Clin Cancer Res 15 : 995-1004. Pospisilova S et al
(2012) Leukemia 26 : 1458-1461.