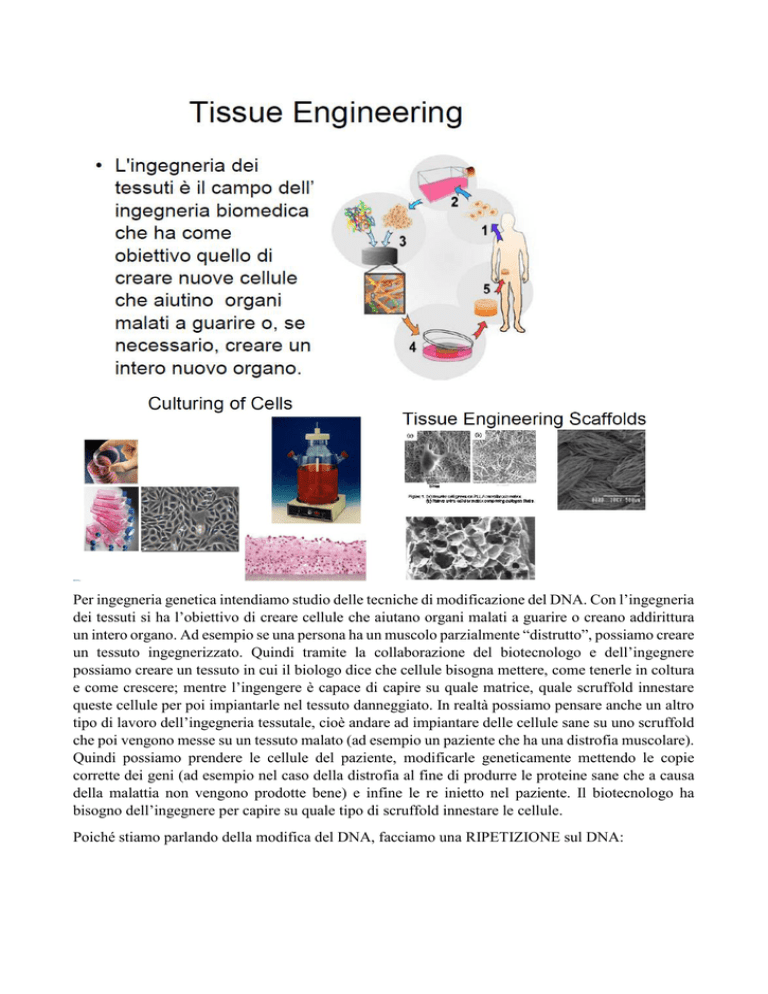
Per ingegneria genetica intendiamo studio delle tecniche di modificazione del DNA. Con l’ingegneria
dei tessuti si ha l’obiettivo di creare cellule che aiutano organi malati a guarire o creano addirittura
un intero organo. Ad esempio se una persona ha un muscolo parzialmente “distrutto”, possiamo creare
un tessuto ingegnerizzato. Quindi tramite la collaborazione del biotecnologo e dell’ingegnere
possiamo creare un tessuto in cui il biologo dice che cellule bisogna mettere, come tenerle in coltura
e come crescere; mentre l’ingengere è capace di capire su quale matrice, quale scruffold innestare
queste cellule per poi impiantarle nel tessuto danneggiato. In realtà possiamo pensare anche un altro
tipo di lavoro dell’ingegneria tessutale, cioè andare ad impiantare delle cellule sane su uno scruffold
che poi vengono messe su un tessuto malato (ad esempio un paziente che ha una distrofia muscolare).
Quindi possiamo prendere le cellule del paziente, modificarle geneticamente mettendo le copie
corrette dei geni (ad esempio nel caso della distrofia al fine di produrre le proteine sane che a causa
della malattia non vengono prodotte bene) e infine le re inietto nel paziente. Il biotecnologo ha
bisogno dell’ingegnere per capire su quale tipo di scruffold innestare le cellule.
Poiché stiamo parlando della modifica del DNA, facciamo una RIPETIZIONE sul DNA:
Il DNA è il materiale
ereditario
che
si
trasmette
da
una
generazione ad un’altra
ed è fondamentale
perché serve ad una
cellula per far capire
quali proteine produrre
e quindi, assicurarsi il
corretto funzionamento
di quella cellula. Quindi
se la cellula funziona
correttamente ha tutto
un assetto di proteine
corretto
(come
ad
esempio la cellula
muscolare assicurerà la
corretta funzionalità anche del muscolo). Il DNA ha una struttura a doppio filamento, ogni filamento
si avvolge l’uno sull’altro, ed è formato da monomeri (che sono le unità fondamentali). Ogni
monomero è costituito da: uno zucchero, una base azotata ed un gruppo fosfato. Tali “unità” si
uniscono l’uno all’altro mediante il gruppo fosfato. Innazitutto lo zucchero nel DNA è uno zucchero
a 5 atomi di carbonio chiamato deossiribosio, che è diverso dall’RNA che ha il ribosio. Quindi nel
DNA abbiamo il deossiribosio perché presenta un H anziché un OH (gruppo ossidrilico). Di solito
sono 5 atomi di carbonio, quindi si numerano a partire da 1,2,3,4,5 e in corrispondenza del 5° atomo
di carbonio c’è un legame con l’azoto che si definisce “fosfodiestereo” che unisce il fosfato al 1°
zucchero, dopodicchè questo fosfato è unito al secondo zucchero in posizione 3 e di nuovo il 2°
fosfato ha una posizione al carbonio 5 un altro fosfato che sarà unito al 3° in posizione 3 e così via
(vedi fig.).
Allora c’è una polarità in cui 3’ ha l’OH e
il 5’finisce con un fosfato. Ciò permette
di creare dei legami covalenti tra i diversi
nucleotidi perché c’è questo continuo
legame zucchero-fosfato che mantiene
uniti i nucleotidi lungo l’elica. Questi
legami sono legami covalenti. Però il
DNA è costituito da 2 eliche che hanno un
orientamento opposto: in corrispondenza
dell’OH c’è il fosfato ed in
corrispondenza del fosfato c’è l’OH,
quindi ho il cosidetto orientamento
antiparallelo (5’3’-3’5’). Le due eliche si
avvolgono l’una sull’altra perché ci sono
dei legami tra le basi azotate
corrispondenti. Queste ultime non
combaciano tra loro in maniera casuale ma sappiamo che nel DNA:
𝑡𝑖𝑚𝑖𝑛𝑎 − 𝑎𝑑𝑒𝑛𝑖𝑛𝑎
{
𝑐𝑖𝑡𝑜𝑠𝑖𝑛𝑎 − 𝑔𝑢𝑎𝑛𝑖𝑛𝑎
Questi legami sono legami deboli e quindi possono essere facilmente rotti a differenza degli altri che
sono legami covalenti e quindi difficili da rompere perché c’ bisogno di un’energia maggiore. Il DNA
nelle cellule eucariotiche (nostre cellule, che hanno un nucleo e organelli e sono molto più complesse
rispetto a quelle dei batteri che sono cellule procariotiche) è compattato perché si avvolge su se stesso.
La doppia elica infatti si avvolge intorno a delle proteine dette “istoniche” 1.3 volte per guadagnare
spazio e formando la struttura nucleosoma (DNA+proteine istoniche). I diversi nucleosomi si
dispongono l’uno accanto all’altro per creare una fibra che ha un diametro di 30 nanometri. Questa si
avvolge a sua volta su un’altra proteina (scaffold) e quest’ultima si avvolgerà ancora su se stessa a
formare il cromosoma. Tutto questo avviene perché una cellula è molto piccola (ordine del micron),
invece il DNA può arrivare a 2 metri, quindi tale sistema di riorganizzazione del DNA è di
fondamentale importanza. Noi abbiamo 23 coppie di cromosomi mentre il cromosoma batterico è
un’unica molecola di DNA circolare chiusa (ovviamente ci sono sempre le due eliche. Anche esso è
compattato).Però per i batteri abbiamo anche i plasmidi che sono altre molecole di DNA presenti nei
batteri molto più piccole. Se vogliamo duplicare una cellula, occorre duplicare tutto di essa (i
mitocondri, tutti gli organelli, oltre il DNA).
Come avviene la replicazione del DNA? Ci sono 2 eliche che possono separarsi (perché non ci sono
legami forti ma deboli) e vi è un’enzima che si chiama la “DNA-polimerasi”, che seguendo la
complementarietà delle basi azotate (A-T, C-G), riesce a sintetizzare un nuovo filamento che è
proprio uguale al primo. Da un filamento riusciamo ad ottenerne 2 e le cellule riescono così a separarsi
e duplicarsi perché hanno due copie uguali del DNA. Ogni volta che parlaremo di enzimi sono delle
proteine che hanno un’attività enzimatica ossia riescono a fare una reazione enzimatica, cioè riescono
a trasformare un substrato in un prodotto (ad esempio quest’enzima riesce a prendere i singolo
monomeri (zucchero, fosfato con la base azotata) a metterli in corrispondenza con la basse
corrispondente e a unirli con un altro successivo per formare la catena completa unita tra di loro con
tutti legami covalenti).
Quando parliamo di
enzimi sentiremo spesso
polimerasi,
fosfatasi,
chinasi, ecc. Sono enzimi
che fanno qualcosa. Una
chinasi ad esempio è un
enzima che mette un
fosfato, una ligasi è un
enzima che unisce, la
polimerasi
unisce
nucleotidi e forma un
nuovo filamento, però
non sa farlo da sola ma ha
bisogno di essere guidata
sul punto dove deve
aggiungere i nucleotidi e
lo fa perché ci sono delle
piccole sequenze che vengono messe sul DNA, che vengono chiamate “primer”, quindi la DNA
polimerasi in corrispondenza dei primer si attacca al filamento separato e inizia a sintetizzare il
secondo filamento. Una volta avvenuta la duplicazione c’è bisogno di produrre le proteine necessarie
per il suo corretto funzionamento (ad esempio supponiamo che sia una cellula muscolare, essa è
caratterizzata da alcune proteine quali miosina e actina che sono proteine che si mettono una
intercalata all’altra e quando c’è il segnale di contrarre il muscolo scivolano uno verso l’altra e
accorciano il muscolo e si tira l’osso e si alza il braccio. Per cui la cellula muscolare ha bisogna di
produrre queste proteine e quest’informazione viene appunto dal DNA), quindi ci sono delle regioni
chiamate geni (sequenze di circa 3-4000 basi) e bisogna passare l’informazione dal nucleo al
citoplasma, e come si fa? C’è un processo chiamato trascrizione, cioè l’informazione del gene viene
copiata in una molecola di mRNA che è molto simile al DNA eccetto per:
-Ha un singolo filamento;
- Si appaia sempre in base alla complementarietà ma al posto della timina c’è l’uracile.
-Ribosio anziché deossiribosio
Tale molecola di mRNA viene trasportata nel citoplasma e da informazioni per sintetizzare la proteina
di interesse. Perché? Si appaia a piccole molecole di DNA chiamate tnRa, che sono caratterizzate da
2 zone: una zona dove ci sono tre basi che possono essere riconosciute per complementarietà sulla
sequenza dell’mnRA e un’altra zona che lega l’amminoacido, che è l’unita fondamentale della
proteina. Quindi se ad esempio la tripletta A-G-G viene riconosciuta per complementarietà T-C-C e
questo tnRA trasporta un amminoacido la lanina significa che in questa posizione deve stare la lanina,
dopo la meteonina, la lanina di nuovo dopo ad esempio, e in questo modo si crea una catena fatta da
amminoacidi che è la proteina e in questo modo si sintetizza correttamente la proteina del muscolo.
Siccome noi dobbiamo
parlare di ingegneria
genetica, vuol dire che
andiamo a modificare il
DNA e con l’ingegneria
genetica
alteriamo
volontariamente
il
materiale genetico di un
organismo. Lo si fa
tramite delle tecniche che
prendono il nome di
“DNA ricombinante”.
Per definizione è la
produzione
di
una
molecola
attraverso
l’unione di due o più
frammenti di DNA che
non sono normalmente associati e possono derivare da diverse sorgenti biologiche. Si può prelevare
l’informazione voluta da un qualsiasi organismo, isolarla ed associarla ad un plasmide, creando
un’unica molecola chiamata “DNA ricombinante”e produrre grazie ad essa la proteina di interesse.
La tecnologia del DNA ricombinante ha reso possibile clonare geni. Possiamo prendere il gene che
produce l’informazione del colore rosso degli occhi della mosca, ad esempio, (quindi dal suo genoma
isolare solo quella porzione di quel gene) e associarla ad un plasmide (quei piccoli elementi di DNA
che si trovano nei batteri insieme ai cromosomi); quindi unire i geni della mosca ai plasmidi, creare
un’unica molecola chiamata DNA ricombinante e produrre grazie ad essa la proteina di interesse.
Che
significa
clonare i geni?
Vuol dire isolare
un tratto di DNA e
amplificare questo
tratto tante volte
(fino ad ottenere
una popolazione di
DNA identica alla
molecola
di
partenza).
Per isolare il gene ci sono diverse tecniche. Esistono degli enzimi, detti di restrizione che tagliano il
DNA di interesse e quindi in corrispondenza di specifiche sequenze definite “palindromi” (nomi che
letti da destra a sinistra o da sinistra a destra sono uguali, ad es. Anna). Quando riconosce tali sequenza
l’enzima rompe i legami fosfodiesterici e taglia, esso però non taglia in maniera netta ma solamente
in maniera spezzettata quindi avrò un filamento più lungo rispetto ad un altro e se quindi taglio un
secondo DNA che ha un origine differente (come per esempio il plasmide) con lo stesso enzima di
restrizione produrrà allora le stesse sequenze sfalsate che si possono appaiare a quelle tagliate in
precedenza seguendo la redola della complementarietà e si forma così una nuova molecola
ricombinante (molecola circolare chiusa). Esiste un ulteriore enzima che unisce successivamente le
rotture sullo scheletro del DNA che è la LIGASI (crea legami covalenti tra gli zuccheri ed il fosfato).


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)






![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)


