SOC di Farmacologia
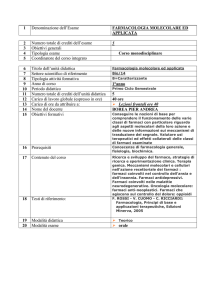
Sperimentale e Clinica
VI° CONGRESSO NAZIONALE
FITeLab
Slow Medicine Laboratory:
IT’s the Future
1-2-3 Ottobre 2015
Ospedale Borgo Roma
(Verona)
La farmacogenetica nella
gestione del paziente con
tumore colorettale,
l’esperienza traslazionale al
CRO di Aviano
Parte tecnico-pratica
Franca SARTOR
TLBM
Farmacologia Sperimentale e
Clinica
CRO – Aviano
Screening pre-terapeutico di markers farmacogenetici
correlati alle tossicità da chemioterapia
0 alleli
Dose Standard
varianti DPYD
Pazienti candidati a
chemioterapia a base di
Fluoropirimidine e/o
Irinotecano
Screening genetico pre-terapia
DPYD*2A
DPYD*13
DPYD-2846A>T
UGT1A1*28
1 allele
variante
DPYD
2 o più alleli
varianti
DPYD
UGT1A1*28/*28
Dose IRI 70%
UGT1A1*1/*1
UGT1A1*1/*28
Possibile inclusione negli studi
di fase 1b
Riduzione Dose
Fluoropirimidine
del 50%
Prediligere una
terapia
alternativa
DESCRIZIONE DEL PROCESSO – Diagramma di flusso
INIZIO
Arrivo campione da analizzare
Accettazione e registrazione
NO
Identificazione campione
CORRETTA
Ricontattare il richiedente
SI
NO
Stoccaggio temporaneo
Analisi immediata
SI
Analisi campione
SI
NO
Analisi effettuata
correttamente
SI
Emissione del referto e stoccaggi
del campione biologico residuo
FINE
SOC di Farmacologia
Sperimentale e Clinica
TIPO DI ANALISI
RICHIESTE/DISPONIBILI
Polimorfismi del gene DPYD e tossicità severa
da fluoropirimidine
Metabolismo cellulare dei
Fluoro-derivati
DPD E’ L’ENZIMA CHIAVE LIMITANTE
PER LA DETOSSIFICAZIONE DEI
FLUORO-DERIVATI NELLE CELLULE
Polimorfismi indagati:
DPYD *2A
(IVS14+1A/G, rs3918290)
DPYD *13
(1679T/G, rs55886062)
DPYD 2864 A/T
(rs67376798)
Polimorfismi delle isoforme UGT1A1 e UGT1A7
e terapia basata sull’IRINOTECANO
UGT1A1 E UGT1A7
SONO LE ISOFORME
DELL’ENZIMA UGT
MAGGIORMENTE
IMPLICATE NEL
CATABOLISMO
DELL’IRINOTECANO
UO Complessa di Farmacologia
Sperimentale e Clinica
STRUMENTI UTILIZZATI PER L’ANALISI
Pyrosequencing
e
Sequenziatore per analisi frammenti
Pyrosequencing™
PSQ 96 MA (Biotage AB)
PyroMark Q96 ID (Qiagen)
Pyrosequencing™
Reazione di Minisequenziamento per sintesi
Tale metodica si basa su una quantificazione luminometrica
indiretta dei gruppi pirofosfato (PPi) che vengono liberati dai
nucleotidi man mano incorporati durante la sintesi di un
filamento di DNA complementare ad un templato stampo.
Assay design
Richiesti 3 PRIMERS
Due primers (di cui uno biotinilato) per l’amplificazione tramite PCR della
zona target contenente lo SNP
• Primer biotinilato [conc]< 0,2µM
• Prodotto PCR 200-300 bp
Un primer di sequenza
•
•
•
•
Complementare al filamento biotinilato
Tm: 40°C-50°C
Lunghezza: circa 15 basi.
Posizionamento flessibile:
terminale 3’, dopo appaiamento col filamento biotinilato (toglierei), deve risultare
collocato adiacentemente al sito polimorfico
Quando la base polimorfica forma un omopolimero con le basi adiacenti il primer di
sequenza deve sovrapporsi alla regione omopolimerica
Forward / Reverse Pyrosequencing assay
Pyrosequecing Assay
PREPARAZIONE del CAMPIONE
1) Amplificazione della regione di DNA
contenente lo SNP (Y/R) tramite PCR
con un primer BIOTINILATO (B)
IMPIEGO del PYROSEQUENZIATORE
Aggiunta di un nucleotide alla volta secondo un
preciso ordine di dispensazione
primer di
sequenza
nucleotidi incorporati
NNNNNNN
Y/R
B
1) liberazione di gruppo PPi
2)
separazione del singolo
filamento biotinilato
Y/R
3)
2) emissione di luce
visibile che viene
evidenziata nel
pirogramma tramite un
picco proporzionale ai
dNTPs incorporati
B
appaiamento con il
primer di sequenza
primer di sequenza
Y/R
B
3) spegnimento del segnale
(apirasi) e aggiunta del
nucleotide successivo
Sequenza nucleotidica
Nucleotidi aggiunti
4) sintesi del filamento
complem. e
determinazione della
sequenza
Pyrosequecing: esempio di risultati
C/C wild type
C/T eterozigote
T/T mutato
DPYD in DIAGNOSTICA – SNP analizzati
DPYD *2A
(IVS14+1A/G, rs3918290)
DPYD *13
(1679T/G, rs55886062)
DPYD 2864 A/T
(rs67376798)
SOC di Farmacologia
Sperimentale e Clinica
SEQUENZIATORE PER
ANALISI DEI FRAMMENTI
Sequenziatore per analisi frammenti
genetic analyzer ABI PRISM 3100
Analisi dei frammenti
Metodica basata su una elettroforesi capillare associata ad un’analisi di fluorescenza
impiegata per studiare STR (short tandem repeats) o delezioni/inserzioni in quanto
permette di separare frammenti di DNA di lunghezza diversa, amplificati utilizzando un
primer marcato con un fluoroforo, in base alla loro dimensione.
2
3
1
Schema del processamento di
frammenti di DNA all’interno dello
strumento Genetic Analyzer ABI
Prism 3100
4
Interpretazione dei risultati con il software Gene Scan
Separazione di frammenti di DNA di varie
dimensioni marcati con differenti fluorofori
(rosso=ROX, verde=JOE, giallo=LIZ) Il più piccolo
è il più veloce ed esce per primo (t1) mentre il più
grande esce per ultimo (t3) .
Esempio: determinazione dell’UGT1A1*28
94bp
6/6
ripetizioni
94bp
96bp
6/7
ripetizioni
96bp
7/7
ripetizioni
SOC di Farmacologia
Sperimentale e Clinica
ANALISI DEI DATI E
REFERTAZIONE
DNLAB
DNfirma
INTERPRETAZIONE RISULTATI SUGGERIMENTI
DPYD
WT per tutte e tre le varianti:
Non sono state riscontrate controindicazioni genetiche all'utilizzo di un dosaggio standard di fluoro
pirimidine.
Eterozigote per una sola variante :
Sulla base della variazione genetica riscontrata è consigliabile una riduzione del dosaggio iniziale di
fluoroprimidine al 50% della dose standard per limitare l’incidenza di tossicità severa al trattamento,
in base alle linee guida SIF-AIOM. Un aggiustamento successivo della dose può essere effettuato in
accordo con le norme della normale pratica clinica.
Eterozigote per più di una variante
Sulla base delle variazioni genetiche riscontrate è consigliabile valutare l’utilizzo di una terapia
alternativa alle fluoroprimidine per l’alto rischio di sviluppare tossicità severa al trattamento.
Omozigote mutato per una delle varianti:
è consigliabile valutare l’utilizzo di una terapia alternativa alle fluoroprimidine per l’alto rischio di
sviluppare tossicità severa al trattamento.
UGT1A1*28
*1/*1 (WT):
Non sono state riscontrate controindicazioni genetiche all'utilizzo di un dosaggio standard di
irinotecano.
*1/*28 (ETEROZIGOTE):
Non sono state riscontrate controindicazioni genetiche all'utilizzo di un dosaggio standard di
irinotecano.
*28/*28 (MUTATO):
Sulla base della variazione genetica riscontrata è consigliabile una riduzione del dosaggio iniziale di
irinotecano al 70% della dose standard per limitare l’incidenza di tossicità severa al trattamento, in
base alle linee guida SIF-AIOM. Un aggiustamento successivo della dose può essere effettuato in
accordo con le norme della normale pratica clinica.
SOC di Farmacologia
Sperimentale e Clinica
GRAZIE PER
L’ATTENZIONE