
DNA batterico
Il DNA batterico si replica in modo
semiconservativo, utilizzando entrambi i filamenti
come stampo e richiede l’intervento di numerosi
enzimi.
9
Nei batteri le mutazioni possono essere indotte e/o
spontanee
9
Nei batteri può avvenire uno scambio (trasferimento)
di informazioni geniche:
- unidirezionale da donatore a ricevente
- non è mai trasferito l’intero cromosoma
- può avvenire tra specie diverse
DNA: materiale genetico primario
Ogni tripletta di basi consecutive
definisce un codone e codifica per un
aminoacido;
1000-1500
coppie
di
nucleotidi costituiscono un gene (proteina
di 300 – 500 AA)
Cromosoma batterico
1-5×106 coppie di basi, circa 3.000- 6.000
geni. Non ci sono introni.
DNA polimerasi: III interviene nella
replicazione, I e II nei meccanismi di riparo
Elicasi: intervengono nello svolgimento
dell’elica
Topoisomerasi I e II (girasi): intervengono
nella separazione delle eliche
Primasi: RNA polimerasi che sintezzano i
“primers”, corte sequenze nucleotidiche che
fungono da innesco
Schema della replicazione semiconservativa del DNA: Eliche figlie
(nuove) complementari alle eliche parentali (vecchie). Ciascuna copia
contiene un’elica vecchia ed una nuova
Replicazione del DNA Le elicasi, con l’aiuto delle girasi,
Il cromosoma batterico
rimane circolare durante
la replicazione
La replicazione inizia da
un’origine, prosegue
nelle 2 direzioni (2 forche
replicative), termina
quando le forche si
incontrano: intermedi a
forma di “theta”
svolgono la doppia elica
La primasi sintetizza un corto
innesco di RNA (1-5 nucleotidi)
complementare alla elica stampo;
DNA pol III a partire da 3’-OH del
primer sintetizza (5’ Æ 3’) un’elica
complementare
al
filamento
“leading” e
“frammenti di
Okazaki” discontinui (circa 1000
nucleotidi) (5’ Æ 3’) sull’altro
filamento (lagging o lento)
La DNA pol I rimuove gli RNA
primers e sintetizza i pezzetti
mancanti
La ligasi lega i frammenti tra loro
per ricostituire la molecola
Potenzialità di errore
10-8-10-9 (mutazioni)
errori corretti da DNA pol III e I
Replicazione del DNA
Modello di replicazione a “rolling circle”
(cerchio rotante)
Utilizzato durante la coniugazione e dai plasmidi oltre che
da alcuni virus
I.
in corrispondenza di un’origine (O) una delle 2 eliche
viene tagliata
II. l’estremità libera 3’-OH funziona da primer per la
DNA pol III, che inizia a copiare l’elica
complementare. La catena interrotta si dipana e forma
una coda libera con l’estremità 5’
III. la neosintesi procede
IV. e la coda si allunga
V. la nuova catena è completamente sintetizzata e la
parentale, completamente dipanata, si distacca
VI. quest’ultima catena serve come stampo per la sintesi di
una catena complementare, che può a questo punto
circolarizzare (alla fine del processo si hanno 2 nuove
molecole di DNA)
Anche in questo caso la replicazione è semiconservativa
Il codice genetico
♦ universale
♦ degenerato
♦ senza sovrapposizioni
Codoni:
triplette
Codone AUG
(Met): inizio
sintesi
proteica
Codoni
STOP:
fine sintesi
proteica
Trascrizione: sintesi di RNA
RNA polimerasi sintetizza RNA (mRNA, rRNA, tRNA) su di uno
stampo di DNA, rappresentato dal filamento “senso”
l’RNA polimerasi batterica è costituita da 6 subunità (α2, β, β’, ω, σ);
gli mRNA sono frequentemente policistronici e colineari con il DNA
trascritto: non c’è “splicing”
Dato che la sintesi di mRNA nei
batteri avviene nel citoplasma,
spesso si ha accoppiamento di
trascrizione e traduzione, cioè la
sintesi proteica inizia già prima
della fine della trascrizione
Traduzione dell’mRNA: sintesi proteica
3 fasi
inizio, allungamento, terminazione
A) Schema generale
B) Inizio: mRNA si lega alla subuità
30S libera Æ l’iniziatore (Nformilmetionil-tRNA, fMet-tRNA)
si lega con l’anticodone ad AUG nel
sito P; la subunità 50S si associa al
complesso
Allungamento: il ribosoma scorre
sull’mRNA
di
un
codone
(traslocazione), il secondo AAtRNA entra nel sito A e si forma il
legame peptidico con fMet ad opera
di peptidil-trasferasi. Il tRNA
scarico si sposta nel sito E ed esce
dal ribosoma; vengono aggiunti i
nuovi AA (sito A, legame nel sito P,
uscita tRNA nel sito E)
Terminazione: si ha quando nella
sequenza dell’mRNA si trova un
codone STOP. L’ultimo AA viene
chiamato carbossi-terminale.
Il ribosoma si dissocia, la proteina
viene rilasciata e il gruppo formilico
viene rimosso da fMet.
Meccanismi di regolazione genica
Consentono alla cellula di produrre proteine (enzimi) solo quando servono
Ø
ECONOMIA CELLULARE
•
Instabilità dell’mRNA
(nei batteri, emivita: 4-6 minuti, necessità di continua sintesi)
• Controllo della sintesi proteica a livello di trascrizione
Enzimi costitutivi: prodotti continuamente
(es. enzimi per la degradazione del glucosio)
Enzimi inducibili: prodotti in presenza del substrato
(es. β–galattosidasi per lattosio)
3 meccanismi fondamentali (schema di Jacob e Monod)
1) INDUZIONE
2) REPRESSIONE
3) FEED BACK
Meccanismi di regolazione genica
I geni strutturali per la sintesi di enzimi di una data via metabolica sono sotto il
controllo di un gene operatore (l’insieme è chiamato operon). Tutto l’operon è a
sua volta sotto il controllo di un gene regolatore che codifica per una particolare
proteina (repressore) in grado di legarsi all’operatore, bloccandolo e bloccando
tutto l’operon. In assenza del substrato, l’operon è represso. Quando è presente,
il substrato si lega al repressore inattivandolo: il gene operatore si attiva ed attiva
tutto l’operon (Induzione da parte del substrato). Alcune volte, il repressore è
inizialmente inattivo; legandosi ad un prodotto finale della via metabolica si
attiva, bloccando il gene operatore (Repressione da parte del substrato). In altri
casi, il prodotto finale si lega ad uno degli enzimi della via metabolica (di solito
l’enzima 1) bloccandolo (Inibizione da feed-back)
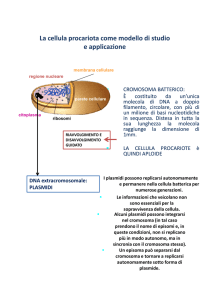
DNA extracromosomico: plasmidi
Elementi genetici accessori (DNA bicatenario circolare),
in grado di replicarsi autonomamente rispetto al
cromosoma batterico (repliconi)
Dimensioni minori del cromosoma
(1.000 - 200.000 coppie di basi)
Contengono un limitato numero di geni (meno di 30)
(autoreplicazione + geni caratteristici di ogni plasmide)
- informazione genetica non essenziale
(possono essere persi – “batteri curati”)
- presenti da 1 a centinaia (multicopia) di molecole
per cellula
Episomi
plasmidi in grado di "integrarsi" nel cromosoma batterico
Principali tipi di plasmidi
Fattore F (di fertilità) (94,5 kb)
- responsabile della formazione del pilo sessuale nella coniugazione
- responsabile del possibile trasferimento di materiale cromosomico da
F+ a F-
Plasmidi R
- responsabili della resistenza agli antibiotici (enzimi inattivanti)
- alcuni coniugativi (RTF), diffusione delle resistenze
Plasmidi Col
- produzione di batteriocine
Plasmidi di virulenza
- produzione di tossine o fattori di colonizzazione (fimbrie)
Plasmidi metabolici
- produzione di enzimi degradativi
- resistenza a metalli pesanti (Pb, Hg, As, ecc.)
Plasmidi
Mappa genetica di un plasmide
(pBR322)
Ogni plasmide contiene:
Ori, origine per la
replicazione autonoma;
eventuali geni che
codificano per il proprio
trasferimento attraverso
coniugazione (TF);
altri geni, che codificano
per resistenza agli
antibiotici (es. ampicillina–
Ap e tetraciclina–Tc),
batteriocine, tossine.
A volte sono presenti più
geni contemporaneamente.
Sigle e numeri della figura
indicano la posizione dei
siti di restrizione (quali
enzimi di restrizione
possono tagliare il plasmide
ed in quale posizione).
Enzimi di restrizione
- endonucleasi prodotte dai batteri per difendersi dai
batteriofagi
- tagliano il DNA bicatenario in corrispondenza di
sequenze specifiche, generando estremità coesive o
piatte (4, 5, 6, 7, 8, ecc. coppie di basi)
- utilizzati per trasferire geni su vettori (es. plasmidi),
rilegati dalle ligasi
DNA ricombinante Æ ingegneria genetica
Dpn II
Dde I
Eco RI
Alu I
Dra I
Pme I
Fse I
BsaB I
DNA extracromosomico:
Elementi trasponibili (geni mobili)
Elementi genetici accessori
(DNA bicatenario lineare)
molto diffusi sia tra i procarioti sia tra gli
eucarioti, non in grado di replicarsi
autonomamente, ma in grado di integrarsi nel
DNA
cromosomico
o
in
plasmidi
(trasposizione), responsabili di molti fenomeni
di variabilità genetica
Scoperti negli anni ’40 da Barbara McClintock
(premio Nobel 1983)
Sequenze di inserzione e Trasposoni
Sequenze di inserzione (IS)
Elementi trasponibili più semplici: brevi sequenze (750-1600 coppie
di basi, bp) contenenti il gene per la trasposasi (tnp), affiancato da
brevi sequenze (15-40 bp) ripetute e invertite, che permettono
l’inserzione nel cromosoma batterico
Struttura di una IS
Esempi di trasposoni composti A) Tn5; B) Tn3
Trasposoni composti (Tn)
Nel “core” si trovano
diversi tipi di geni
(resistenza agli antibiotici,
produzione di tossine,
ecc.) affiancati da IS, che
ne permettono la trasposizione
DNA extracromosomico: Batteriofagi
Virus dei batteri
il loro genoma può rappresentare un elemento
genetico accessorio
Responsabili della trasduzione e della
lisogenia
conversione lisogena: acquisizione di nuove
caratteristiche da parte del batterio;
ad es. produzione di tossine
Variabilità genetica
Evoluzione
Microrganismo ancestrale Ö Mutazioni spontanee
Ø
Variabilità genetica Ö Differenziazione
Ø
Maggiore adattabilità all'ambiente
(selezione naturale)
Nei procarioti, per la mancanza di riproduzione
sessuale, la variabilità genetica è dovuta a:
♦ Mutazioni
♦ Trasferimento intercellulare di materiale
genetico (ricombinazione)
Ricombinazione genetica
La ricombinazione è un processo unidirezionale
mediante il quale una porzione di materiale genetico
(DNA) è trasferita da un donatore ad una cellula
ricevente che la integra nel suo cromosoma, attraverso
processi di scambio di sequenze omologhe, con
acquisizione di nuove caratteristiche fenotipiche
• trasformazione
• trasduzione
• coniugazione
Trasformazione
Fine anni ’20: Griffith
Cellula donatrice lisata Ö DNA
libero Ö cellula ricevente
“competente”
La cellula è normalmente impermeabile
al DNA, a meno che non si trovi in uno
stato di competenza (formazione di pori
transitori
nella
membrana
che
permettono il passaggio di molecole di
DNA)
I frammenti di DNA devono avere certe
dimensioni (> 5×105 daltons)
Occorre elevata omologia tra il DNA di
donatore e ricevente Ö cellule della stessa
specie
I frammenti di DNA entrano, si appaiano
e possono ricombinarsi (a sinistra)
Lo stato di competenza può essere indotto
artificialmente (ad es. trattando la cellula
ricevente con ioni positivi, quali Ca++,
mediante elettroporazione, ecc.)
Trasformazione con plasmidi
La trasformazione è il
metodo più utilizzato
in ingegneria genetica
per introdurre
plasmidi
(o altri vettori),
contenenti DNA
ricombinante,
all’interno di cellule
ospiti
Trasduzione
Ricombinazione batterica mediata da batteriofagi (fagi)
Trasduzione generalizzata
Durante il ciclo litico il
DNA batterico può essere
degradato e sue parti
possono essere impacchettate per errore nelle teste
fagiche (particelle trasducenti)
Quando queste infettano
altre cellule, trasferiscono il
DNA (geni) batterico, che
può integrarsi con quello
del ricevente
Trasduzione specializzata o ristretta
Il più importante fago
temperato è il fago λ
(lambda)
Durante il ciclo lisogeno il
fago temperato si integra
nel cromosoma batterico
(profago) in un punto ben
definito (sito di integrazione).
Durante la fase di
escissione per errore può
portarsi dietro parti del
genoma batterico che
possono essere trasferite
e possono ricombinarsi
con il cromosoma del
ricevente.
Si definisce trasduzione
ristretta, perché solitamente solo pochi geni
vicini al punto di inserzione del profago possono
essere trasferiti.
Coniugazione
1946: Lederberg e Tatum
avviene attraverso il contatto fisico tra
2 cellule, la cellula F+ (donatrice)che
presenta F libero nel citoplasma e la
cellula F- (ricevente) priva di F
Plasmide F (94,5 kb)
contiene geni (geni tra) per il proprio
trasferimento e la formazione del pilo
sessuale
e sequenze IS che
consentono l’appaiamento a sequenze
omologhe sul cromosoma e la successiva
integrazione
OriT indica l’origine di replicazione ed il
sito in cui comincia il trasferimento
orientato nella coniugazione
Coniugazione
Fotografia al M.E. di una
cellula F+ (a destra) e di una
F- (a sinistra) collegate dal
pilo sessuale durante la
coniugazione
I plasmidi possono trasferirsi
per coniugazione, ma non si
tratta di ricombinazione
Responsabile della ricombinazione mediante coniugazione è
una Cellula HFR (ad alta
frequenza di ricombinazione)
in cui il plasmide F si trova
integrato
nel
cromosoma
batterico
Coniugazione
incrocio HFR × FVera ricombinazione
F si apre in corrispondenza di O e
comincia a trasferirsi trascinandosi
dietro i geni del cromosoma, che
possono ricombinarsi con i geni del
ricevente (questo rimane F-)
Il passaggio dell’intero cromosoma
richiede 90’.
Il pilo si rompe facilmente per cui
passano geni diversi a seconda del
tempo di contatto
è possibile la definizione della
mappa cromosomica
Coniugazione
incrocio F’ × F-
Vera ricombinazione
Quando F si libera dal cromosoma
(escissione) per errore può portare con
sé qualche gene cromosomico (F’)
Mutazioni
Alterazioni stabili ed ereditarie dell'informazione genetica, cioè alterazioni
della sequenza di basi di un gene che possono portare all’alterazione della
sequenza aminoacidica e all’eventuale alterazione della struttura e della
funzione
Mutazioni macrolesionali: delezione di più di una coppia di basi, duplicazione
di sequenze preeesistenti e alterazioni di ampi tratti del DNA in conseguenza
di fenomeni di inversione e traslocazione che possono verificarsi con
frequenze molto più elevate di quelle dovute a errore di duplicazione
Effetti delle mutazioni sul fenotipo
Retromutazione: La mutazione ripristina una funzione perduta con una
precedente mutazione
Di senso: La tripletta modificata codifica per un aminoacido (uguale o diverso
dall'originale)
Non senso: La tripletta modificata diventa un codone stop (UAA, UAG,
UGA) con conseguente interruzione della catena polipeptidica
Silente: La sostituzione di una base con un'altra nella tripletta porta alla
codifica dello stesso aminoacido (es. da UUU - Phe a UUC - Phe) e il prodotto
genico è inalterato, oppure la mutazione altera un aminoacido ma non la
funzionalità del prodotto genico
Leaky: La mutazione porta ad un prodotto genico alterato, ma ancora
funzionante anche se in modo ridotto (blocco metabolico incompleto)
Permissiva: La mutazione porta ad un prodotto genico alterato, in grado
di funzionare solo in particolari condizioni (mut. letale condizionale); es.
mutanti termosensibili
Letale: La mutazione porta ad un prodotto genico alterato, inattivo. Se esso
può essere surrogato da precursori forniti dall'esterno si ha la produzione di
mutanti auxotrofi.
Valutazione delle proprietà genotossiche
dei composti chimici: Test di Ames
(Individuazione di agenti mutageni)
Si utilizzano ceppi
di
Salmonella
mutanti nel gene per
l’istidina (HIS-), che
crescono solo in
terreno contenente
istidina.
In ognuno di tali
ceppi si possono
verificare
retromutazioni spontanee
con
una
certa
frequenza (HIS+).
Valutazione delle proprietà genotossiche
dei composti chimici: Test di Ames
(Individuazione di agenti mutageni)
Se ad un terreno privo di istidina viene aggiunto un mutageno, la
frequenza di retromutazione aumenta significativamente,
consentendo la crescita di un numero di colonie maggiore.
In assenza del mutageno, si produce un certo numero di colonie
(retromutanti spontanei). Se, in presenza di diverse concentrazione
del composto chimico, si osserva un aumento dose-dipendente del
numero delle colonie (di almeno 2-3 volte), il composto è giudicato
mutageno.
Dal momento che alcuni composti chimici non sono mutageni
(potenzialmente cancerogeni) in quanto tali, ma possono
diventarlo se metabolizzati all’interno dell’organismo (nell’uomo e
negli animali soprattutto nel fegato), il saggio è stato perfezionato
aggiungendo alla miscela un sistema di metabolizzazione esogeno,
costituito da un omogenato (microsomi) di fegato di ratto.

![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)









