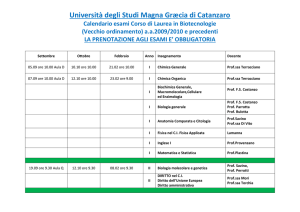
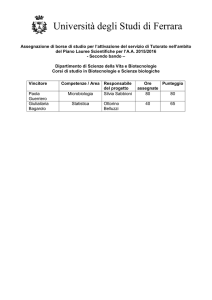
Il metabolismo dell’RNA
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
I vari tipi di RNA
• Il filamento di DNA che dirige la sintesi dello
mRNA è chiamato filamento stampo o
filamento antisenso.
• L’altro filamento che ha sequenza identica a
quella dello mRNA è chiamato filamento
codificante o filamento senso.
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
Il messaggero è tradotto in proteine
Ogni mRNA è associato a diversi
ribosomi: il complesso prende il nome di
poliribosoma o polisoma. Ogni
ribosoma nel polisoma sintetizza
indipendentemente un polipeptide, che
cresce di dimensioni man mano che il
ribosoma si muove dall’estremità 5’
all’estremità 3’.
• Per esempio, la globina è sintetizzata da un gruppo
di 5 ribosomi per molecola di mRNA.
• I ribosomi appaiono come oggetti sferici di 7 nm
di diametro, connessi da un filamento di mRNA.
• I ribosomi sono posizionati in diversi punti del
messaggero
– quelli all’estremità 5’ hanno appena iniziato la
sintesi della globina
– quelli all’estremità 3’ hanno quasi completato la
proteina.
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
Il messaggero è tradotto in proteine
I ribosomi liberi formano un pool,
da cui vengono reclutati quelli
ingaggiati nella sintesi proteica. Il
numero di ribosomi su un
messaggero dipende dalla
efficienza del mRNA nel reclutarli.
• I 19.000-20.000 ribosomi di un batterio
rappresentano circa un quarto della sua
massa.
• Il numero di tRNA eccede quello dei
ribosomi di circa 10 volte.
• Si stima che ci siano circa 1.500 molecole di
mRNA in un batterio (difficile da calcolare,
il messaggero dei batteri è molto instabile).
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
Il messaggero dei procarioti
• L’RNA messaggero ha la stessa funzione in
procarioti ed eucarioti, ma ci sono differenze
importanti nella sintesi e nella struttura del mRNA
nei due sistemi.
• Nei batteri, il messaggero è trascritto e tradotto nello
stesso compartimento cellulare, ed i due processi
sono collegati così strettamente che avvengono
contemporaneamente.
• La trascrizione del messaggero avviene a circa
40 nucleotidi/sec a 37 °C
– ci vogliono circa 2 min a trascrivere un mRNA di
5000 nucleotidi.
• La sintesi delle proteine avviene a circa 15 aa/sec
– 45 nucleotidi/sec di mRNA tradotto.
• La trascrizione e la traduzione viaggiano alla stessa
velocità.
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
Il messaggero dei procarioti
Trascrizione e traduzione nei batteri possono essere visualizzate tramite la microscopia
elettronica.
• Nella fotografia, molti
mRNA sono trascritti
simultaneamente, e ogni
mRNA è tradotto da molti
ribosomi.
• Un RNA la cui sintesi non
è stata ancora completata
si chiama RNA nascente.
• Gli mRNA batterici sono
policistronici.
–
–
–
–
regione codificante
leader (5’UTR)
trailer (3’ UTR)
regioni intercistroniche (0-30 nt)
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
Il messaggero degli eucarioti
Entrambe le estremità
del messaggero degli
eucarioti sono
modificate dalla
aggiunta di nucleotidi
successivamente alla
trascrizione.
• L’estremità 5’ è modificata dall’aggiunta di un
cappuccio, che avviene immediatamente.
• L’estremità 3’ è generata tramite taglio del
trascritto primario anzichè terminazione della
trascrizione.
• Immediatamente dopo il taglio, l’estremità 3’ è
modificata dalla aggiunta di una serie di nucleotidi
di acido adenilico: acido poliadenilico o poli(A).
• Solo dopo questa serie di modificazioni il
messaggero è pronto per essere trasportato nel
citoplasma dove è relativamente stabile, con una
emivita di 4-24 ore.
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
Il Cappuccio
La trascrizione comincia con un nucleoside trifosfato (tipicamente A o G), di modo
che il trascritto primario ha al 5’ struttura pppApNpNpNp….
• In seguito, l’enzima
guanililtransferasi catalizza
l’aggiunta di una G con un
legame 5’-5’ (orientazione
inversa rispetto agli altri
nucleotidi).
• La guanina-7-metiltransferasi
aggiunge un metile in posizione 7
– cappuccio tipo 0, presente negli
eucarioti unicellulari.
• La 2’-O-metiltransferasi
aggiunge un alro metile
– cappuccio tipo 1, presente nella
maggior parte degli eucarioti
Quando un posizione 2 (ex 1) c’è una Adenina, può essere aggiunto un metile in
posizione 6 dopo l’aggiunta del metile in posizione 2’-O.
Infine, se sono state efettuate tutte le modificazioni precedenti, può essere aggiunto un
metile in 2’-O della terza base: cappuccio tipo 2, 10-15% degli eucarioti superiori.
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
L’estremità 3’ del messaggero
Non è chiaro se esista un sito di terminazione per la RNA polimerasi II. Sono state
evidenziate terminazioni non specifiche più di 1000 paia di basi a valle della estremità
3’ del messaggero maturo.
• La polimerasi trascrive oltre l’estremità 3’
del messaggero maturo.
• Un complesso enzimatico esegue il taglio e
la successiva poliadenilazione del trascritto.
• La poliadenilazione stabilizza il trascritto
all’estremità 3’.
• L’estremità 5’ del trascritto è già
stabilizzata dalla presenza del cappuccio.
• La polimerasi prosegue oltre il sito di taglio
e poliadenilazione, ma l’estremità 5’
generata dal taglio non è protetta.
• Di conseguenza, il resto del trascritto
primario è degradato molto rapidamente
– è difficile determinare cosa succede oltre il
sito di taglio e poliadenilazione.
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
La poliadenilazione
•
Nei messaggeri degli eucarioti superiori (non nel lievito)
è presente la sequenza AAUAAA 11-30 nt a monte della
coda di poli(A)
– saudi α-talassemia.
•
•
•
•
•
•
•
Un fattore multisubunità, chiamto CstF, lega una
sequenza ricca in G e U a valle del sito di taglio.
Un fattore (composto da 4 subunità) chiamato CPSF
(Cleavage and Polyadenylation Specificity Factor)
riconosce la sequenza AAUAAA quando CstF ha legato
la regione ricca in G e U.
Una endonucleasi, composta dalle subunità CFI e CFII
taglia il trascritto.
La poliadenilato polimerasi sintetizza la coda di poli(A)
Inizialmente vengono aggiunti circa 10 nucleotidi.
In seguito, la PABP (poly(A)-Binding Protein) lega la
coda di poli(A) nascente.
Questo stimola la poli(A) polimerasi ad aggiungere acido
adenilico fino ad una lunghezza di circa 200 nucleotidi.
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
Il messaggero
degli eucarioti
Trascritto primario
La vera funzione delle
proteine che legano il
trascritto primario resta
sconosciuta.
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
Taglio e saldatura dell’RNA: splicing
Donor splice site
Acceptor splice site
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
La rimozione dell’introne
• Struttura a cappio.
• Punto di ramificazione
– si trova 18-40 nucleotidi a monte del 3’ splice site.
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
La trans-esterificazione
Adenine
G
A
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
La struttura degli intermedi di splicing
Posizione 5’
Posizione 4’
Posizione 3’
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
Gli snRNA, le snRNP e lo splicing
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
•
•
•
•
Lo splicing
Assemblaggio dello spliceosoma
U1 si stacca; U5 si sposta.
U6 si stacca da U4 (sarà rilasciato)
e lega U2 formando in centro
catalitico).
U2 e U6 catalizzano la transesterificazione
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
Eucarioti
La maturazione
dell’RNA ribosomale
Procarioti
Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie