Testi x pdf
Nucleotidi, acidi nucleici
I Nucleotidi Hanno Tre Componenti
Nucleotidi (1)
Un Nucleotide è una
molecola costituita da
un composto ad anello
contenente azoto (N)
legato ad uno zucchero
con 5 atomi di carbono.
Lo zucchero può essere
o un riboso o un
desossiribosio ed è a
sua volta collegato ad
uno o più gruppi
fosfato.
Nucleotidi (2)
Gli anelli che contengono N sono chiamati “basi” per
ragioni storiche: in condizioni acide ciascuno di essi
può legare un protone (H+) e quindi aumentare la
concentrazione di ioni OH- in soluzione acquosa.
Vi è una forte somiglianza chimica fra le diverse basi
azotate:
La Citosina (C), la Timina (T) e l’Uracile (U) sono chiamate
pirimidine in quanto tutte derivano da un anello a sei
atomi detto anello pirimidinico.
La Guanina (G) e l’Adenina (A) sono composti purinici ed
hanno un secondo anello a 5 atomi fuso con l’anello a 6
atomi.
Basi azotate presenti
negli acidi nucleici
Negli acidi nucleici e nei
nucleotidi, l’azoto in
posizione 9 delle purine
e l’azoto in posizione 1
delle pirimidine (rosso)
sono legati al carbono 1’
del ribosio o del
desossiribosio.
http://www.ncbi.nlm.nih.gov/books/NBK21514/figure/A805/?report
=objectonly
I nucleosidi e i loro mono-, di- e tri-fosfati
Base
DNA
Nucleoside
Nucleotidi
Adenina (A)
Deossiadenosina
dAMP
dADP
dATP
Guanina (G)
Deossiguanosina
dGMP
dGDP
dGTP
Citosina (C)
Deossicitidina
dCMP
dCDP
dCTP
Timina (T)
Deossitimidina
dTMP
dTDP
dTTP
Adenina (A)
Adenosina
AMP
ADP
ATP
Guanina (G)
Guanosina
GMP
GDP
GTP
Citosina (C)
Citosina
CMP
CDP
CTP
Uracile (U)
Uridina
UMP
UDP
UTP
RNA
Nucleotidi (4)
• Oltre ad essere componenti degli acidi nucleici (DNA
e RNA) i nucleotidi possono svolgere altre importanti
funzioni. Es.
– La maggior parte dell’energia utilizzata dagli esseri viventi
deriva dall’adenosina trifosfato (ATP)
– La guanosina trifosfato (GTP) è un nucleotide si lega d un
gran numero di proteine (“proteine G”) e agisce da
interrutore per attivarle.
• Proteine G monomeriche
• Proteine G trimeriche
Nucleotidi : Altre funzioni
• I nucleotidi possono giocare altri ruoli
importanti:
– Il ribonucleotide ATP funge da trasduttore di energia
in molte reazioni biochimiche.
– Il ribonucleotide GTP fornisce l’energia per la sintesi
proteica.
– Il cAMP (AMP ciclico) è un ribonucleotide speciale
que è essenziale per l’azione ormonale e per il
trasferimento di informazione nel sistema nervoso.
“Proteine G”
Funzionano da interrutori
molecolari. Quando legano la
guanosina trifosfato (GTP) si
attivano (“on”) e quando legano
la guanosina difosfato si
disattivano (“off”).
Le proteine G regolano enzimi
metabolici, canali ionici,
trasportatori di membrana, e
altre componenti delle attività
cellulari, controllando la
trascrizione, motilità,
contrattilità e secrezione che, a
loro volta, regolano funzioni
sistemiche quali lo sviluppo
embrionale, l’apprendimento e
la memoria e l’omeostasi.
GTP
Caratteristiche Distintive del DNA e del RNA
DNA RNA Proteina
• Il DNA è una molecola informativa. L’informazione è
immagazzinata nell’ordine in cui sono disposte le
sue quattro basi differenti.
• Questo ordine è trasferito alle molecole di RNA, che
sono usate per dirigere l’ordine degli amminoacidi
delle proteine.
Acidi Nucleici : Il RNA può essere catalitico
• Alcune molecole di RNA dette ribozimi possono
fungere da catalizzatori.
• La scoperta dei ribozimi ha fornito una soluzione
per la questione su chi è comparso prima
quando la vita è iniziata, le proteine o gli acidi
nucleici?
• Dato che il RNA può essere sia informazionale
che catalitico, esso potrebbe avere funzionato
come catalizzatore sia per la sua stessa
replicazione che per quella delle proteine.
Le cellule contengono istruzioni che:
Specificano STRUTTURA
Dettano FUNZIONE
Regolano ATTIVITA’
Le istruzioni vengono trasmesse fedelmente alle
cellule figlie
L’informazione ereditaria é trasmessa sotto forma di
unità distinte, i GENI costituiti da DNA.
L’informazione del DNA passa:
fra generazioni di cellule: REPLICAZIONE DEL DNA
all’interno di ogni cellula: TRASCRIZIONE +
TRADUZIONE
Nucleotidi (4)
Sia il DNA che l’RNA vengono sintetizzati a
partire da nucleosidi trifosfati:
Per il DNA: dATP, dCTP, dGTP e dTTP
Per il RNA: ATP, CTP, GTP e UTP.
In entrambi i casi, dal momento in cui ogni
nucleoside viene legato, i secondo e terzo
gruppi fosfato vengono rimossi (idrolizzati).
RNA
Da un punto di vista chimico, l’RNA è molto simile al DNA.
Tuttavia, ci sono alcune differenze principali:
o L’RNA usa lo zucchero RIBOSIO invece del Desossiribosio
nel suo scheletro (“backbone”).
o L’RNA usa la base URACILE (U) invece della Timina (T).
Anche U è complementare ad A.
o L’RNA tende ad essere a singolo filamento (“single
strand”), ma alcune regioni possono ripiegarsi formando
zone a doppio filamento.
Differenze funzionali tra RNA e DNA
o Il DNA ha una singola funzione; l’ RNA ha molteplici
funzioni
Esempi di tipi di RNA: tRNA, mRNA, rRNA.
Differenze Chimiche fra DNA e RNA (2)
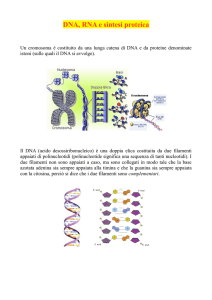
Sia il RNA che il DNA formano lunghe catene
polinucleotidiche, non ramificate, in cui diverse basi puriniche
o pirimidiniche sono riunite mediante legami N-glicosidici ad
un’impalcatura strutturale di zuccheri-fosfato.
Le catene hanno una polarità. La sequenza di un acido
nucleico viene per convenzione letta da 5’ a 3’. Ad esempio, la
sequenza della molecola di RNA qui rappresentata é AUGC,
mentre quella della molecola di DNA é ATGC.
La sequenza di basi porta in se l’informazione, ossia, la
sequenza ATGC comporta un’informazione diversa di quella di
AGCT nonostante le stesse basi siano coinvolte.
Conseguenze della struttura chimica del RNA/DNA
L’impalcatura strutturale del DNA é più stabile, specialmente
in condizione alcalina. Il gruppo OH nella posizione 2’ del RNA
può formare intermediari 2’-3’ fosfodiestere in condizioni
basiche, che si rompono, dando una miscela di nucleosidi 2’ e
3’ monofosfati. Perciò il polinucleotide RNA é instabile.
Il 2’ desossiribosio permette allo zucchero di assumere una
conformazione a minore energia nell’impalcatura del DNA.
Ciò aiuta ad aumentare la stabilità dei polinucleotidi.
La deaminazione della Citidina a Uridina può essere
identificata nel DNA, ma non nel RNA, perchè la
deaminazione della citidina nel DNA porta a Uridina non a
Timidina. Le basi Uridina nel DNA vengono rimosse da un
sottogruppo di enzimi di riparo e sostituita con basi citidina.
Regola di
Chargaff
James Watson & Francis Crick
http://www.achievement.org/autodoc/
photocredit/achievers/wat0-013
Conclusioni di Watson & Crick sulla struttura del DNA
(1953)
•Tratte
dai dati cristallografici di Rosalind Franklin e
Maurice Wilkins
Due catene polinucleotidiche elicoidali si avvolgono
attorno ad un asse centrale con un diametro di 20
angstroms (10-10 m).
Le due catene sono antiparallele (scorrono in direzioni
opposte).
Le impalcature di zucchero-fosfato rimangono all’esterno
della struttura.
Le basi azotate sono perpendicolari all’asse dell’elica e
separate da 3.4 angstroms.
La struttura elicoidale si ripete ogni 34 angstroms, e
quindi ci sono 10 basi per giro dell’elica.
... una modesta proposta...
It has not escaped our notice that the specific pairing we have
postulated immediately suggests a possible copying mechanism for the
genetic material.”
-Watson & Crick, “A Structure for Deoxyribose
Nucleic Acid,” Nature 1953.
“Non è sfuggito alla nostra attenzione che l’appaiamento
specifico che abbiamo postulato suggerisce
immediatamente un possibile meccanismo di copiatura
per il materiale genetico”
DNA polimerasi
Tutti le DNA polimerasi note condividono due proprietà
fondamentali che hanno implicazioni critiche per la
replicazione del DNA:
Tutte le polimerasi sintetizzano il DNA soltanto nel verso 5’ a
3’, aggiungendo un dNTP al gruppo idrossilico della catena in
crescita.
Le DNA polimerasi possono aggiungere un nuovo
desorriribonucleotide soltanto ad un “primer” (innesco)
preformato che è legato mediante ponti di idrogeno al
templato; non sono in grado di iniziare la sintesi del DNA de
novo catalizzando la polimerizzazione di dNTPs liberi.
Da questo punto di vista, le DNA polimerasi differiscono
dalle RNA polimerasi, che possono inziare la sintesi di un
nuovo filamento di RNA in assenza di un primer.
TIPI PRINCIPALI DI RNA
PRODOTTI NELLE CELLULE
Tipo di RNA
mRNA
rRNA
tRNA
snRNA
snoRNA
Funzione
RNA messaggeri, codificano per
proteine
RNA ribosomiali, formano la
struttura di base dei ribosomi e
catalizzano la sintesi proteica
RNA transfer, fondamentali per la
sintesi proteica come adattatori fra
mRNA e aminoacidi
piccoli RNA nucleari, agiscono in una
varietà di processi nucleari, compreso lo
splicing del pre-mRNA
piccoli RNA nucleolari, usati per
processare e modificare chimicamente gli
rRNA
Altri RNA non codificanti
Agiscono in diversi processi cellulari,
compreso sintesi dei telomeri, disattivazione
del cromosoma X, trasporto di proteine
nell’ER, regolazione genica (microRNA)*.
Funzione e struttura del RNA
La struttura terziaria del RNA é simile a quella del
DNA, ma ci sono diverse differenze importanti:
Il RNA di solito forma coppie di basi intramolecolari
L’informazione trasportata dal RNA non é ridondante
perchè queste coppie di basi sono intramolecolari.
I solchi maggiore e minore sono molto meno pronunciati.
Tutti i ruoli svolti dal RNA (strutturale, adattatore
informazionale, trasferimento di informazione) sono
coinvolti nella decodificazione trasportata dal DNA.
RNA polimerasi nelle cellule eucariotiche
Tipo di polimerasi
Geni trascritti
RNA polimerasi I
geni degli rRNA 5.8S, 18S e 28S
RNA polimerasi II
tutti i geni che codificano
proteine, più geni di snoRNA e
alcuni geni di snRNA
RNA polimerasi III
geni dei tRNA, geni di rRNA 5S,
alcuni geni di snRNA e geni
per altri piccoli RNA
RNA polimerasi mitocondriale*
geni mitocondriali
RNA polimerasi dei cloroplasti*
geni dei cloroplasti
*Le RNA polimerasi mitocondriale e dei cloroplasti sono simili agli enzimi batterici
TERMINOLOGIA DEL PROCESSAMENTO
DEL DNA E DEL RNA
Termine
Significato letterale
Replicazione
Fare un duplicato
identico
Trascrizione
Copiare informazione
da un supporto ad un
altro, come avviene ad
es. quando si passa da
un nastro audio ad un
testo scritto
Prendere informazione
da una lingua e
convertirla in un’altra
lingua
Traduzione
Processo in cui sono
coinvolti gli acidi
nucleici
Usare una molecola di
DNA come riferimento
per ottenere due copie
di molecole figlie
identiche
Copiare la sequenza di
basi di una molecola di
DNA sotto forma di
sequenza di basi di una
molecola di RNA
Prendere la sequenza di
codoni del mRNA e
convertirla in una
catena di aminoacidi di
una proteina
Maturazione del RNA
TRASCRITTO primario: molecola di RNA appena prodotta per
trascrizione del DNA.
MATURAZIONE: Modificazioni chimiche necessarie per
produrre un mRNA funzionale a partire dal trascritto primario
(precursore):
RIMOZIONE DI REGIONI DEL TRASCRITTO PRIMARIO (es.
splicing del mRNA).
AGGIUNTA DI MODIFICAZIONI DI SPECIFICI NUCLEOTIDI
(particolarmente negli tRNA).
Associazione con proteine.
Passaggio dal nucleo al citoplasma
STRUTTURA DEI GENI DEGLI EUCARIOTI
La maggior parte dei geni degli eucarioti contiene segmenti di
sequenze codificanti (esoni) interrotte da sequenze noncodificanti (introni).
Le sequenze di spaziatura (“spacers”) sono lunghe sequenze
di DNA non codificanti che rimangono fra i geni.
Cooper – The Cell, a Molecular Approach (2nd ed.)
mRNA (1)
Il RNA messaggero o mRNA é una copia
dell’informazione trasportata da un
gene sul DNA.
Il ruolo del mRNA é quello di trasportare
l’informazione contenuta nel DNA fino
all’apparato di traduzione.
Il mRNA é eterogeneo in dimensioni e
sequenza.
mRNA (3)
Il mRNA non viene sintetizzato direttamente nella cellula
eucariotica.
Esso é trascritto nel nucleo sotto forma di RNA nucleare
eterogeneo (“heterogeneous nuclear“ RNA, hnRNA).
Il hnRNA contiene introni ed esoni.
Gl introni sono rimossi mediante “splicing” del RNA, lasciando
gli esoni, che contengono l’informazione, riuniti fra di loro.
In alcuni casi, singoli nucleotidi possono venire aggiunti nel
mezzo di una sequenza di mRNA mediante un processo detto
di “editting” del RNA.
Subunità dei ribosomi
In tutte le cellule, ogni ribosoma consiste di una
subunità grande e una piccola.
Le due subunità contengono rRNAs di diversa
lunghezza e un insieme diverso di proteine.
Tutti i ribosomi contengono due molecole principali di
rRNA (rosso scuro) – 23S e 16S rRNA nei batteri, 28S e
18S rRNA negli eucarioti – e uno o due piccoli rRNA
(rosso chiaro).
Le proteine sono disegnate L1, L2, ecc e S1, S2, ecc., a
seconda che siano presenti nella subunità grande
(“large”) o piccola (“small»).
http://www.ncbi.nlm.nih.gov/books/NBK21603/figure/A888/?report=objectonly
PROCARIOTI
EUCARIOTI
La molecola di tRNA viene
sintetizzata in due parti, Il corpo
del tRNA é trascritto da un gene
tRNA. Il braccio accettore
(“acceptor stem”) é lo stesso
per tutte le molecole di tRNA ed
é aggiunto dopo che il corpo é
stato sintetizzato. Esso viene
sostituito spesso durante il
tempo di vita di una molecola di
tRNA.
Il braccio accettore è il sito che
trasporta uno specifico
aminoacido, che é stato
attaccato da una amino-aciltRNA sintasi.
L’anticodone legge
l’informazione in una sequenza
del mRNA mediante
accoppiamento di basi.
STRUTTURA DEGLI RNA
A FORMA TRIDIMENSIONALE COMPLESSA
(es. rRNA, tRNA) (1)
Sia gli rRNA che i tRNA devono la loro attività alle loro complesse
strutture secondaria e terziaria.
Diversamente dal DNA che ha struttura a doppia elica relativamente
definita indipendentemente dalla sua origine, gli RNA si ripiegano in
complesse forme tridimensionali, notoriamente diverse da un tipo di RNA
all’altro.
Perciò, come le proteine, gli RNA sono in grado di svolgere molte funzioni,
perchè possono assumere un gran varietà di forme.
Come per le proteine, il ripiegamento degli RNA segue certe regole.
Durante il ripiegamento delle catene proteiche che lavorano in ambiente
acquoso i residui idrofobici vengono portati all’internoNell’RNA il ripiegamento dipende dalla formazione di regioni che hanno
coppie di basi complementari.
Le regioni di basi appaiate formano tipicamente degli “steli” a doppio
filamento (e a doppia elica), connessi ad “anse” a filamento singolo.
STRUTTURA DEGLI RNA
A FORMA TRIDIMENSIONALE COMPLESSA
(es. rRNA, tRNA) (2)
A differenze del DNA, costituito unicamente dalle coppie di
basi A-T e G-C, il RNA contiene spesso coppie di basi non
convenzionali e basi azotate modificate.
Queste regioni peculiari spesso fungono da siti di
riconoscimento per proteine ed altri RNA
Promuovono il ripiegamento del RNA
Aiutano a stabilizzare la struttura della molecola.
L’appaiamento delle basi fra le diverse molecole di RNA svolge
un ruolo cruciale nella gran parte delle attività in cui sono
coinvolti gli RNA.
L’RNA si può ripiegare in strutture specifiche. L’RNA è in gran parte a singolo filamento, ma
spesso contiene brevi tratti di nucleotidi che possono formare coppie di basi convenzionali con
sequenza complementari presenti altrove sulla stessa molecola. Queste interazioni, insieme ad
ulteriori interazioni di appaiamento “non convenzionale” di basi, permettono ad una molecola
di RNA di ripiegarsi in una struttura tridimensionale che è determinata dalla sua sequenza di
nucleotidi. (A) Diagramma di una struttura ripiegata di RNA che mostra soltanto interazioni di
appaiamento convenzionale; (B) struttura con interazioni di accoppiamento convenzionale
(rosso) e non convenzionale (verde); (C) struttura di un RNA reale, una porzione di un introne di
gruppo 1. Ciascuna interazione di accoppiamento convenzionale é indicata da un “piolo” nella
doppia elica. Le basi in altre configurazioni sono indicate da pioli spezzati.
RNA transfer (tRNA)
Il tRNA é la molecola portatrice di informazione con funzione
di adattatore.
Rappresenta l’interfaccia diretta fra la sequenza di aminoacidi
di una proteina e l’informazione sul DNA. Perciò essa
decodifica l’informazione sul DNA.
Ci sono più di 20 diverse molecole di tRNA. Tutte hanno da 75
a 95 nucleotidi.
Tutti i tRNA di tutti gli organismi hanno una struttura similare:
in verità, un tRNA umano può funzionare in un lievito.
Nelle molecole di tRNA ci sono 4 braccia (accettore, braccio D,
braccio T pseudouridina C, braccio anticodone) e 3 “loops.”
(loop D, loop T e loop anticodone).
Talvolta le molecole di tRNA hannno un loop extra o variabile
(uno é illustrato in giallo nella figura).
Struttura dei tRNA
Struttura primaria e secondaria (1)
Tutti i tRNA hanno una sequenza simile di 73 a 93
nucleotidi.
L’estremità 3’ finisce sempre con la sequenza CCA, in
cui il gruppo idrossilico 3’ del ribosio della A
terminale é il punto di legame covalente per
l’aminoacido.
Essi contengono un certo numero (7-15%) di basi
uniche/ o modificate. Queste vengono modificate
post-trascrizionalmente dopo la sintesi, mediante
l’enzima RNA polimerasi.
Struttura dei tRNA
Struttura primaria e
secondaria (2)
In particolare, l’adenosina
(A) nella prima o 5’
posizione dell’anticodone
(corrispondente alla terza
o posizione 3’ del codone)
é sempre modificata in
inosina (I) che manca del
gruppo aminico (-NH2)
nell’anello purinico.
L’inosina può appaiarsi con
A, U o C e perciò spiega
molta della degenerazione
del Codice Genetico
(Teoria di Wobble).
Adenosina
Struttura dei tRNA
Struttura Terziaria del tRNA (1)
La struttura terziaria del tRNA é meglio descritta
come una forma compatta a “L”.
L’anticodone é un loop a singola elica all’estremità
della Figura, che ulteriormente si appaierà con la
tripletta codone.
L’aminoacido é legato alla A terminale sulla parte
superiore destra.
I siti attivi (anticodone e aminoacido) sono separati
al massimo.
Struttura dei tRNA
Struttura Terziaria del tRNA (2)
Come succede con le
proteine, la struttura
terziaria é dettata dalla
sequenza primaria.
La struttura terziaria é
stabilizzata da appaiamento
tra basi e accatastamento di
basi.
Due aree (braccio
anticodone e braccio
accettore) formano una
doppia elica.
CODICE GENETICO
Codice mediante il quale la sequenza nucleotidica di
una molecola di DNA o di RNA specifica la sequenza
amminoacidica di un polipeptide.
Consiste di codoni a tre nucleotidi che specificano un
particolare amminoacido oppure dicono al ribosoma
di fermare la traduzione e rilasciare il polipeptide.
Con poche eccezioni, tutti gli organismi viventi
utilizzano lo stesso codice.
(Mathews et al., Biochimica)
Codiced Genetico (segue)
Un gene
una catena polipeptidica
Corrispondenza lineare solo nei PROCARIOTI.
EUCARIOTI: sequenze codificanti (esoni) +
sequenza non codificanti (introni)
ATTENZIONE: alcuni geni NON codificano per
proteine (mediante un mRNA), codificano per
gli altri RNA: tRNA, rRNA, snRNA, snoRNA, ecc.
Codiced Genetico (segue)
CODICE GENETICO detto «DEGENERATO» O
«RIDONDANTE»:
Un singolo amminoacido può essere specificato da
più di una tripletta di nucleotidi.
Non significa malfunzionalità (ambiguità).
Aumenta l’adattabilità del sistema di codificazione.
Il codice genetico non è sovrapposto:
– Ogni nucleotide è parte di una sola tripletta.
Decodificazione di una
molecola di mRNA
Ogni aminoacido addizionato
all’estremità crescente della catena
polipeptidica viene selezionato
mediante appaiamento
complementare di basi fra
l’anticodone nel tRNA a cui é legato
e il codone successivo nella catena
del mRNA
One gene --> one protein
Same language/monomers
Different language/monomers
Genetic code
• Triplet code
Three basepairs specify one
amino acid = codon
Genetic code
• Triplet code
DNA Template strand =
complementary to mRNA
Coding strand =
nontemplate strand =
similar sequence to mRNA
Genetic code
• Triplet code
DNA Template strand =
complementary to mRNA
Coding strand =
nontemplate strand =
similar sequence to mRNA
Uracil used instead of Thymine
Genetic code
• Triplet code
• Nonoverlapping
Genetic code
• Triplet code
• Nonoverlapping
• Degenerate
- 4 nucleotides -- > 20 amino acids doublet
code --> 42 --> 16 combinations
triplet code --> 43 --> 64 combinations
Genetic code
• Triplet code
• Nonoverlapping
• Degenerate
Each amino acid specified by more than one
nucleotide triplet (codon)
61 codons code for amino acids
triplet code --> 43
--> 64 combinations
61 codons code for amino acids
•
•
•
•
AUG = Met = start
UAG
UAA
Stop
UGA
Genetic code
Genetic code
«WOBBLE» NEL CODICE GENETICO
(TENTENAMENTO) (1)
D’accordo con il codice genetico, una cellula
avrebbe bisogno di tRNA con 61 diversi
anticodoni per complementare i 61 codoni
disponibili.
Tuttavia, dovuto alla degenerazione del codice
genetico, la terza base è meno discriminatoria
per l’aminoacido delle altre due basi.
«WOBBLE» NEL CODICE GENETICO
(TENTENAMENTO) (3)
I tRNA inoltre contengono una serie nucleotidi
modificati.
Uno di questi nucleotidi è l’inosina (I).
Se il tRNA contiene una Inosina nell’anticodone in
posizione “wobble”, questo tRNA può leggere
codoni che hanno A, U oppure C nella terza
posizione.