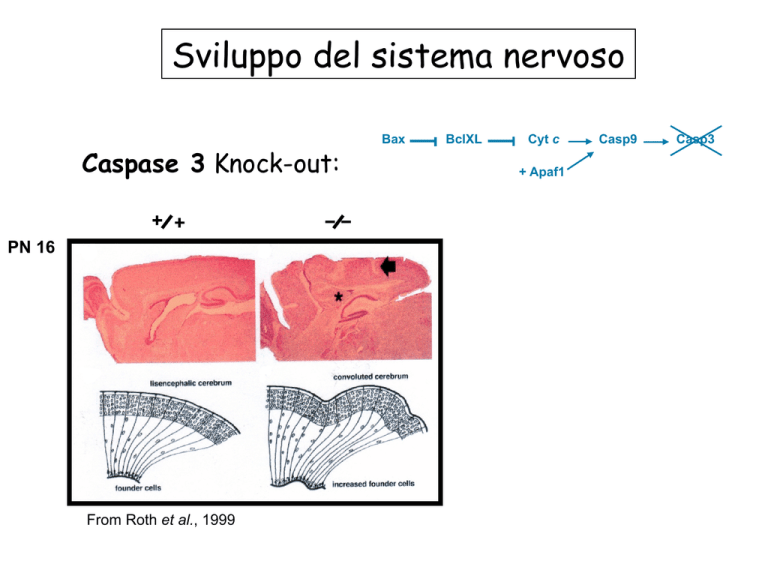
Sviluppo del sistema nervoso
Caspase 3 Knock-out:
PN 16
From Roth et al., 1999
Bax
BclXL
Cyt c
+ Apaf1
Casp9
Casp3
Sviluppo del sistema nervoso
BclXL Knock-out:
Bax Knock-out:
From Kuan et al., 2000
Bax
BclXL
Cyt c
+ Apaf1
From White et al., 1998
Casp9
Casp3
Bax
BclXL
Cyt c
+ Apaf1
Casp9
Casp3
Livelli diversi di proteine apoptotiche determinano
una diversa risposta agli stimoli apoptotici:
ES. Apaf1 in Cellule progenitrici vs differenziate
From Schafer et al., 2006
L’aggiunta di cyt c non induce l’attivazione della casp 3 in neuroni maturi
La molecola chiave che determina questa differenza è APAF1, presente
a diversi livelli nei precursori neuronali e nei neuroni differenziati
From Johnson et al., 2007
Apoptosi
e
danno al DNA
Quando la cellula ha il DNA danneggiato:
1. Attua meccanismi di riparo
2. Se il danno è irreparabile o il sistema
di riparo non funziona la cellula va in apoptosi
Esistono vari sistemi di riparo del danno
al DNA, a seconda del tipo di danno.
Se il danno è caratterizzato dalla rottura del
doppio filamento (DSB), due sono i processi
che possono essere attivati:
1. Ricombinazione omologa (HR)
2. Ricombinazione non omologa (NHEJ)
(il pathway più usato poichè non prevede
la presenza del cromatide fratello e può
essere utilizzato durante tutto il ciclo cellulare )
p53:
il “guardiano del genoma”
p53 subisce modificazioni
post-traduzionali (tipo fosforilazione)
che ne aumentano i livelli e
l’attività trascrizionale
p53 attiva la trascrizione
di vari geni apoptotici (Noxa
PUMA, Bax, Bid, Apaf1)
danno al DNA
(chemioterapici, radiazioni, ROS)
Fosforilazione
di H2AX
HR
NHEJ
Ku70/86:
la proteina di riparo del danno al DNA (NHEJ)
Ku70/86 reprime l’espressione
del gene Apaf1
From De Zio et al., 2011
Ku70 citosolico inibisce la traslocazione
di Bax nel mitocondrio
From Amsel et al., 2008
Ku70 citosolico inibisce l’attivazione della
Caspasi 8 stabilizzando FLIP
From Kerr et al., 2012
Problem solving:
a) il DNA è danneggiato in cellule precursori
neuronali….
b) osservo morte cellulare apoptotica solo molto
tempo dopo….
QUALI SONO I MECCANISMI CHE
DETERMINANO TALE DESTINO CELLULARE?
E QUALI LE MOLECOLE COINVOLTE?
A seguito dell’induzione del danno al DNA:
I° fase: risposta di sopravvivenza:
Ku70 si accumula nel nucleo
Ku70/86 reprime la trascrizione di Apaf1
From De Zio et al., 2011
I° fase: risposta di sopravvivenza
Ku70/86 reprime l’apoptosi
II° fase: morte cellulare apoptotica
From De Zio et al., 2011
II° fase: morte cellulare apoptotica
La modulazione negativa di Ku70/86 induce apoptosi
a tempi più brevi
Referenze reviews:
1. Apoptosis: controlled demolition at the cellular level
R.C. Taylor, S.P. Cullen and S.J. Martin (2008)
Nature Reviews. Mol Cell Biol 9, 231-241
2. Mitochondria and cell death: outer membrane permeabilization
and beyond
S.W.G. Tait and D.R. Green (2010)
Nature Reviews. Mol Cell Biol 11, 621-632
3. Programmed cell death in animal development and disease
Y. Fuchs and H. Steller (2011)
Cell, 147, 742-758
4. Expanding roles of programmed cell death in mammalian
neurodevelopment
D. De Zio, L. Giunta, M. Corvaro, E. Ferraro and F. Cecconi (2005)
Semin Cell Dev Biol 16, 281-294

![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)


![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)