Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 – The McGraw-Hill Companies srl
CAPITOLO 3
PER IL RIPASSO
1.
2.
3.
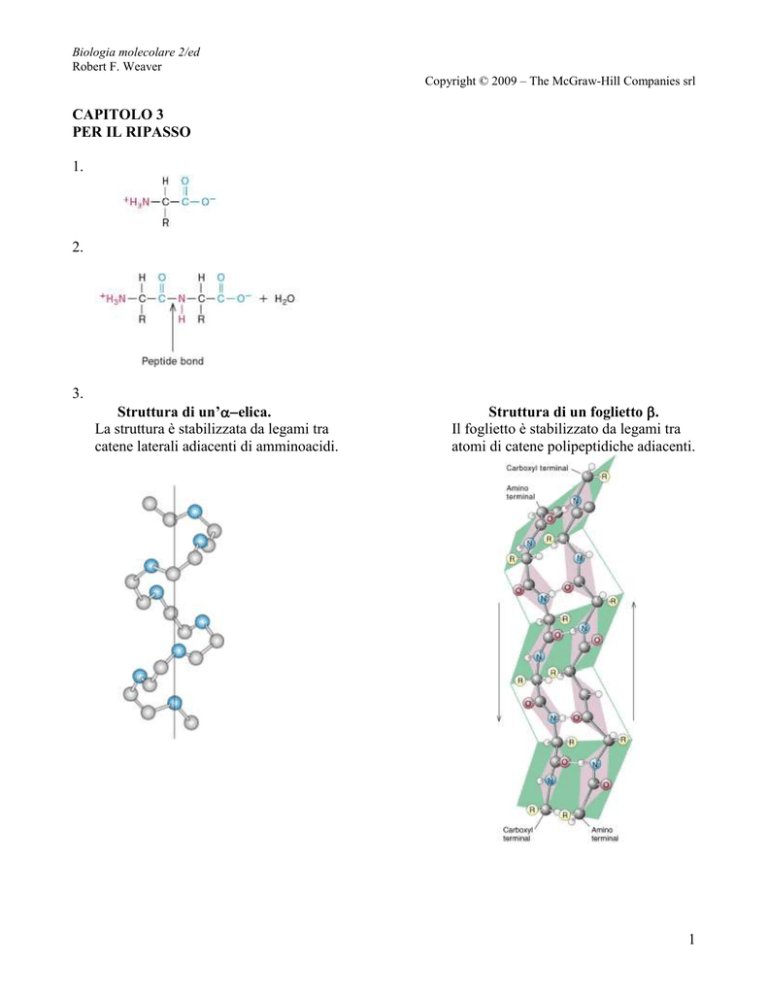
Struttura di un’elica.
La struttura è stabilizzata da legami tra
catene laterali adiacenti di amminoacidi.
Struttura di un foglietto .
Il foglietto è stabilizzato da legami tra
atomi di catene polipeptidiche adiacenti.
1
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 – The McGraw-Hill Companies srl
4. La struttura primaria di una proteina è la successione lineare di amminoacidi nella catena
polipeptidica, uniti da legami peptidici covalenti. La struttura secondaria si riferisce alla
formazione di -eliche o foglietti da parte della catena peptidica. Le -eliche sono stabilizzate da
legami a idrogeno tra le catene laterali degli amminoacidi, mentre i foglietti derivano
dall’impacchettamento affiancato delle catene polipeptidiche, unite da legami a idrogeno tra le
ossature parallele della catena. I due tipi di struttura secondaria possono coesistere, e risultare uniti
da tratti curvilinei, in una catena polipeptidica. La struttura terziaria descrive la forma
tridimensionale di una proteina che si ripiega nella sua conformazione funzionale. Questa
conformazione è il risultato di una serie di diverse interazioni tra gruppi funzionali degli
amminoacidi, che comprendono legami ionici, legami a idrogeno ed interazioni idrofobiche.
Sebbene le interazioni non covalenti giochino il ruolo più significativo nella stabilizzazione della
struttura terziaria di una proteina, i legami covalenti, ed in particolar modo i ponti disolfuro tra
cisteine, possono anche essere presenti.
La struttra quaternaria di una proteina descrive l’assemblaggio di due o più catene polipeptidiche, a
formare la proteina funzionale. La struttura quaternaria di una proteina è stabilizzata in maggior
misura da interazioni idrofobiche.
5. Archibald Garrod è il fisico accreditato per aver notato la natura ereditaria della malattia
Alcaptonuria. Essendo a conoscenza del lavoro di Mendel, egli propose che un singolo gene
recessivo fosse responsabile della malattia. Egli ipotizzò anche che la malattia fosse causata da un
difetto in un pathway metabolico, e quindi di un enzima. Associando queste osservazioni, egli
stabilì un nesso tra la proteina enzimatica ed il gene recessivo. Ciò suggerì che i geni codificano
per proteine.
6. Beadle and Tatum usarono il sistema sperimentale di Neurospora crassa per dimostrare la
relazione tra geni e proteine. Neurospora è un eucariote aploide in grado di riprodursi sessualmente
per fusione di due nuclei aploidi, da cui, per meiosi, derivano spore aploidi. Per la sua crescita, il
fungo sfrutta una serie di pathway biochimici che sintetizzano un set completo di amminoacidi e
vitamine, in presenza di azoto inorganico e di biotina. In seguito ad irradiazione delle strutture
fungine che producevano le spore con raggi X, e grazie ad analisi genetica, essi produssero diversi
ceppi puri del fungo, ciascuno dei quali non era in grado di crescere su terreno minimo, ma che
cresceva su terreno contenente gli amminoacidi e le vitamine che potevano essere sintetizzate dai
funghi selvatici. Quindi intrapresero il paziente lavoro di classificare i diversi ceppi per difetti negli
specifici pathway biochimici, stabilendo quale ceppo era capace di crescere con l’aggiunta di una
particolare proteina, ad esempio il pantotenato. Un ceppo che cresceva solo in presenza della
particolare vitamina aveva evidentemente mutazioni in quel pathway. Quindi verificarono la
capacità di vari intermedi che permettevano la crescita del fungo (per complementazione della
mutazione), il che consentì loro di determinare con maggiore accuratezza il passaggio alterato del
pathway. Se un ceppo cresceva con un dato intermedio, essi concludevano che la mutazione agisse
prima (a monte) della produzione di quell’intermedio. Così, essi costruirono una serie di mutanti,
ciascuno con uno specifico difetto enzimatico ereditato come singolo gene. Ciò dimostrò il nesso
tra geni singoli e i singoli polipeptidi corrispondenti a ciascun enzima.
2
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 – The McGraw-Hill Companies srl
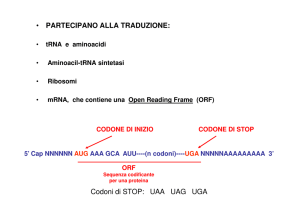
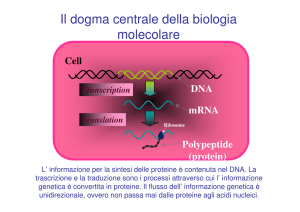
7. I due principali passaggi nell’espressione di un gene sono la trascrizione, che consiste nella
generazione di una molecola di mRNA da uno stampo di DNA, e la traduzione, ovvero la
generazione di una catena polipeptidica secondo l’informazione contenuta nella sequenza lineare di
codoni nell’RNA messaggero.
8. Osserva la Figura 3.12. Il lavoro di Jacob e colleghi dimostrò l’esistenza di una molecola di RNA,
l’RNA messaggero, che si associa in maniera transiente con il ribosoma e dirige la sintesi proteica.
Essi fornirono evidenze in contrasto con l’ipotesi di Crick, secondo cui l’informazione genetica era
portata dall’RNA ribosomale, e che quindi sarebbe stata parte integrante del ribosoma. Essi
usarono cellule di E. coli infettate con il batteriofago T2 come sistema sperimentale. In seguito
all’infezione di E. coli con il fago T2, c’era un’alterazione della sintesi proteica, perché i batteri
cominciavano a sintetizzare le proteine fagiche. Jacob e collaboratori ipotizzarono che, se la sintesi
era diretta da una molecola di RNA messaggero non associata permanentemente con i ribosomi,
un’alterazione della sintesi proteica non avrebbe dovuto richiedere la sostituzione di ribosomi con
quelli che trasportavano l’informazione per la sintesi delle proteine fagiche. Per marcare e seguire i
ribosomi prima e dopo l’infezione fagica, essi fecero crescere E. coli in presenza di isotopi pesanti
dell’azoto e del carbonio, prima dell’infezione. Queste colture di E. coli venivano quindi trasferite
in mezzi di coltura con i normali isotopi leggeri di azoto e carbonio e, allo stesso tempo venivano
infettate con il batteriofago T2, con l’aggiunta di 32P per marcare gli mRNA neosintetizzati.
Sfruttando la centrifugazione su gradiente di densità fu possibile separare i ribosomi vecchi,
pesanti, dai ribosomi leggeri, neosintetizzati. Quindi stabilirono se l’RNA associato a ciascun tipo
di ribosomi fosse marcato con 32P, e quindi appena sintetizzato. Essi dimostrarono che l’RNA
marcato era associato con i ribosomi prodotti prima dell’infezione con il fago. Ciò dimostrò
l’esistenza di una molecola messaggera di mRNA che si associa transientemente con i ribosomi per
dirigere la sintesi di una specifica proteina. Quindi, la proteina sintetizzata da un ribosoma poteva
cambiare, a seconda di quale mRNA fosse associato con il ribosoma.
9. I tre passaggi della trascrizione sono l’inizio, l’allungamento e la terminazione.
L’inizio consiste nel legame dell’RNA polimerasi al promotore e nella denaturazione
localizzata di una regione di circa 12 coppie di basi. La polimerasi quindi inizia la sintesi di una
catena di RNA complementare al filamento stampo.
3
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 – The McGraw-Hill Companies srl
La fase di allungamento inizia dopo che sono stati assemblati i primi pochi nucleotidi, e la
polimerasi abbandona il promotore, spostandosi in avanti, e catalizza l’allungamento della
molecola di RNA.
Specifiche sequenze all’estremità 3’ dei geni sono responsabili della terminazione, il passaggio
finale nella trascrizione genica. L’RNA polimerasi interagisce con queste sequenze, e il
risultato è l’allentamento dell’associazione tra RNA polimerasi, RNA e lo stampo di DNA.
10. Una molecola di RNA ribosomale 5S, una di 16S e una di 23S insieme costituiscono un ribosoma
di E. coli. Gli RNA 5S e 23S si trovano nella subunità maggiore 50S del ribosoma 70S. L’RNA
ribosomale 16S è presente nella subunità minore 30S del ribosoma di E. coli.
11. La struttura a trifoglio del tRNA.
L’amminoacido
si lega qui
4
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 – The McGraw-Hill Companies srl
12. Una molecola di tRNA possiede due estremità funzionali, il sito di legame dell’amminoacido, e
l’anticodone. La chiave per la sua funzione di adattatore tra i codoni dell’mRNA e gli amminoacidi
nella proteina è l’abbinamento specifico di amminoacidi ai tRNA. Il codone per l’amminoacido
legato ad un tRNA è complementare all’anticodone su quella specifica molecola di tRNA. La
specificità dell’abbinamento è il risultato dell’azione di specifiche amminoacil tRNA sintetasi che
catalizzano la generazione di molecole di tRNA cariche. Ad esempio, l’enzima fenilalanina-tRNA
sintetasi lega l’amminoacido fenilalanina a molecole di tRNA con il codone GAA, che si appaierà
ai codoni della fenilalanina UUC o UUU, assicurando la corretta aggiunta di questo amminoacido
alla catena peptidica in crescita. Quindi, le molecole di tRNA consentono la generazione di una
catena polipeptidica, in cui la sequenza lineare di amminoacidi è determinata dalla sequenza dei
codoni nell’mRNA.
13. Una singola sostituzione nucleotidica in un gene può risultare nella terminazione prematura della
traduzione dell’mRNA corrispondente a quel gene, se la sostituzione genera un codone di stop al
posto di un codone per un amminoacido. Ad esempio, il codone UUG specifica l’amminoacido
leucina; una singola sostituzione nucleotidica che risulti nell’alterazione di quel codone al codone
di stop UAG porterà alla terminazione prematura della catena polipeptidica sulla posizione della
leucina.
14. Il codice genetico si presenta sotto forma di codoni tri-nucleotidici, il cui ordine sequenziale dirige
la sequenza lineare di amminoacidi in una catena polipeptidica. Se una singola coppia di basi
risulta deleta nel DNA, la mutazione influenzerà non solo quel particolare codone, ma anche tutti i
successivi codoni, poiché questi avranno perso la successione originale delle triplette. Questa
mutazione è definita “di scivolamento “ (frameshift), e dopo l’alterazione saranno specificati
amminoacidi diversi rispetto a quelli identificati dalla successione originale delle triplette. Tutto
ciò comporterà, con elevata probabilità, l’introduzione di un codone di stop nella sequenza
nucleotidica, facendo terminare prematuramente la traduzione del corrispondente mRNA.
Considera questo esempio:
La sequenza ACU AUA GAA GGU dirige la sintesi della sequenza peptidica treoninaIsoleucina-glutammato-glicina. La delezione della “A” in posizione 4 risulterà nella sequenza
ACU UAG AAG GU. Il secondo codone è ora un codone di stop, UAG, e la traduzione si
arresterà in quel punto.
15. La sostituzione di un nucleotide per un altro nella regione codificante di un gene cambierà la
sequenza di un codone. A causa della degenerazione del codice genetico, questo potrebbe lasciare
inalterato l’amminoacido specificato da quel codone, ma spesso lo altererà. Ad esempio, il codone
GGC specifica per l’amminoacido glicina. La sostituzione della seconda G con una C genererà
GCC, che specifica l’alanina.
PER L’APPROFONDIMENTO
1. Data la porzione di un gene batterico con il filamento stampo in basso:
5’ GTATCGTATGCATGCATCGTGAC 3’
5
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 – The McGraw-Hill Companies srl
3’ CATAGCATACGTACGTAGCACTG 5’
a) L’mRNA trascritto da questo gene a cominciare dalla prima T sarà:
5’AUCGUAUGCAUGCAUCGUGAC 3’
b) Il codone di inizio (AUG) è sottolineato.
c) Il cambio della prima G nel filamento stampo ad una C risulterebbe in un codone di
inizio AUG tre codoni a monte rispetto al caso b), generando una cornice di lettura più
estesa.
dsDNA
5’ GTATCGTATGCATGCATCGTGAC 3’
3’ CATACCATACGTACGTAGCACTG 5’
mRNA
5’ AUGGUAUGCAUGCAUCGUGAC 3’
d) Il cambio della seconda T nel filamento stampo a G risulterebbe in una cornice di
lettura aperta più corta, in quanto il codone di inizio AUG si troverebbe più a valle.
dsDNA
5’ GTATCGTATGCATGCATCGTGAC 3’
3’ CATAGCAGACGTACGTAGCACTG 5’
mRNA
5’ AUCGUCUGCAUGCAUCGUGAC 3’
e) Il cambio dell’ultima T nel filamento stampo ad una C cambierebbe il codone di stop
UGA con il codone UGG, per il triptofano. Ciò risulterebbe in una proteina più lunga,
in quanto la traduzione procederebbe oltre questo codone, fino al successivo codone di
stop.
dsDNA
5’ GTATCGTATGCATGCATCGTGAC 3’
3’ CATAGCATACGTACGTAGCACCG 5’
2. Un mutante per la biosintesi del pantotenato, in grado di crescere su pantoato deve avere la
capacità di completare il terzo passaggio, quello finale, nel pathway biosintetico del pantotenato.
Possiamo concludere che la mutazione coinvolga il passaggio 1 o il passaggio 2 del pathway.
6
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 – The McGraw-Hill Companies srl
7