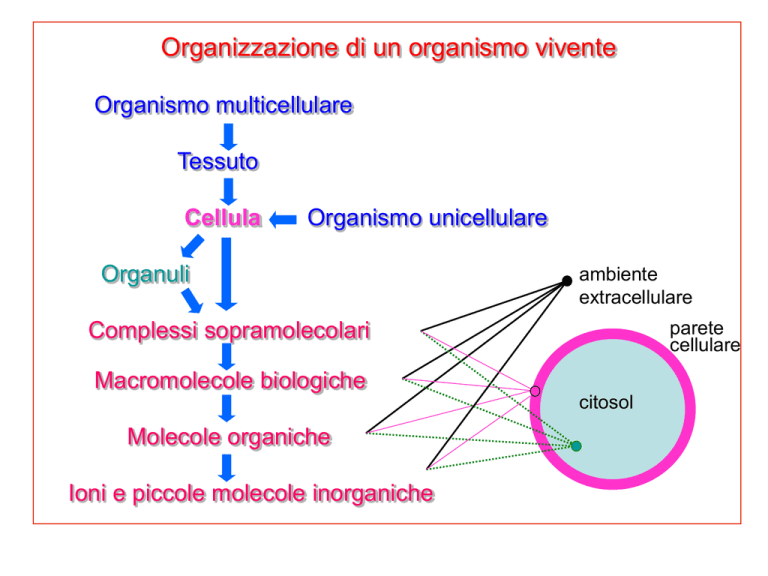
Organizzazione di un organismo vivente
Organismo multicellulare
Tessuto
Cellula
Organismo unicellulare
Organuli
ambiente
extracellulare
parete
cellulare
Complessi sopramolecolari
Macromolecole biologiche
citosol
Molecole organiche
Ioni e piccole molecole inorganiche
Organizzazione di una cellula
procariotica
1 mm (10-6m)
Organizzazione di una cellula
eucariotico
La cellula procariotica
La cellula eucariotica
membrane
ribosome
Sistemi biologici - dimensioni
complessi
macromlecole sopramolecolari
cellule
tessuti ed organismi
Microscopia ottica
Microscopia elettronica
Cristallografia raggi X / Risonanza magnetica
SAR (Structure Activity Relationship)
Forma nativa ben definita
Complementarietà di forma
Interazione fra biomolecole
Variazione nella struttura molecolare
Variazione dell’attività molecolare
Effetto cellulare
Effetto tessutale
interazioni deboli
intramolecolari
Impalcatura
covalente
ripiegamento
interazione
forma amorfa
(disordinata)
forma ‘nativa’
(strutturata)
interazioni deboli
intermolecolari
Legami ed interazioni chimiche nelle biomolecole
Legami deboli
Legami forti
-
-
interazioni normalmente irreversibili
impalcature molecolari
Legami covalenti
interazioni reversibili
interazioni fra molecole
Legami non-covalenti
Legami forti: legami covalenti
(nelle molecole organiche)
singolo
doppio
triplo
principali tipi di legame covalente nelle biomolecole
C
300-400 kJ/mole
H
C
C
C
C
O
O
H
C
N
N
H
S
S
H
S
S
S
M
O
N
O
P
O
M
N
M
C = Carbonio
H = Idrogeno
O = Ossigeno
N = Azoto
S = Zolfo
P = Fosforo
M = ione metallico
(Fe, Zn, Cu, Ni, Ca ecc.)
Principali gruppi funzionali chimici nelle biomolecole
Amminoacidi
Nucleotide
gruppo
sulfidrilico
gruppo carbonilico
gruppi amminici
(1°, 2°)
(basici)
gruppo
fosfato
anello aromatico
(eteroaromatico)
gruppo
carbossilico
(acido)
gruppo ammidico
gruppi
idrossilici
Eme
centro di
coordinazione
metallico
–COOH
–NH2
–COO –
–NH3 +
Struttura delle biomolecole - conformazione e configurazione
due conformazioni
(facilmente intercambiabili)
due configurazioni
(difficilmente intercambiabili)
trans
cis
2) Legami deboli: legami elettrostatici (legami ionici / ponti salini)
- attrazioni fra gruppi con cariche opposte
-
::::::
+
- legami relativamente deboli e reversibili
- molto dipendenti dall’ambiente (più deboli in acqua che in un ambiente organico)
in acqua D = 80 F 4-7 kJ/mole
in cicloesano D = 4 F > 200 kJ/mole
3) Legami deboli: legami idrogeno (legami-H)
- condivisione di un protone fra due gruppi chimici
(proton donatore e proton accettore)
R-OH
O=R
-CH3
- legami deboli e facilmente reversibili
4-30 kJ/mole
- altamente dipendenti dalla distanza e direzione
-O-H --- O=R-
-O-H
O=R-
Proton
donatore
legami-H fra le basi del DNA
Proton
accettore
legami-H fra gruppi amminici/di e carbonili
negli amminoacidi
Regola: le macromolecole biologiche tendono a formare il massimo numero di
legami-H possibile (fra loro gruppi proton-accettori/-donatori o fra
questi e molecole d’acqua)
4) Legami deboli: Forze attrattive di van der Waals (VDH)
- interazioni non-specifiche fra dipoli transienti
negli atomi di carbonio
-
- +
+
C
C
- legami molto deboli e facilmente reversibili
- diventano importanti solo se coinvolgono moltissimi atomi e interazioni
(macromolecole biologiche)
superficie molecolare
- richiedono complementarità di forma
fra estese superfici molecolari
superficie molecolare
5) Legami deboli: interazioni idrofobiche
-
dovute alla polarità e alla coesione dell'acqua
-
molecole apolari (incapaci di formare legami-H) si aggregano per:
• minimizzare la superficie esposta all’acqua
• permettere la formazione del massimo numero di legami-H nell’ambiente
acquoso
• aumentare l’entropia del solvente (H20)
entropia
aumentata
entropia elevata
entropia ridotta
Alcune regole per la strutturazione delle macromolecole
1) Le macromolecole biologiche sfruttano la libertà conformazionale per accedere
alla struttura più stabile
2) Interazioni idrofobiche lungo la catena di residui che le compongono
favoriscono la formazione di strutturi compatti poiché questo riduce l’area di
superficie molecolare apolare esposta al solvente acquoso
3) Nella struttura finale si forma il massimo numero possibile di legami-H
4) Legami elettrostatici e di VDW contribuiscono a rendere più stabile la struttura
5) La struttura finale è quella termodinamicamente più stabile (con minore energia)
• Per un processo od una reazione chimica;
• G negativa – reazione spontanea
G = H - TS
• G positiva – reazione sfavorita
• G = 0
– reazione ad equilibrio
• ATP (adenosina trifosfato): elevato potenziale di trasferimento per gruppi
fosfato
ATP + H20
ATP + H20
E1
E2
ADP + P + H+
G° ~ -30 kJ/mole
AMP + P P + H+
G° ~ -32 kJ/mole
E3
P P + H20
2 P
+ H+
G° ~ -33 kJ/mole
• Una reazione sfavorevole può essere spinta dall’accoppiamento con un’altra
reazione con G negativo
A
ATP + H20
A + ATP + H20
B
ADP + P + H+
B + ADP + P + H+
G +16 ki/mole
G
G
-30 kJ/mole
-14 kJ/mole
Ioni e molecole di rilevanza biologica
IONI
• elettroni e protoni: e- / H+
• SALI:
Na+, K+, Ca++, Mg++
(Cl-)
• IONI MINERALI: Mn++, Fe++ e Fe+++, Co+,++,+++, Zn++, Cr++,+++, Cu+, Mo, Se(presenti come tracce in enzimi, proteine di trasporto, vitamine etc.)
MOLECOLE INORGANICHE
H20 (>70% del peso della maggior parte degli organismi
solvente che modula la struttura/interazioni/reazioni delle biomolecole)
• O2
(fotosintesi, processi metabolici)
• CO2, NH3, NO3- ,NO, N2 (processi metabolici)
Metaboliti, signalling
• HPO42- H2PO4- H3PO4 ( Pi = ortofosfato = fosfato inorganico)
• HP2O73-
( PPi = pirofosfato)
P
P P
Fabbisogno (RDA): ~ Na 2.4g, K 3.5g, Ca 1g, Mg 0.4g, P 1g, Fe 18mg, Zn 15mg, Cu 2 mg, Se/Mo 75 mg
Fonti principali:
Na - sale; K – Verdure, frutta, noci; Ca – latticini, legumi, verdure; Mg – verdure;
Fe, Co, Cr, Se – carne e verdure
BIOMOLECOLE ORGANICHE
le principali classi di molecole organiche di rilevanza biologica sono:
• AMMINOACIDI
• CARBOIDRATI (Glucidi; zuccheri ed altri metaboliti)
• NUCLEOTIDI
• MOLECOLE LIPIDICHE (acidi grassi e molecole derivate, steroidi)
• COENZIMI (derivati da vitamine) e COFATTORI (ioni metallici)
Molecole delle prime quattro classi sono anche unità costitutive di
macromolecole biologiche (proteine, acidi nucleici, fosfolipidi...)
Precursori
inorganici
18-64 d
CO2, H2O
N2, O2, NH3
Metaboliti
organici
50-150 d
piruvato,
citrato ecc.
Unità
costitutive
100- 350 d
amminoacidi
nucleotidi, glucidi
acidi grassi
glicerolo
Macromolecole
biologiche
103-109 d
proteine,
acidi nucleici,
polisaccaridi,
lipidi
Complessi
supra_
molecolari
106-109 d
ribosomi,
citoscheletro,
virus,
compl.multienzimatici
interazioni deboli
intramolecolari
Impalcatura
covalente
ripiegamento
interazione
forma amorfa
(disordinata)
forma ‘nativa’
(strutturata)
interazioni deboli
intermolecolari
Alcune regole per la strutturazione delle macromolecole
1) Le macromolecole biologiche sfruttano la libertà conformazionale per accedere
alla struttura più stabile
2) Interazioni idrofobiche lungo la catena di residui che le compongono
favoriscono la formazione di strutturi compatti poiché questo riduce l’area di
superficie molecolare apolare esposta al solvente acquoso
3) Nella struttura finale si forma il massimo numero possibile di legami-H
4) Legami elettrostatici e di VDW contribuiscono a rendere più stabile la struttura
5) La struttura finale è quella termodinamicamente più stabile (con minore energia)
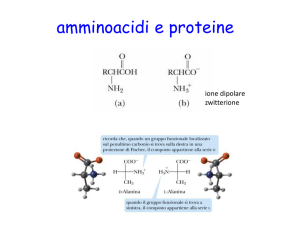
AMMINOACIDI
catena laterale
struttura
carbonio
gruppo -amminico
R
H2N C COOH
idrogeno
gruppo
-carbossilico
H
stereochimica
Configurazione D
Configurazione L
• Gli AmminoAcidi (AA) sono unità costitutive delle proteine
• Ci sono 20 diversi amminoacidi proteinogenici (tutti con configurazione L)
• Possiamo suddividerli in diversi gruppi a secondo delle caratteristiche chimico-fisiche
delle loro catene laterali
AMMINOACIDI
catena laterale
struttura
carbonio
gruppo -amminico
H
H
R
H
C
L
R
C
COOH
H2N
gruppo
-carbossilico
H2N C COOH
idrogeno
stereochimica
R
configurazioni
H2N
COOH
D
• Gli AmminoAcidi (AA) sono unità costitutive delle proteine
• Ci sono 20 diversi amminoacidi proteinogenici (configurazione L)
• Possiamo suddividerli in 7 gruppi a secondo delle caratteristiche chimicofisiche delle catene laterali
1) AMMINOACIDI CON CATENE LATERALI ALIFATICHE
HN
CH3
H C CH3
O C
C CH
CH3
alanina
Ala (A)
valina
Val (V)
Idrofobico
nm
idrofobico
nm
CH3
C CH2 CH
CH3
CH2 CH3
C CH
CH3
leucina
Leu (L)
isoleucina
molto idrofobico
nm
molto idrofobico
nm
Ile (I)
catene laterali idrofobiche
catene laterali chimicamente inerti e quindi non modificabili (nm)
partecipano solo ad interazioni idrofobiche e di VdW.
2) AMMINOACIDI CON CON CATENE LATERALI AROMATICHE
HN
H C CH 2
C CH 2
OH
C CH2
NH
~C=O
fenilalanina
Phe (F)
molto Idrofobico
nm
debole assorbimento della
luce a ~ 250nm
tirosina
Tyr (Y)
moderatamente idrofobico
m
triptofano
Trp (W)
molto idrofobico
m (ox, Br)
P
280 nm
assorbe luce ~ 280 nm
ed è fluorescente
Partecipano ad interazioni idrofobiche e di VdW.
La catena laterale di Phe è idrofobica ed inerte (nm).
Tyr e Trp partecipano a legami-H e sono suscettibili a modificazioni chimiche (m)
(Tyr per fosforilazione - importante, Trp per ossidazione, brominazione)
3) GLICINA e PROLINA
ridotta libertà
di rotazione
~HN
poco ingombro
sterico elevata libertà
di rotazione
H
C
H
~C=O
glicina
Gly (G)
nm
residuo flessibile
N C H
C O
prolina
Pro (P)
~m
residuo rigido
importanti caratteristiche strutturali (flessibilità/rigidità)
lievemente idrofilici (polari)
la catena laterale di Pro è inerte ma può essere idrossilata da
specifici enzimi (~m).
4) AMMINOACIDI CON CATENE LATERALI POLARI
~HN
H
O
O
C CH 2 OH
~C=O
serina
C CH CH 3
OH
C CH 2 C
NH 2
C CH 2 CH 2 C
treonina
asparagina
glutammina
Ser (S)
Thr (T)
Asn (N)
Gln (Q)
polare
m
polare
m
polare
m
polare
P
P
(O -glicosilazione )
(O -glicosilazione )
NH 2
( N-glicosilazione)
le catene laterali sono polari ed idrofiliche
partecipano alla formazione di legami -H
Ser, Thr ed Asn possono essere modificati enzimaticamente. La loro
fosforilazione e/o glicosilazione hanno importanti conseguenze
strutturali nelle proteine
chinasi
ADP
ATP
R-OPO32-
R-OH
HOPO32fosfatasi
glucotrasferasi
R-OH
R-O
glicosidasi
5) AMMINOACIDI CON CATENE LATERALI ACIDICHE (ANIONICHE)
~HN
O
C CH 2 C
H
O~C=O
acido aspartico
Asp (D)
O
C CH2 CH2 C
O-
acido glutammico
Glu (E)
anionico (-)
anionico (-)
pKa = 3.9
m
pKa = 4.3
m
catene laterali polari cariche negativamente (anioniche)
partecipano ad interazioni elettrostatiche ed alla formazione di legami-H
possono essere modificate enzimaticamente (m) (amidazione, fosforilazione)
e possono chelare con alcuni metalli (es. calcio, zinco, rame)
6) AMMINOACIDI CON CATENE LATERALI BASICHE (CATIONICHE)
+
~HN
C (CH2 )4 NH3
e
~C=O
+
H
lisina
Lys (K)
NH
NH
C (CH2 )3 NH C
NH2
C CH2
+
arginina
Arg (R)
cationico
cationico
pKa = 10
pKa = 12
istidina
His (H)
neutro /cationico
pKa=6.5
m
(forma basi di Schiff e
legami ammidici )
NH
m
(forma legami di
coordinazione con metalli)
catene laterali polari cariche positivamente (cationiche)
partecipano ad interazioni elettrostatiche e nella formazione di legami-H
Lys è modificabile (m) mediante reazioni enzimatiche o chimiche (base di Schiff
His può chelare molto efficientemente con metalli (Fe, Zn ecc.)
fosforilazione transiente
idrossilazione)
7) AMMINOACIDI CON ATOMI DI ZOLFO
~HN
H
C CH2 SH
C CH2 S S
CH2 C
C CH2 CH2 S
CH3
~C=O
cisteina
cistina
metionina
Cys (C)
S-S
idrofobico
Met (M)
idrofobico
polare
m
nm
la catena lateraledi Cys è polare mentre la cistina (S-S) è idrofobica
Cys partecipa alla formazione di legami covalenti reversibili (ponti disolfuro)
che ha importanti conseguenze strutturali nelle proteine.
Forma legami di chelazione con metalli
Il codone per Met coincide con il codone di Start
Leu (L)
Ile (I)
Ala (A)
Val (V)
Phe (F)
Gly(G)
Tyr (Y)
Trp (W)
Pro (P)
legami-H
legami elettrostatici
Met (M)
Ser (S)
legami-H
legami-H (Y e W)
legami elettrostatici
legami elettrostatici
interaz.VDW
interaz. di VDW
interaz.idrofobiche
interaz. idrofobiche
Thr (T)
legami elettrostatici
interaz. di VDW
interaz. idrofobiche
legami coordinati (*)
*Cys (C)
interaz. idrofobiche
Asn (N)
legami-H
Gln (Q)
interaz. di VDW
Lys (K)
Arg (R)
His (H)
legami-H
legami elettrostatici
interaz. di VDW
interaz. idrofobiche
legami coordinati
Asp (D)
Glu (E)
• Questi venti -aminoacidi sono denominati proteinogenici per distinguerli da
numerosi altri -aminoacidi non impiegati per la formazione di proteine.
• Le catene laterali sono diverse per dimensioni, carica, capacità di formare
legami idrogeno e reattività chimica.
• La diversità e la versatilità di queste 20 unità attribuisce alle proteine una grande
varietà di strutture e funzioni.
• Nell’uomo esistono vie metaboliche solo per la sintesi di una decina di questi
aminoacidi; gli altri debbono provenire dalla dieta e sono considerati
“essenziali”: (Ile, Leu, Lys, Met, Phe, Thr, Trp, Val e nei bambini anche His e
Arg).
• Non abbiamo la capacità di immagazzinare gli amminoacidi (eccetto che nelle
proteine muscolari o del siero, utilizzabili in caso di digiuno prolungato)
CARBOIDRATI
es. gli zuccheri (saccaridi, glucidi): Aldeidi o chetoni con gruppi idrossilici multipli
H
aldosi
D-gliceraldeide
chetosi
O
H
C
C
OH
O
CH2 OH
CH2 OH
zuccheri:
CH2 OH
diidrossiacetone
C
zuccheri
…osio
…ulosio (4,5 C)
e i loro derivati:
alcolici
CH 2 O H
CH
O H
CH 2 O H
D-glicerolo
amminici
desossi
HO CH 2
H
O
H
H
H
OH
OH
H
D- desossiribosio
OH
CH 2 O H
O OH
OH H
H
H
H
NH 2
b- D -galattosammina
fosfoesterici
CH
O
P
O
Base
CH OH
C H2 OPO32
gliceraldeide
-3-fosfato
P
acidi nucleici
Le fonti principali di carboidrati nella dieta sono l’amido (origine vegetale, composto da amilosio e
amilopectina, abbondante in legumi, tuberi e cereali) e glicogeno (cellule animali). Altra fonte di glucidi è
la cellulosa (ma non per l’uomo). Gran parte degli organismi utilizzano il glucosio come fonte principale di
energia. Altri zuccheri come il fruttosio e galattosio, per poter essere utilizzati, devono essere trasformati.
SACCARIDI (zuccheri, glucidi): Aldeidi o chetoni con gruppi idrossilici multipli
Formula generale: (CH2O)n
H
O
C
HO
C* H
CH2 OH
L-gliceraldeide
•
H
(CH2O)n
O
1
C
2
C* OH
3
H
CH2 OH
D-gliceraldeide
idrato del carbonio (carboidrato)
CH2 OH
1
C
2
O
CH2 OH
3
diidrossiacetone
La configurazione è determinata dalla proiezione del carbonio chirale
più distante dal gruppo ossidrile
• La configurazione D domina in natura ma non in maniera assoluta
MONOSACCARIDI: triosi, tetrosi, pentosi, esosi, eptosi
(soln. acquosa)
Forma lineare
H
Forma ciclica
6 CH 2OH
O
1
C
O
H C OH
HO C H
H C OH
H C * OH
6
CH 2OH
C OH
OH
H
C
OH
C
C C 1 O
HO
OH
anomeri
OH
6 CH OH
2
CHOH
O
carbonio
anomerico
HO
5
O
C
5
CH 2--OH
H C OH
H C OH
H C * OH
5
CH2OH
D-ribosio
PIRANOSIO
6 atomi
(5C + O)
OH
FURANOSIO
5 atomi
(4C + O)
1
D-glucosio
1
1
HO
CH2OH
6
H
OH
OH
C
HO
OH
H
OH
C
C C 1 O
OH
anomeri
HO
HO
OH
O
OH
PIRANOSIO
OH
5 CH 2OH
O
OH
FURANOSIO
HO
OH
carbonio
anomerico (riducente)
5’
Nucleotidi e Nucleosidi
P
CH 2
Base
O
4’ H
OH
Citosina (C)
Pirimidine
H
3
N
5
2
O
N
Z
6
1
N
R’
5
2
O
N
Z
Guanina (G)
NH2
4
3
H, OH
Adenina (A)
O
4
R
Purine
Timina (T) R’= C H 3
Uracile (U) R’= H
NH2
D-ribosio
2’
3’
BASI AZOTATE:
1’
H
6
1
6
1
N
5
O
H
N7
6
1
8
2
3
N
4
N
Z
N
5
8
2
9
N7
H2N
3
N
4
N
Z
9
NUCLEOSIDI:
BASE + RIBOSIO
ADENOSINA
CITIDINA
GAUNOSINA
TIMIDINA
URIDINA
NH 2
SYN
N
N
5
HO
CH2
N
O
4
N
1
2
3
OH
NUCLEOTIDI:
OH
BASE + RIBOSIO FOSFATO
P
O
OO
P
O
O
O-
POLIMERO
(DNA/RNA)
P
BASE
P P P
O
OH
B
B
ENERGIA
ATP,CTP,GTP,TTP,UTP
ENERGIA COENZIMI
OH
H/ O H
P
O
B
MESSAGGERO
INTRACELLULARE
I nucleotidi sono sintetizzati de novo a partire dal ribosio (zucchero) e dalle basi azotate, che derivano da tre
amminoacidi (Gly, Gln e Asp).
COENZIMI
Coenzima
Coenzima
stechiometrico catalitico
-OH
+
ATP
CE1
-O P
+ ADP
E1
substrato
co-substrato
enzima con
coenzima
prodotto
co-prodotto
E2
ADP + Pi
ATP
• Un catalizzatore partecipa nella reazione ma non ne viene modificato
• È presente a concentrazioni anche molto inferiori a quelle del substrato
• Il cosubstrato invece deve avere una concentrazione almeno uguale a quella del substrato
• Viene ricostituito ad opera di un altro enzima
ATP
(Adenosina trifosfato)
Legame fosfoanidrico
• Il ciclo ATP-ADP-AMP è il
modo fondamentale per lo
scambio di energia nella cellula
Mg++
• ATP è un donatore immediato di energia
con un elevato potenziale di trasferimento
di Pi o PPi (separazione e delocalizzazione
delle cariche)
• Il trasferimento avviene solo in reazioni
catalizzate da enzimi (En = chinasi)
• Pi è intercambiabile con altri nucleotidi
(CTP, GTP, UTP)
• Molecole come la fosfocreatina
nelle cellule del muscolo, hanno
un potenziale di trasferimento
di Pi superiore a quello
di ATP e possono ricostituirla
ATP + H20
ATP + H20
PPi + H20
E1
E2
E3
ADP
fosfocreatina
ADP + Pi + H+
AMP + PPi + H+
2Pi
ATP
+ H+
nicotinammide (derivato dall’acido nicotinico / niacina)
NAD+
adenosina difosfato
(ADP)
E2
+
E1 H + 2e
OR
..
ribosio
R = H NAD+
• NAD+ partecipa in reazioni del tipo
R = PO42- NADP+
+ NAD+
NADH
trasferimento e- catalizzato da deidrogenasi
Nicotinammide adenin dinucleotide (NAD+) e dinucelotide fosfato (NADP)
+ NADH + H+
• NADH è un trasportatore di elettroni in reazioni di ossido-riduzione utilizzate
principalmente per scopi bioenergetici. (H+ + 2e- H- ione idruro)
• NADPH è un trasportatore di elettroni in reazioni di ossido-riduzione utilizzate
principalmente in biosintesi riduttive. es.. R2C=O + 2NADPH R2CH2
FAD
(flavin adenin dinucleotide) e FMN (flavin mononucleotide)
FAD
forma ossidata
D-ribitolo
riboflavina
(vit. B2)
flavin mononucleotide
(FMN)
H+ + e-
FADH
AMP
•
flavina
• FAD è un trasportatore di elettroni in reazioni di
ossido-riduzione
+
-
semichinone
(2H + 2e )
R–CH2 – CH2 –R’ + FAD
R–CH = CH –R’ + FADH
E
• L’anello isoalloossazinico può accettare 1 o 2 elettroni
H+ + e-
FADH2
• FAD si lega non-covalentemente ma saldamente a
specifici enzimi. FMN si lega covalentemente a
residui negli enzimi del quale è cofattore
forma ridotta
anello
isoalloossazinico
Coenzima A (CoA)
4-fosfopanteina
trasportatore attivato di unità bicarboniose
acido pantotenico
b-mercaptoetilammina
acido panotenico
(vitamina B5)
legame tioestere
gruppo
acetile
gruppo acetile
(o acile)
E
3’,5’-ADP
CH3CO-
G - 30 kJ/mol
Acetil-CoA
O
RCH2 – C –S –
-idrogeno
O
Colina
acetilcolina
1) acil-CoA ha un elevato potenziale per il
trasferimento di gruppi acile
(2)
RCH – C –S –
-
2) attiva C per la rimozione
dell’-idrogeno
MOLECOLE LIPIDICHE
Acidi grassi – costruiti da unità di acetato (CH3CO- da AcCoA) idrocarburi
2
molecola anfipatica
cis
trans
molecole derivate da isoprene
isoprene
squalene
(triterpene / poliisoprene)
colesterolo
Alcuni acidi grassi non sono sintetizzati dai mammiferi ma sono necessari per la vita (acidi grassi
essenziali) e devono essere assunti con la dieta, principalmente da fonti vegetali. Appartengono a due
classi, omega 3 (es. acido linolenico) e omega 6 (es. acido linoleico). Dall’acido linolenico, viene
sintetizzato l’acido arachidonico, precursore delle prostaglandine
acidi grassi - caratteristiche e nomenclatura
Nome comune
N° di carboni
Nome IUPAC
N° di doppi legami
posizione doppi legami
w
acidi grassi essenziali
n -tetradecanoato
n -esadecanoato
n -ottadecanoato
9-ottadecenoato
9,12-ottadecenoato
9,12,15-ottadecenoato
riferimento
al carbonile
rif. a
w
14:0
16:0
18:0
18:1 9
18:2 9,12
18:3 9,12,15
18:1 ( w -9)
18:2 ( w -6)
18:3 (w -3)