21) Il pH influenza l'attività enzimatica modificando la struttura del sito attivo con il cambiamento della
distribuzione delle cariche coinvolte nei legami tra il substrato e il sito attivo. L'intervallo di pH ottimale è
ristretto e spesso prossimo alla neutralità.
Un aumento della temperatura accelera le reazioni, ma oltre un certo valore (50°C) gli enzimi si
denaturano. Nel corpo umano la temperatura raggiunge un limite pari a quello della temperatura corporea.
Infine anche la concentrazione del substrato fa variare la velocità delle reazioni enzimatiche. Quando tutti i
siti attivi sono occupati, la velocità raggiunge il suo valore massimo.
22) La concentrazione dei substrati influenza la velocità di reazione secondo l'equazione di MichaelisMenten
. Per basse concentrazioni di substrato la velocità cresce proporzionalmente
all'aumentare di S, perchè l'enzima è in eccesso rispetto al substrato. Invece, per alte concentrazioni la
velocità tende a un massimo, perchè i siti attivi degli enzimi sono tutti occupati.
La
, costante di Michaelsen-Menten, è la concentrazione di substrato necessaria a raggiungere la metà
della velocità massima
di reazione. E' una caratteristica sperimentale specifica per ogni enzima e
indica l'affinità tra l'enzima e il substrato: più la
è alta, minore è l'affinità (serve una maggiore quantità
di substrato per raggiungere la metà della velocità massima), viceversa per una
bassa.
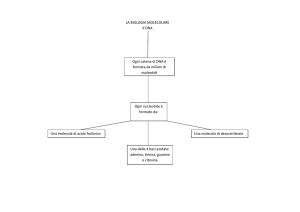
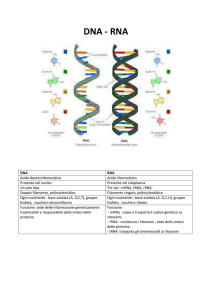
23) Le basi azotate del DNA sono 4 composti eterociclici: 2 purine (adenina A e guanina G) e 2 pirimidine
(citosina C e timina T). Nell'RNA invece della timina c'è l'uracile. I nucleosidi sono composti da uno zucchero
pentoso (ribosio per l'RNA, 2-desossiribosio per il DNA) e una base azotata legata al carbonio 1. Quando al
carbonio 5 si lega anche un gruppo fosfato, la molecola diventa un nucleotide, il monomero degli acidi
nucleici (Dna e RNA). Questi ultimi sono una lunga catena di nucleotidi uniti per mezzo di legami covalenti
tra il gruppo ossidrile del carbonio 3 di una molecola e il gruppo fosfato di un altra molecola. Pertanto ogni
nucleotide forma legami con altri 2 nucleotidi.
24) Il DNA è composto da 2 catene polinucleotidiche avvolte attorno allo stesso asse in modo da formare
una doppia elica con andamento costante e avvolgimento destrogiro. Gli zuccheri e i gruppi fosfati sono
rivolti verso l'esterno e formano lo scheletro dell'elica. Le basi azotate invece, rivolte verso il centro dell'elica,
si legano con le basi complementari dell'altra catena attraverso legami a idrogeno (adenina e timina formano
2 legami a idrogeno; citosina e guanina ne formano 3). Le 2 catene inoltre sono anti parallele perchè hanno
versi opposti: se all'estremo di una catena corrisponde il carbonio 3', esso sarà appaiato al carbonio 5'
dell'altra catena.
25) Duplicazione del DNA:
1. il complesso di duplicazione si lega al DNA in particolare tratto (ricco di AT, che formano soli 2 legami
a idrogeno), detto origine della duplicazione.
2. l'enzima elicasi rompe i legami a idrogeno tra le basi azotate e forma le bolle di duplicazione; nel
frattempo un complesso proteico impedisce che si riformi la spirale
3. la primasi crea un piccolo filamento di avvio, detto primer, complementare al DNA che permette
l'inizio della duplicazione
4. la DNApolimerasi comincia a duplicare il filamento stampo in direzione 5'-->3' : un filamento
duplicato sarà continuo e veloce, l'altro sarà frammentato e lento nel processo di duplicazione
5. la ligasi unisce i frammenti del filamento lento
6. in alcune cellule è presente l'enzima telomerasi che sintetizza un piccolo frammento di dna al posto
del primer (il primer finito il processo si stacca dal filamento lasciando una piccola sequenza di dna
disaccoppiata)
26) il DNApolimerasi duplica i 2 filamenti stampo aggiungendo un nucleotide alla volta all'estremità 3' del
filamento che sta costruendo. Questo enzima procede sempre in direzione 5' --> 3'.
Un filamento è veloce e continuo perchè è orientato nel verso con cui avanza la polimerasi.
L'altro filamento è lento e frammentario perchè è anti parallelo rispetto il filamento veloce: man mano che
l'elica viene despiralizzata l'estremità 3' del primer si allontana e si crea uno spazio vuoto. La polimerasi
quindi sintetizza piccoli frammenti alla volta, detti frammenti di Okazaki, impiegando molto più tempo.
Mentre nel filamento veloce basta un solo primer, in quello lento ne servono tanti quanti sono i frammenti.
27) Dopo che la duplicazione finisce, il primer si stacca lasciando una sequenza di dna spaiata, detta
telomero, da entrambe le estremità. Allora si attivano dei meccanismi che tagliano via questo filamento
singolo assieme a una piccola parte di filamento duplicato. Ogni processo di duplicazione il filamento di dna
si accorcia finchè diventa inutilizzabile portando la morte della cellula. In alcune cellule (midollo osseo e
gameti) esiste un enzima DNAtelomerasi che sintetizza un filamento complementare al telomero.
Questo enzima è presente nella maggior parte delle cellule tumorali, permettendo loro di continuare a
divedersi. Poichè la maggior parte delle cellule umane non ha queste capacità, i farmaci antitumorali che
attaccano questo enzima potrebbero essere una possibile cura.
28) Esistono 3 meccanismi di riparazione che aggiustano il dna per errori durante la duplicazione o per
mutazioni dovute ad agenti esterni:
1. correzione di bozze: man mano che la DNApolimerasi aggiunge nucleotidi al nuovo filamento,
controlla che quelli aggiunti prima siano corretti. Se non lo sono li elimina e riprova ad aggiungere
quello giusto
2. riparazione di anomalie di appaiamento: dopo che il dna è stato duplicato, una serie di proteine lo
controlla. Quando si accorge che è stata formata una coppia sbagliata (es. AC), sostituisce la base
sbagliata dal filamento appena formato. Queste proteine riescono a riconoscere il filamento appena
duplicato perchè ancora non è stato metilato (non sono state aggiunti gruppi metili ad alcune
adenine)
3. riparazione per escissione: la molecola di dna si può danneggiare anche in seguito alla duplicazione,
per agenti mutageni (radiazioni o sostanze chimiche). Alcuni enzimi ispezionano il dna e quando
trovano l'errore, eliminano la base danneggiata e quelle vicine. Quindi intervengono la
DNApolimerasi e la DNAligasi che sintetizzano una nuova sequenza
29) Gli acidi ribonucleici RNA sono formati da un solo filamento e nelle cellule se ne differenziano 3 per
forma e funzione.
RNA messaggero (mRNA): serve a portare fuori dal nucleo della cellula l'informazione genetica del
dna. Ne esiste uno per ogni proteina da sintetizzare.
RNA ribosomiale (rRNA): è il principale costituente dei ribosomi, che servono a sintetizzare le
proteine
RNA transfert (tRNA): è formato da pochi nucleotidi (75-90) ma è molto diversificato. Infatti esiste
un tipo di tRNA specifico per ognuno dei 20 amminoacidi che deve trasportare durante la traduzione.
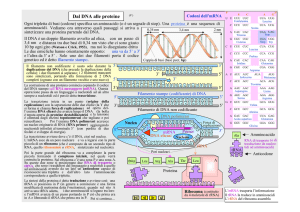
30) La trascrizione è un processo simile alla duplicazione tranne per il fatto che vengono usati ribonucleotidi
e alla fine non si avrà una nuova molecola a doppia elica, ma un solo filamento, detto mRNA. Grazie a questo
processo si può portare fuori dal nucleo l'informazione genetica, evitando che il dna subisca danneggiamenti.
Avviene in 3 fasi:
1. inizio: l'enzima RNApolimerasi si lega al promotore, una sequenza iniziale, e inizia e despiralizzare
l'elica
2. allungamento: la polimerasi legge il filamento e inizia la sintesi dell'mRNA aggiungendo nucleotidi in
direzione 5'-->3'
3. terminazione: la polimerasi raggiunge una sequenza di dna che gli dice di finire e il processo e dal
dna si stacca l'mRNA, che prima di essere usato dovrà essere ancora perfezionato.
31) Nella fase di inizio della trascrizione la RNApolimerasi si lega a un promotore, una sequenza particolare
del dna, che dice all'enzima 3 cose: dove far partire la trascrizione; quale filamento di dna trascrivere; in
quale direzione procedere.
Nei procarioti il promotore è formato solo dalla sequenza di riconoscimento e dalla TATA BOX, chiamata cosi
perchè è ricca di basi A e T.
Negli eucarioti, invece, l'RNApolimerasi non si può legare direttamente al promotore ma ha bisogno di
proteine regolatrici, detti fattori di trascrizione, che si leghino al TATA BOX e favoriscano il legame
dell'enzima.
32) Dopo che l'RNApolimerasi si è legato al promotore inizia il processo di allungamento. L'enzima comincia
a leggere il filamento stampo e senza il primer aggiunge un nucleotide alla volta in direzione 5'-->3'. A
differenza del dna, il complesso di trascrizione non controlla eventuali errori perchè spesso non
compromettono i processi successivi e perchè l'mRNA ha una vita breve. Le mutazioni del dna invece sono
molto più pericolose perchè l'errore si trasmette a tutte le cellule figlie
Quando l'RNApolimerasi incontra sul dna una particolare sequenza di basi che stabilisce la terminazione, il
filamento di rna si stacca per essere perfezionato nel processo di splicing: le sequenze non codificanti
(introni) vengono eliminate, e la parti codificanti (esoni) vengono unite.
33) Il tRNA è un filamento breve di rna, che presenta una configurazione specifica mantenuta dai legami a
idrogeno tra le basi azotate. L'estremità 3' del filamento, sito di attacco, presenta un gruppo ossidrile e 3
specifiche basi azotate che servono a legare l'amminoacido. Nel mezzo del filamento esiste una sequenza di 3
basi azotate, anticodone, che permette l'appaiamento del tRNA con l'mRNA. Questa molecola svolge 3
funzioni:
1. si lega a un preciso amminoacido
2. si associa alle molecole di mRNA
3. interagisce con i ribosomi per formare la catena polipeptidica
34) I ribosomi sono il luogo fisico del citoplasma in cui avviene la sintesi della proteine. E' costituito da 2
subunità separate quando non sono impegnate nel processo di traduzione: una maggiore formata da 3
diverse molecole di rna e 45 proteine; una minore composta da una molecola di tRNA e 33 differenti
proteine.
Sulla subunità maggiore sono presenti 3 siti di legame con il tRNA:
sito A (attacco): qui l'anticodone del tRNA cario si lega al codone dell'mRNA
sito C (condensazione): il tRNA cede il proprio amminoacido che si lega alla catena polipeptidica in
formazione
sito D (distacco): luogo dove si trova il tRNA ormai scarico, prima di staccarsi dal complesso e tornare
nel citosol per caricare un altro amminoacido
35) Il codice genetico è la chiave di interpretazione tra la sequenza di mRNA e gli amminoacidi che
compongono le proteine. Ogni sequenza di 3 basi lungo la catena dell'mRNA rappresenta un codone,
specifico per ogni amminoacido, che si lega per complementarietà con l'anticodone del tRNA. Tutte le
possibile triplette quindi sono 64= (4 basi azotate in gruppi da 3), ma gli amminoacidi in natura sono solo
20. Questo perchè il codice genetico è ridondante (un amminoacido può essere codificato da più triplette),
ma univoco (ad ogni tripletta corrisponde uno ed un solo amminoacido). Inoltre è universale, cioè è uguale
in quasi tutti gli esseri viventi della terra.
36) La traduzione si divide in 3 tappe:
1. inizio: la subunità ribosomiale minore si lega all'mRNA e per complementarietà di basi si aggiunge
anche il tRNA che trasporta la metionina. Quindi la subunità maggiore si unisce al complesso e il
tRNA con metionina scorre nel sito C, mentre nel sito A si allinea un secondo tRNA
2. allungamento: la subunità maggiore presenta l'enzima ribozima che catalizza 2 reazioni
rompe il legame tra il tRNA nel sito C e il suo amminoacido
catalizza la formazione del legame peptidico tra questo amminoacido e quello ancora legato
al tRNA nel sito A
Quindi il tRNA scarico va nel sito D e si stacca e il tRNA con la catena peptidica va nel sito C. Nel sito A
libero si attacca il tRNA successivo e il processo si ripete
3. terminazione: quando nel sito A si lega un tRNA con un codone di stop, che non codifica alcun
amminoacido, si lega al complesso un fattore di rilascio che rompe il legame tra la catena
polipeptidica e il tRNA nel sito C
37) La catena polipeptidica che si forma dai ribosomi non è ancora una proteina funzionante. La sequenza
degli amminoacidi stabilisce la configurazione tridimensionale che la catena assume e una sequenza segnale,
che fornisce alla catena ulteriori istruzioni:
la traduzione è finita e spedisce la catena in un preciso organulo (nucleo, mitocondri, plastidi)
la traduzione si interrompe e prosegue nel reticolo endoplasmatico ruvido RER. Una volta
completata la sintesi la catena raggiungerà l'apparato di Golgi
38) Durante la duplicazione del dna possono verificarsi errori che varranno trasmessi alle cellule figlie. Le
mutazioni si dividono in:
mutazioni somatiche che si verificano nelle cellule del soma (organismo), che durante la mitosi
vengono trasmesse alle cellule figlie, ma non vengono ereditate dalla prole dell'organismo. Ad
esempio una mutazione delle cellule della pelle può provocare una macchia cutanea, che però non
sarà presente nei figli
mutazioni nella linea germinale si verificano nelle cellule germinali, specializzate nella produzione
dei gameti. Quindi queste mutazioni vengono trasmesse ai figli
39) Le mutazioni puntiformi colpiscono una singola coppia di basi, quindi riguardano un solo gene. Si
dividono in:
mutazioni silenti: viene modificata l'ultima base della tripletta. Sono molto frequenti ma non fanno
danni perchè un amminoacido viene codificato da più triplette.
mutazioni di senso: il cambiamento di base nella sequenza modifica il messaggio genetico in modo
che nella proteina ci sia un amminoacido sbagliato. La proteina perde o diminuisce le sue funzionalità
come nell'anemia falciforme
mutazioni non senso: la sostituzione di base fa sì che nell'mRNA si formi un codone di stop creeando
una proteina più breve del normale e non funzionante
mutazioni per scorrimento della finestra di lettura (frame shift): una singola coppia di basi viene
aggiunta o rimossa dalla sequenza del DNA, alterando tutta la codificazione, perchè tutte le triplette
vengono sfalsate di una posizione
40) Nelle mutazioni genomiche l'organismo presenta dei cromosomi in più o in meno rispetto al normale:
euploidia aberrante: ci sono in più o mancano interi corredi cromosomici
aneuploidia: manca o c'è in più solo una parte del corredo cromosico. Questa mutazione è frequente
negli organismi diploidi: per la mancanza di un cromosoma (monosomia) come nella sindrome di
Turner nella quale manca un cromosoma X e porta alla nascita di una femmina X0; o per la presenza
di un cromosoma in più (trisomia) come la sindrome di Down (trisomia 21) o la sindrome di
Klinefelter che porta alla nascita di un maschio XXY a causa di una mancata disgiunzione.
41) Le mutazioni cromosomiche riguardano pezzi di dna:
delezione: una molecola di dna si spezza in 2 punti e le 2 porzioni esterne si ricongiungono in modo
sbagliato
duplicazione: due cromosomi omologhi si spezzano in due punti diversi e ognuno si ricongiunge con
il pezzo dell'altro. Un cromosoma avrà una parte ripetuta, l'altro avrà una parte inmeno
inversione: un segmento di dna si stacca e si riunisce nel verso opposto
traslocazione: due cromosomi diversi si spezzano e si ricongiungono con il pezzo dell'altro
cromosoma. Può provocare la sterilità perchè nella meiosi i cromosomi omologhi non riescono più ad
appaiarsi
42) Le mutazioni sono alla base dell'evoluzione degli esseri viventi: infatti forniscono la variabilità genetica su
cui agiscono la selezione naturale e gli altri agenti dell'evoluzione. Le mutazioni sono molto rare e spesso
sono puntiformi, cioè modificano un solo nucleotide. Possono nuocere all'organismo, essere neutre oppure
possono anche migliorare le capacità di sopravvivenza e di adattamento all'ambiente. Negli organismi che
possiedono migliaia di geni durante la duplicazione si può formare un altro gene identico. Una copia continua
a produrre le proteine giuste, l'altra subendo varie mutazioni, alla fine può produrre proteine migliori.







![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)